2. 大洋渔业资源可持续开发省部共建教育部重点实验室, 上海 201306;
3. 国家远洋渔业工程技术研究中心, 上海 201306;
4. 农业农村部大洋渔业开发重点实验室, 上海 201306
2. The Key Laboratory of Sustainable Exploitation of Oceanic Fisheries Resources, Shanghai Ocean University, Ministry of Education, Shanghai 201306, China;
3. National Engineering Research Center for Oceanic Fisheries, Shanghai 201306, China;
4. Key Laboratory of Oceanic Fisheries Exploration, Ministry of Agriculture and Rural Affairs, Shanghai 201306, China
水生生态系统(aquatic ecosystem)是指由水生生物与水体环境构成的统一体, 可简单分为淡水生态系统和海洋生态系统。环境变化会导致水生生态系统结构的转变, 最为直接的影响就是引起系统中生物营养级的升高或降低。如何评估水生生物在食物网中的关系及其变化是学者们研究水生生态系统的一个重要领域, 由此而产生了摄食生态学(trophic ecology), 即对群落或生态系统中生物体摄食关系的研究。摄食生态学中研究的基本问题主要分为:食物链长度与生产者生物量之间的关系; 资源的供应量与每一营养级生物相对丰度的关系; 食物链的实际长度及限制因素[1]。食物链的本质是生物的捕食—被捕食关系, 因此, 了解研究对象的摄食情况是进行摄食生态学研究的基础。
在水生生态系统中, 捕食者-被捕食者之间的相互作用关系是很难被直接观察, 因此, 科学家们最初常用视觉方法——胃含物分析来了解这一关系, 即在特定水域采样通过观察样品胃含物形态学特征确定其成分[2]。Hyslop[3]提出, 基于胃含物分析的方法是摄食生态学研究的标准方法, 他总结了几种胃含物分析方法, 讨论这些方法存在的困难和局限性, 并对此提出了改进方法。薛莹等[4]对国内外鱼类食性和食物网研究的内容和方法加以总结, 并在胃含物分析方法中提出了“单一指数”和“综合性指数”的概念。由于传统胃含物分析方法的局限性及科学技术的发展, 一些学者陆续提出了在传统胃含物分析方法的基础上运用稳定同位素、脂肪酸组成分析或DNA条形码等分子技术对其进行补充, 以更能反映客观实际[5-6]。尽管如此, 胃含物分析这一传统的方法在实验室或野外研究中仍是不可或缺的, 其可以给我们提供一个最直接的“快照”[7-8], 是一种能获取到水生生物摄食信息的最简单方法。
本文基于文献计量学的统计结果, 分析了在水生生态系统中运用胃含物分析及其与其他食性分析方法结合研究的发展动态, 并对其在食物网研究发展中的应用进行了梳理, 以期为今后的研究工作提供参考。
1 胃含物分析及其补充方法在水生生态系统研究中的发展动态胃含物分析(stomach content analysis)是一种传统物理性食物网科学的研究方法, 即通过测定动物胃肠中的食物组成, 获取动物近期的摄食情况, 是了解生物“吃了什么”最直接和最便捷的方法。随着科学研究的进步, 基于胃含物分析的水生生物摄食生态学研究方法可满足更为广泛和深层的研究需求。为了解摄食生态学研究方法的发展, 采用文献计量学的方法, 在Web of Science所有数据库和中国知网(CNKI)数据库中进行文献检索(查询日期: 2017年5月23日)。中文文献的检索关键主题词为“胃含物”, 检索结果中与水生生态系统摄食生态学研究有关的非会议论文有203篇; Web of Science数据库中的检索主题词为“stomach content analysis”, 再以“feeding ecology”在检索结果中搜索, 选择相关的研究方向和领域, 共检索出1801篇英文文献。将检索出的中英文文献按其采用的研究方法主要可分为4类, 即仅采用胃含物分析法(SCA)、胃含物分析法和稳定同位素技术结合(SCA+SIA)、胃含物分析法和脂肪酸组成分析结合(SCA+FA)以及胃含物分析法和DNA分子技术结合(SCA+DNA)。
早在1930年, 国外学者便意识到了解鱼类摄食对其洄游及养殖的重要性, 因此陆续出现了对抽取活鱼胃含物方法的介绍。如White[9]提出可用玻璃管抽取东溪鳟(Salvelinus fontinalis)胃含物的方法; Dubtes[10]直接将金属管插入鱼的食道中, 以抽取其胃含物进行分析。随着胃含物抽取法的改进及其分析法的便捷性, 胃含物分析法逐渐受到了学者们的青睐, 1980年前后已应用于水生生态系统的摄食生态学研究中, 并从1990年开始广泛得到了应用, 发展迅速, 至今仍是水生生态系统食性研究的最主要方法之一(图 1a)。随着胃含物分析法的广泛应用, 人们亦逐渐发现其存在着一定的局限性, 即其“分辨率”会受到消化程度的直接影响。因此学者们逐渐引入了稳定同位素技术、脂肪酸组成分析和DNA/RNA条形码技术作为补充研究方法。以稳定同位素应用于水生生态系统食物网的研究主要出现在1996年前后(图 1b); 国内在2005年才有记载, 总体呈波动上升的态势。与稳定同位素技术几乎同步出现的脂肪酸组成分析法, 却鲜见其与胃含物分析法结合使用的研究, 在国外每年的文献统计量仅为个位数, 发展平缓(图 1b); 在国内几乎未见记录(图 1a)。作为“最年轻”的DNA/RNA条形码技术, 其作为补充方法在水生生态系统食物网研究也有一定的发展(图 1)。
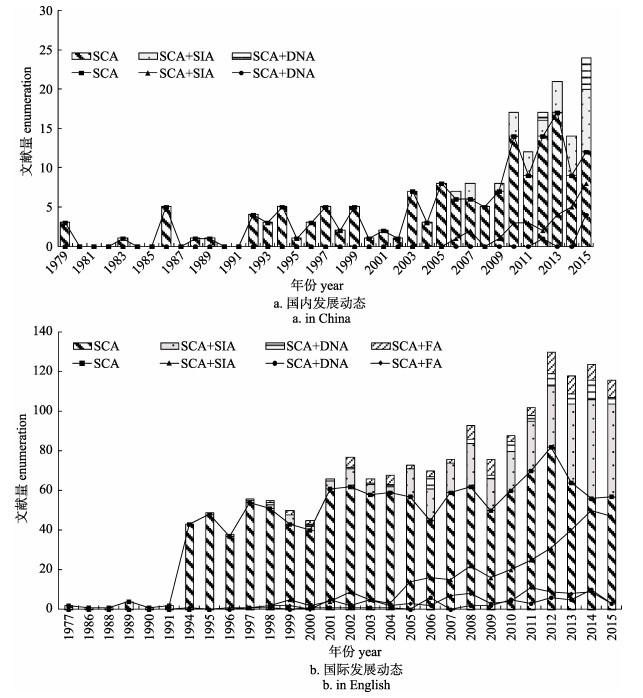
|
图 1 1977—2015年食物网研究方法的发展动态 SCA:仅采用胃含物分析法; SCA+SIA:胃含物分析法和稳定同位素技术结合; SCA+FA:胃含物分析法和脂肪酸组成分析结合; SCA+DNA:胃含物分析法和DNA条形码技术结合, 数据来源于Web of Science所有数据库(http://isiknowledge.com)和中国知网数据库(http://www.cnki.net). Fig.1 Enumeration of published literature of aquatic food web research method in 1977-2015 SCA: stomach content analysis; SCA+SIA: stomach content analysis combined with stable isotope; SCA+FA: stomach content analysis combined with fatty acid; SCA+DNA: stomach content analysis combined with DNA barcoding data from databases of the Web of Science (http://isiknowledge.com) and China National Knowledge Internet (http://www.cnki.net). |
总体而言, 在基于胃含物分析的水生生态系统的食性研究中, 单独采用胃含物分析法仍是主流, 所占比重最大; 其次应用的广泛程度由高到低依次是SCA+SIA、SCA+FA与SCA+DNA。采用这4种方法进行的研究总体呈递增的趋势, 但中国略滞后于国外。
2 传统胃含物分析法及其发展 2.1 传统胃含物分析法介绍传统胃含物分析的描述方法主要有出现法、数量法、体积法、重量法和主观观测法5种[3], 分别从出现频率、数量、体积和重量等角度描述某种食物在总食物中占据的比例。然而, 在样本量较小或者食物种类变化较大的情况下, 不同的方法将会产生不同的结果, 因此, Pinkas等[13]认为结合多种方法(指数)计算的结果更具代表性。薛莹等[4]将描述食物组成的指数形式分为两种:单一指数和综合性指数(表 1), 前者即与5种传统胃含物分析法一一对应, 后者指的是将多种指数合并修正成一个指数, 例如相对重要性指数(IRI)、绝对重要性指数(AI)等, 其原理为消除各个单一指数的偏差[11]。其中, 相对重要性指数及其百分比形式(IRI%)在胃含物分析中使用频率最高[12], IRI越高, 即代表该种食物对生物越重要。
|
|
表 1 描述食物组成的单一指数和综合性指数 Tab.1 Single index and comprehensive index in describing food composition |
胃含物分析主要研究生物的摄食组成和摄食偏好, 学者们通常采用表格法及统计图法的形式展示研究对象“吃什么”及其所摄食的食物在研究对象生活史过程中的重要性, 由于生活史过程的复杂性及多变性, 这一过程不断被细化。周婉霞等[16]分析了1982年6月至1983年12月鱼山、舟山、长江口南部的蓝圆鲹(Decapterus maruadsi)的胃含物组成, 发现不同月份的食物组成不同, 当龄鱼与成鱼之间存在着食性转换; 李军[17]研究了1982—1984年的渤海鲈(Lateolabrax japonicas)幼鱼和成鱼的摄食组成和摄食强度的月变化及相似性系数; 张波[18]通过聚类分析和列联表检验等方法对2004年1月—2005年10月黄海中部的高眼鲽(Cleisthenes herzensteini)摄食随体长的变化进行研究; Rosas-Luis等[19]分别从性别、成熟度及体长组三个角度对Santa Rosa 2014年5—12月的茎柔鱼(Dosidicus gigas)的摄食组成进行了分析; 这些研究说明对生物基于胃含物的研究处于不断细化的过程中, 以充分考虑其复杂的生活史过程、种群动态及栖息环境的多变性。
2.2 胃含物分析法的发展 2.2.1 在基础生物学中的应用在对水生生态系统中基于胃含物分析的食性研究的文献统计发现, 研究对象主要有鱼类、鸟类、头足类、两栖类、哺乳动物、爬行动物以及软体动物7种, 其中80%是鱼类的摄食生态学研究范畴, 主要包括两个方面:一是鱼类的摄食, 二是鱼类的生态地位及功能。颜云榕等[20]将鱼类的摄食研究归结为4个问题的解决, 即“吃什么”、“怎样吃”、“吃多少”以及“食物在有机体中的能量分配”。因此, 基于胃含物分析食性研究的应用方向主要为5个, 即研究生物的摄食组成、摄食行为/选择、摄食强度、生态转换效率及生态位。
2.2.2 新兴的应用方向目前摄食生态的研究越来越注重其对生产生活的指导意义, 已不再局限于对水生生态系统单一物种的食性研究。Beattie等[21]发现五大湖(Great Lakes)处于资源衰退状态的斑泥螈(Necturus maculosus)的胃含物出现了该地区的入侵物种虾虎鱼(Neogobius melanostomus、Orconectes rusticus)等, 表明其生态位发生变化, 虾虎鱼的入侵改变了群落结构。Rohan等[22]在没有足够的数据评估白令海章鱼的种群状况时, 利用太平洋鳕(Gadus microcephalus)长期的胃含物数据估算其自然死亡率和最小生物量, 以作为资源评估的基础。Bizzarro等[23]对美国太平洋海岸18种商业性底层鱼类的胃含物数据及生活史阶段进行了整合分析(meta-analysis), 综合考虑了4个变量:捕食者种类、生命阶段(如幼鱼、成鱼)、功能组(如底栖、中上层)和分类组(如岩鱼、比目鱼), 以划分底栖鱼类同资源种团(guild), 为当地基于栖息地的渔业管理提供信息。Navia等[24]重建热带食物网结构, 以评估人类压力对营养结构的影响, 采用文献计量的方法收集了1990—2009年间在Golfo de Tortugas(哥伦比亚)的80种捕食者的胃含物信息进行模型分析, 发现基于生态系统的渔业管理不仅应优先考虑高度重要的物种(如经济价值高等), 还应考虑在食物网中具有独特结构性质的物种, 因此提出在渔业资源的管理中需要更注重顶级捕食者的保护, 如鲨。
3 基于传统胃含物法食性研究的发展及应用传统胃含物分析法是基于形态学的基础上对样本进行分类, 尽管它可以进行不同角度下(如体长、性别和成熟度等)详细的摄食分析, 从而提供有价值的直接信息, 但受主观及客观条件限制, 分析结果具有很大的偶然性及局限性。传统胃含物分析法仅能提供最近的摄食信息(通常 < 24 h), 不能反映已消化和过去一段时间内的摄食信息[20], 且无法判断某种食物出现的偶然性和必然性[25]; 一些如胶质浮游动物等的食物能被快速消化, 从而难以辨认。一些学者在水生生物学的食性研究中陆续提出了一些新的方法, 并将这些方法与传统胃含物分析法同时运用到摄食生态学的研究中, 力求减小实验过程中的不确定性及保证实验结果的完整性。
3.1 稳定同位素技术稳定同位素技术(stable isotope analysis)目前已广泛应用于食物网研究中, 其应用原理是生物圈中重同位素和轻同位素含量存在着自然差异, 二者参与生物新陈代谢过程的速率不同。与胃含物分析相比, 对动物组织中的碳、氮稳定同位素分析可提供其在一段时间内的综合摄食信息[26]。每一物种间同位素比值存在着较大的差异; 同一物种不同个体间的同位素比值也反映了其独特的摄食行为信息; 测定不同的组织可反映不同的摄食周期[27-29]。研究发现, 消费者组织中的碳、氮稳定同位素比值(δ13C和δ15N)可用于确定初级生产力来源, 且随着营养级的增加, 消费者的δ13C和δ15N值与其食物之间分别呈现出+1‰和+3.4‰的富集水平[30-31]; 即消费者组织中的δ13C值与其食物较为接近, 可用于指示食物来源和栖息地特征; 而δ15N值可用于指示生物在其生活史过程中的营养水平[32-33]。在稳定同位素技术自20世纪80年代在生态领域中兴起至今的几十年间, 国内外主要应用其计算生物体内食物的贡献比例[34]、估算消费者营养级[35]、研究水生生物的食性转换[36]、水生生物的洄游与分布[37], 解决了以下几大问题:区分在食物网研究中的不同营养功能群[38-39], 在水生群落中比较生态位差异[40-41], 研究生物生活史过程中的食性变化[42-43], 消费者栖息地的可塑性[44]等。
2000年前后, 陆续有学者采用胃含物分析和稳定同位素技术两种方法同时进行摄食生态学研究[45-48], 更真实地反映生物的营养状况及其所占据的生态位, 完善及细化食物网结构。Fenton[45]对塔斯马尼亚岛(Tasmania)东南部的3种糠虾(Tenagomysis tasmaniae、Anisomysis mixta australis、Paramesopodopsis rufa)及采用围网捕捞的鱼类进行胃含物分析, 测定采样海域的藻类、浮游植物、浮游动物、底栖无脊椎动物和鱼类的δ13C和δD值(氢稳定同位素比值); 胃含物分析结果表明, 3种糠虾均为杂食性, 但其摄食组成具有差异; 稳定同位素比值反映出P. rufa的摄食以浮游生物为基础, 其δ13C和δD值与T. tasmaniae、A. mixta australis的同位素比值具有明显差异; 而T. tasmaniae、A. mixta australis相近的同位素比值显示二者具有相似的摄食成分, 但从胃含物分析中可以得出二者摄食的生物粒径不同。Reñones等[49]利用碳、氮稳定同位素和胃含物分析探究了地中海西部乌鳍石斑鱼(Epinephelus marginatus)随体长的摄食变化, 指出胃含物分析与稳定同位素技术二者数据之间具有良好的一致性, 二者联用对研究结果可起到协同作用。
当前, 稳定同位素技术已成为国内外学者在基于传统胃含物分析进行食性研究的基础上加入的最为常见的“新元素”。Reñones等[49]采用了出现频率和数量百分比的胃含物分析方法、以及结合δ13C和δ15N值的质量守恒模型综合分析了地中海西部乌鳍石斑鱼(E. marginatus)不同体长组的摄食组分(食物贡献比率); 根据质量守恒方程, n种稳定同位素可以确定n+1种食物源贡献比例[50]。在此基础上, Phillips等[51]提出了一种采用n种稳定同位素计算 > n+1种食物源的贡献比例, 并指出该方法计算出的结果只是一个大致的范围, 需要经过胃含物分析等“额外”方法对一些组合方案进行实际验证。
张宇美等[52]应用N稳定同位素和胃含物分析相关数据研究了2008年11月—2009年9月期间南海北部湾拖网渔获物中的二长棘犁齿鲷(Parargyrops edita Tanaka)的摄食习性和营养级, 根据饵料质量百分比及δ15N值分别估算出其营养级平均值为3.1和3.7; Hernández-Aguilar等[53]对2001年、2005年和2006年2—6月加利福尼亚西海岸的大青鲨(Prionace glauca)进行摄食生态学研究, 采用胃含物分析和稳定同位素技术, 分别计算出4.1和3.8的营养级估计值。胃含物分析和稳定同位素技术均可估算出生物的营养级, 但结果略有差异。Preciado等[54]在研究中指出采用这两种方法表示的营养级出现差异的原因可能是稳定同位素代表的是“上个月”的营养状况; 张宇美等[52]认为是因为对某种饵料生物的“高估”或“低估”而导致的结果差异。Zanden等[55]以淡水鱼类为对象对比研究了两种方法, 认为稳定同位素技术所得出的结果比胃含物分析更为准确。
Nakamura[56]为验证红树林是珊瑚礁鱼类的“保育场”这一观点, 对2004—2006年5—8月从日本南部石垣岛的红树林及其邻近珊瑚礁采集的焦黄笛鲷(Lutjanus fulvus)幼鱼及成鱼进行胃含物分析和稳定同位素研究; 实验中, 其将驼背笛鲷(L. gibbus)和紫红笛鲷(L. argentimaculatus)也作为研究对照, 前者整个生活史周期均栖息于珊瑚礁上(作为珊瑚礁栖息地的对照种), 后者栖息于红树林直至体长达到300 mm(作为红树林栖息地的对照种), 并对珊瑚礁和红树林栖息地表栖(如蟹类、虾类)和底栖(如游走的端足目生物)潜在食物源取样测定碳、氮稳定同位素。根据胃含物分析的数据及焦黄笛鲷(L. fulvus)的δ13C和δ15N值与驼背笛鲷(L. gibbus)、紫红笛鲷(L. argentimaculatus)潜在食物源的δ13C和δ15N值比较结果, 证明焦黄笛鲷(L. fulvus)的幼体期栖息于红树林中。同时采用胃含物分析和稳定同位素技术目前已被证明能有效地解决一些分布范围广、具有洄游性的生物的摄食生态学研究问题[57-58]。
“生态位(niche)”作为用于描述生物体在生态系统中所占据的功能和地位的重要概念着重强调了生物体本身作为单一个体与周围生物、环境相互关系。通过胃含物分析计算食物重叠指数[4], 或根据δ13C和δ15N值建立二维或多维的空间[59]以比较生物间是否在食物、空间利用等方面存在重叠, 确定是否存在竞争关系。Ho等[60]为了解两种三鳍鳚科鱼类条纹异齿鳚(Ecsenius lineatus)和红尾异齿鳚(E. namiyei)以及共同栖息于同一水域的蓝纹高身雀鲷(Stegastes fasciolatus)之间的食物重叠以及共存模式, 以δ13C和δ15N值分别作为二维空间坐标的横纵轴, 通过统计学分析每一物种的δ13C和δ15N平均值, 并将δ13C和δ15N值平均值表示于二维坐标轴中进行比较; Layman等[61]在此基础上根据δ13C和δ15N采用“凸包法”(convex hull)将生态位描绘于二维空间坐标中, 以使生态位“轮廓”更为直观和清晰。Jackson等[62]提出了一种基于贝叶斯模型的“稳定同位素贝叶斯椭圆法” (stable isotope bayesian ellipses, SIBER)计算生态位宽, 以弥补“凸包法”对样本量敏感等缺陷。“贝叶斯椭圆法”在水生生态系统摄食生态学研究中已广泛得到应用[41, 63]。
目前, 稳定同位素技术已实现了对单体化合物稳定同位素比值的测定, 称为特定化合物稳定同位素分析(compound-specific isotope analysis, CSIA)[64]。相较于传统的“整体”稳定同位素技术(bulk stable isotope analysis), 特定化合物稳定同位素分析可减少生物代谢和生理过程对同位素比值的干扰, 能反映更精准的摄食生态学信息。目前, 在水生食物网研究中, 特定化合物稳定同位素分析主要基于两种分子化合物:氨基酸[65]和脂肪酸[66]。在氨基酸中, 某些氨基酸的δ15N相较于其食物而言具有较强的分馏作用, 如谷氨酸、丙氨酸等“营养”氨基酸(trophic amino acid); 而一些氨基酸则相对稳定, 称为“源”氨基酸(source amino acid)[67]。鉴于此种特性, 可通过“营养”氨基酸与“源”氨基酸之间同位素比值估算同位素的分馏系数及在不需要基线生物同位素比值的前提下间接指示捕食者的营养层次。此外, 氨基酸的δ13C在必需氨基酸(essential amino acids)和非必需氨基酸(nonessential amino acids)中也有差异[68]。必需氨基酸较为保守, 而非必需氨基酸受到分解代谢等影响, 在合成期间可能发生不同程度的同位素分馏。因此, 对于特定氨基酸的稳定同位素分析能更本质的揭示引起生物组织中同位素分馏的物质和能量传递过程中的规律。
3.2 特征脂肪酸组成分析脂肪酸(fatty acid)是与生物关键生理和生物化学过程相关的脂质的主要成分, 占水生生物干重的2%~5%[69], 主要以三羧酸甘油酯和磷脂的形式存在。不同种类的水生生物具有其特殊的脂肪酸组成[70]。摄食被认为是影响组织脂肪酸组成的最重要的外部因素[71]。通过对生物体组织脂肪酸进行分析, 可以获取其食物信息, 因为不同种类, 甚至不同体组织之间脂质合成途径的差异均会导致生物体显示出不同特征的脂肪酸组成[72]。食物中的脂肪酸直接被生物同化吸收并保持其固有特性。因此, 通过分析生物脂肪酸信息可以反映最近一段时间内生物的摄食情况[73]。研究表明, 一些具有特定性质的脂肪酸已可作为“天然生物标志物”确定生物体的具体食物来源[74]。
与胃含物分析相比, 脂肪酸组成分析所需样本量较少, 且生物体内的脂肪酸是直接从食物中摄入并经过消化吸收, 在生物新陈代谢过程中结构基本保持不变, 是生物长期摄食活动积累的结果, 可依据选取测定的不同组织反映生物体长期或最近的摄食情况, 减少了胃含物分析结果的偶然性。因此, 有学者提出可将其作为胃含物分析的补充方法应用于摄食生态学研究[75-76]。
在水生生态系统中, 水生生物体脂肪酸碳原子个数一般为12~24个, 分为饱和脂肪酸和不饱和脂肪酸两大类, 而不饱和脂肪酸所占比例较大。某些脂肪酸只能从食物获取, 不能自身合成, 被称为必需脂肪酸, 如n-3和n-6系列高度不饱和脂肪酸[77]。因此, 浮游植物等低营养级生物的脂肪酸成分直接影响着高营养级消费者的脂肪酸组成。目前, 脂肪酸组成分析已被广泛地应用于低营养级和高营养级捕食者的食性研究中[78], 主要脂肪酸标志如表 2所示, 可用于区分种间资源利用模式差异(摄食或栖息地)[79]、确定生态位分化模式及食性的特异化程度[80]、探究食性在空间及时间上的变化[78]、定性和定量分析生物食性[81]。
|
|
表 2 水生食物网中应用的主要特征脂肪酸标志[74, 82-83] Tab.2 Signature fatty acids and corresponding sources within the aquatic food web |
Howell等[84]采用胃含物分析与脂肪酸组成分析对从大西洋Porcupine Seabight和波丘派恩河的海底平原Porcupine Abyssal Plain海区的大陆坡不同深度采集的9种深海海星的食性进行了研究, 通过对其脂肪酸聚类分析, 将海星分成悬浮食性、捕食者/腐食性和泥食性(mud ingester)。悬浮食性的海星对光合碳具有高度依赖性, 显示出进行光合的浮游植物的脂肪酸特征, 相对较高的C22:6 (n-3)可能来源于摄食的植物碎屑; 而泥食性的海星其C18:1 (n-7)和非亚甲基二烯酸(NMIDs)相对比例较高, 显示其摄食的成分主要为细菌; 捕食者/腐食性的海星脂肪酸组成介于二者之间。胃含物分析结果证实了利用脂肪酸确定食物资源利用模式准确性。
Pethybridge等[78]为了解澳大利亚塔斯马尼亚岛大陆坡超过400 m水深的水域主要兼捕渔获物的食物组成, 以特征脂肪酸的多变量分析作为胃含物分析的补充方法对采集的16种软骨鱼类的摄食组成进行分析, 测定其肌肉、肝和消化腺的脂肪酸成分。胃含物与脂肪酸组成分析结果显示, 每一种类有其特殊的摄食和生存模式(如银鲛目在该海域属于低营养级生物, 主要摄食底栖生物; 中型的角鲨科鲨鱼主要摄食处于营养级“中间位置”的鱼类和头足类; 猫鲨科鲨鱼主要摄食头足类等); 栖息于不同水层(如大陆坡上部和中部)的鲨鱼其摄食组成具有较大差异; 同一栖息水层的不同物种之间的摄食具有一定的相似性, 出现摄食生态位分化现象; 此外, 不同组织的脂肪酸组成不同, 但将肌肉、肝和消化腺三者的脂肪酸组成信息相结合可提供生物体长期连续、综合的摄食信息。
Iverson等[81]在人工喂养试验的基础上从定性和定量的角度估算自由放养的海鸟的摄食结构。他通过给海鸟长期投喂单一物种确定个体脂肪酸的校准系数, 依此系数在受控条件下对具有多种食物来源的海鸟脂肪酸特征值进行定量食物组成比例估算(定量脂肪酸特征分析, QFASA)。实验证明, 基于QFASA估算的食物组成结果与胃含物分析结果相似, 海鸟在物种间、时间和空间上存在着摄食差异, 并提出“动物的摄食是决定其脂肪酸组成的最重要因素”的观点。
3.3 DNA分子技术DNA条形码技术(DNA barcoding)是21世纪兴起的一项基于一段或几段短的、通用的标准DNA序列实现快速准确鉴定物种的技术[7], 已被广泛应用于海洋生物系统分类、种间亲缘关系鉴定、分子遗传多样性及肠道微生物多样性、食性分析等领域[85]。DNA条形码技术依据自然界中生物基因序列的唯一性, 通过提取胃含物中的DNA, 使用通用引物扩增目的片段, 纯化PCR (polymerase chain reaction, 聚合酶链式反应)产物后进行基因测序, 将序列结果与基因库进行比对分析, 从而获取相关物种信息。Hebert等[7]率先提出可以采用线粒体细胞色素C氧化酶I (cytochrome c oxidase subunit I, COI)基因作为区分生物种类的依据。Aguilar等[86]以DNA条形码技术为手段鉴定了美国切萨皮克湾(Chesapeake Bay)一种本地鲇和两种入侵鲇的胃含物种类, 成功鉴定出包括美洲狼鲈(Morone americana)、美洲鳗鲡(Anguilla rostrate)等在内的92%的物种, 其分类水平可具体至“种”。Sakaguchi等[87]分别采用形态学观察和DNA条形码技术比较分析了王鲑(Oncorhynchus keta)幼鱼的饵料丰度。结果显示, 在对幼鱼的胃含物分析中, 基于形态学观测到的36种食物种类中, 有11种未能被DNA条形码技术检测到; 而通过DNA条形码技术检测到80种食物种类中, 有61种无法通过形态学进行分辨。因此, DNA条形码作为补充技术可以大幅度提高胃含物分析的分辨率水平。
自DNA条形码技术兴起至今已有14年。随着DNA测序技术的进步和成本的降低, 以及基因数据库的逐步完善, 第二代测序出现(next-generation sequencing, NGS)进一步提高了食性分析的分辨率[88], 并产生了利用高通量测序技术获得条形码基因扩增子序列的方法, 即DNA Metabarcoding[89]。DNA Metabarcoding是基于DNA识别和高通量测序的结合, 将整个混合样本的DNA片段扩增后再进行高通量测序, 结合生物学信息自动识别出混合样品中的多个物种, 可减少采样的工作量, 最大限度地提高对半消化/已消化的组织残留物的物种鉴定水平。Harms-Tuohy等[90]将狮子鱼(Pterois volitans)的胃含物分成已消化(消化液)和未消化的2个样品, 分别采用DNA metabarcoding对其饵料种类进行测序, 将所得的序列与GenBank和Barcode of Life Database数据库比对, 发现在通过未消化成分鉴定出的39种饵料生物中, 仅有4种未能从消化液中鉴定。因此, DNA metabarcoding提供了一种可以解决“消化程度较高的胃含物鉴定”的方法。
4 问题及展望任何一种分析方法只是基于现有水平对生物摄食结构的“重建”, 其结果具有一定的偶然性和主观性。随着科技的发展, 摄食生态学研究方法在发生不断的革新, 以追求还原最真实的“影像”。在食性分析方法的选择上, 不同方法有其特殊的必要条件, 并具有一定的优势和局限性(表 3)。基于形态学观测的胃含物分析最重要的价值在于提供了胃含物基础的分类学数据[78], 但因其被观测到的大多数为难消化的硬组织成分, 如甲壳类, 而消化程度较高的软体动物则难以辨认, 导致结果出现偏差; 同时, 受研究学者知识水平的限制, 对胃含物种类的鉴定难以精确到较低的分类学水平(如属/种)。Kear[91]指出不要单独采用传统胃含物分析确定重要的食物。稳定同位素技术较胃含物分析在计算营养级、食性转换及生态位比较更为简单快捷, 并提出了一种基于δ13C和δ15N值定量估算饵料组成的模型, 可分析较长一段时间内饵料组成的可能性方案; 但受稳定同位素分馏和周转的特殊性及洄游基线差异等影响, 分析结果必须结合实际过程中生物用资源的特点, 即胃含物分析的结果; 并且分馏系数的不同选择会对结果造成很大差异。脂肪酸是生物长期摄食活动积累的结果, 其差异主要取决于动物的摄食, 是一种很好的“生物标志物”, 可对动物饵料进行定性分析, 近几年提出的QFASA模型促使脂肪酸组成分析实现了定量分析; 但该模型的提出是基于室内投喂实验的结果, 对于野外实验研究尚未足够成熟, 且脂肪酸结果定性范围较宽(如细菌、硅藻类等), 难以细化, 无法估算营养级等。DNA分子技术很大程度上提升了胃含物鉴定的分类学水平, 甚至鉴定消化程度较高的胃含物, 大大降低了样本量水平; 但其成本较高, 存在无法与公共基因库比对目标生物群体序列的可能, 且该方法定量分析仅限于饵料的出现频率, 无法从数量、质量各方面评估饵料生物的组成。因此, 在进行摄食生态学研究时, 首先应根据现有的技术、样品条件及研究方向, 综合考虑多方因素选取最合适的方法, 以减小实验偏差。
|
|
表 3 胃含物分析、稳定同位素技术、脂肪酸组成分析和DNA条形码技术4种方法适用性比较 Tab.3 The comparison among stomach content analysis, stable isotope analysis, fatty acid profile and DNA barcoding |
尽管胃含物分析具有很大的局限性, 但该方法作为获取“第一手资料”的途径, 在水生食物网研究中仍是不可缺少的一部分。国内外已有大量文献对胃含物的实验方法进行了介绍, 并对其进行了批判性分析, 但均无法克服该方法本身所存在的局限性(如消化)。DNA metabarcoding技术的发展尽管提供了一种可以解决“消化程度较高的胃含物鉴定”的方法, 但因其成本高昂, 通常只能作为一种辅助手段。目前, 国内学者大多依据相对重要性指数作为分析标准, 缺乏对多种“适用性指数”的选择及检验, 研究结果具有一定的偶然性。因此, 在进行胃含物数据结果分析时, 首先应针对生物的摄食及消化特征, 选择合适的“综合性指数”, 并应用统计学方法进行多重检验。同时, 胃含物所反映信息的“不完整性”要求在基于食物质量、数量或体积百分比观测的基础上, 采用混合模型等统计学方法定量估计食物组成, 以进行更好的样本估计, 从而提高胃含物分析的准确率, 进一步了解生物的摄食策略。
Bowen等[92]在对海洋哺乳动物的食性分析方法的回顾中指出, 每种方法因其特定的假设条件都可能导致食性分析过程中出现偏差, 最为可靠的方法是同时使用多种方法。国内外已开始采用两种或两种以上的方法进行食性研究, 以相互弥补各种方法存在的缺陷。洄游现象一直以来是摄食生态学研究的重点与难点, 其洄游路径决定了生物的摄食组成。目前采用的研究方法主要为稳定同位素分析, 受稳定同位素分馏等特性的影响, 其结果具有一定的偏差。而胃含物数据作为最直观反映生物摄食活动的依据, 可通过其食物组分判定生物摄食活动的区域, 进一步使用特征脂肪酸定性分析, 为制定准确的生物洄游路线提供必要的帮助。同时, 如特征氨基酸和脂肪酸单体同位素等特定化合物分析技术已首先在国外提出, 并逐步开始应用, 但国内对此的研究和应用却鲜有报道, 该种分析技术能更本质的揭示引起生物组织中同位素分馏的物质和能量传递过程中的规律, 能有效减小实验偏差。因此, 在摄食生态学研究中有必要进行特定化合物稳定同位素分析, 提升我国水生生态系统的研究水平。
对水生生态系统的摄食生态学研究的最终目的是为渔业资源的可持续利用提供科学依据。渔业资源的可持续利用不仅体现在单物种资源的养护及管理上, 更应从食物网全局着手, 掌握生物之间的生态关系。当前中国的渔业管理重心是进行基于生态系统的渔业管理(ecosystem based fisheries management), 中国渤海、东海和南海等近海海域已实现了海洋生态系统模型的初步构建, 但模型中准确的生物间摄食矩阵是决定模型输出结果正确性的关键。因此, 在后续的研究中, 应先从近海着手, 利用胃含物数据构建基本食物网结构; 通过稳定同位素技术, 特征脂肪酸标志物来校验生物间的摄食关系, 准确衡量海洋食物网结构及其变动规律。
| [1] |
Blaber S J M. Tropical Estuarine Fishes:Ecology, Exploitation and Conservation[M]. Oxford: Blackwell Science Ltd, 2000: 96-126.
|
| [2] |
Brodeur R D, Smith B E, Mcbride R S, et al. New perspectives on the feeding ecology and trophic dynamics of fishes[J]. Environmental Biology of Fishes, 2017, 100(4): 293-297. DOI:10.1007/s10641-017-0594-1 |
| [3] |
Hyslop E J. Stomach contents analysis-a review of methods and their application[J]. Journal of Fish Biology, 1980, 17(4): 411-429. DOI:10.1111/jfb.1980.17.issue-4 |
| [4] |
Xue Y, Jin X S. Review of the study on feeding habits of fishes and food webs[J]. Marine Fisheries Research, 2003, 24(2): 76-87. [薛莹, 金显仕. 鱼类食性和食物网研究评述[J]. 海洋水产研究, 2003, 24(2): 76-87.] |
| [5] |
Peterson B J, Fry B. Stable isotopes in ecosystem studies[J]. Annual Review of Ecology & Systematics, 1987, 18(1): 293-320. |
| [6] |
Dalsgaard J, St. John M, Kattner G, et al. Fatty acid trophic markers in the pelagic marine environment[J]. Advances in Marine Biology, 2003, 46: 225-340. DOI:10.1016/S0065-2881(03)46005-7 |
| [7] |
Howell D L, Pond D W, Billett W, et al. Feeding ecology of deep-sea seastars (Echinodermata:Asteroidea):a fatty-acid biomarker approach[J]. Marine Ecology Progress Series, 2004, 266(1): 103-110. |
| [8] |
Burns J M, Trumble S J, Castellini M A, et al. The diet of Weddell seals in McMurdo Sound, Antarctica as determined from scat collections and stable isotope analysis[J]. Polar Biology, 1998, 19(4): 272-282. |
| [9] |
White H C. Some observations on the eastern brook trout (S. fontinalis) of Prince Edward Island[J]. Transactions of the American Fisheries Society, 1930, 60(1): 101-108. DOI:10.1577/1548-8659(1930)60[101:SOOTEB]2.0.CO;2 |
| [10] |
Dubets H. Feeding habits of the largemouth bass as revealed by a gastroscope[J]. The Progressive Fish-Culturist, 1954, 16(3): 393-396. |
| [11] |
Bigg M A, Perez M A. Modified volume: a frequency-volume method to assess marine mammal food habits[C]//Beddington J R, Beverton R J H, Lavigne D M. Marine Mammals and Fisheries. London: George Allen & Unwin, 1985: 277-283.
|
| [12] |
Assis C A. A generalised index for stomach contents analysis in fish[J]. Scientia Marina, 1996, 60(2-3): 385-389. |
| [13] |
Pinkas L, Oliphant M S, Iverson I L K. Food habits of albacore, bluefin tuna and bonito in Californian waters[J]. California Fish & Game, 1971, 152(1): 1-105. |
| [14] |
George E L, Hadley W F. Food and habitat partitioning between rock bass (Ambloplites rupestris) and smallmouth bass (Micropterus dolomieui) young of the year[J]. Transactions of the American Fisheries Society, 1979, 108(3): 253-261. DOI:10.1577/1548-8659(1979)108<253:FAHPBR>2.0.CO;2 |
| [15] |
Natarajan A V, Jhingran A G. Index of preponderance-a method of grading the food elements in the stomach analysis of fishes[J]. Indian Journal of Fisheries, 1961, 8(1): 54-59. |
| [16] |
Zhou W X, Bo Z L. A study on the feeding habits of the round scad (Decapterus maruadsi) off Zhejiang[J]. Donghai Marine Science, 1986, 4(2): 65-74. [周婉霞, 薄治礼. 浙江近海蓝圆鲹食性的研究[J]. 东海海洋, 1986, 4(2): 65-74.] |
| [17] |
Li J. Food composition and feeding habits of Japanese sea bass in the Bohai sea[J]. Marine Sciences, 1994(3): 39-44. [李军. 渤海鲈鱼食物组成与摄食习性的研究[J]. 海洋科学, 1994(3): 39-44.] |
| [18] |
Zhang B. Diet composition and ontogenetic variation in feeding habits of Cleithenes herzensteini in central Yellow Sea[J]. Chinese Journal of Applied Ecology, 2007, 18(8): 1849-1854. [张波. 黄海中部高眼鲽的摄食及随体长的变化[J]. 应用生态学报, 2007, 18(8): 1849-1854.] |
| [19] |
Rosas-Luis R, Chompoy-Salazar L. Description of food sources used by jumbo squid Dosidicus gigas, (D'Orbigny, 1835) in Ecuadorian waters during 2014[J]. Fisheries Research, 2016, 173(2): 139-144. |
| [20] |
Yan Y R, Lu H S, Jin X S. Marine fish feeding ecology and food web:progress and perspectives[J]. Journal of Fisheries of China, 2011, 35(1): 145-153. [颜云榕, 卢伙胜, 金显仕. 海洋鱼类摄食生态与食物网研究进展[J]. 水产学报, 2011, 35(1): 145-153.] |
| [21] |
Beattie A M, Whiles M R, Willink P W. Diets, population structure, and seasonal activity patterns of mudpuppies (Necturus maculosus) in an urban, Great Lakes coastal habitat[J]. Journal of Great Lakes Research, 2017, 43(1): 132-143. DOI:10.1016/j.jglr.2016.11.004 |
| [22] |
Rohan S K, Buckley T W. Spatial and ontogenetic patterns of Pacific cod (Gadus macrocephalus, Tilesius) predation on octopus in the eastern Bering Sea[J]. Environmental Biology of Fishes, 2017, 100(4): 361-367. DOI:10.1007/s10641-016-0561-2 |
| [23] |
Bizzarro J J, Yoklavich M M, Wakefield W W. Diet composition and foraging ecology of U.S. Pacific coast groundfishes with applications for fisheries management[J]. Environmental Biology of Fishes, 2017, 100(4): 375-393. DOI:10.1007/s10641-016-0529-2 |
| [24] |
Navia A F, Cruz-Escalona V H, Giraldo A, et al. The structure of a marine tropical food web, and its implications for ecosystem-based fisheries management[J]. Ecological Modelling, 2016, 328(24): 23-33. |
| [25] |
Johnson S P, Schindler D E. Trophic ecology of Pacific salmon (Oncorhynchus spp.) in the ocean:a synthesis of stable isotope research[J]. Ecological Research, 2009, 24(4): 855-863. DOI:10.1007/s11284-008-0559-0 |
| [26] |
Fry B. Stable Isotope Ecology[M]. New York: Springer, 2006: 1-20.
|
| [27] |
Deniro M J, Epstein S. Mechanism of carbon isotope fractionation associated with lipid synthesis[J]. Science, 1977, 197(4300): 261-263. DOI:10.1126/science.327543 |
| [28] |
Haines E B, Montague C L. Food sources of estuarine invertebrates analyzed using, 13C/12C ratios[J]. Ecology, 1979, 60(1): 48-56. DOI:10.2307/1936467 |
| [29] |
Mcconnaughey T, Mcroy C P. Food-Web structure and the fractionation of carbon isotopes in the Bering Sea[J]. Marine Biology, 1979, 53(3): 257-262. DOI:10.1007/BF00952434 |
| [30] |
Minagawa M, Wada E. Stepwise enrichment of 15N along food chains:further evidence and the relation between δ15N and animal age[J]. Geochimica et Cosmochimica Acta, 1984, 48(5): 1135-1140. DOI:10.1016/0016-7037(84)90204-7 |
| [31] |
Goericke R, Fry B. Variations of marine plankton δ13C with latitude, temperature, and dissolved CO2 in the world ocean[J]. Global Biogeochemical Cycles, 1994, 8(1): 85-90. DOI:10.1029/93GB03272 |
| [32] |
Deniro M J, Epstein S. Carbon isotopic evidence for different feeding patterns in two hyrax species occupying the same habitat[J]. Science, 1978, 201(4359): 906-908. DOI:10.1126/science.201.4359.906 |
| [33] |
Minagawa M, Wada E. Stepwise enrichment of 15N along food chains[J]. Geochimica et Cosmochimica Acta, 1984, 48(5): 1135-1140. DOI:10.1016/0016-7037(84)90204-7 |
| [34] |
Phillips D L, Gregg J W. Source partitioning using stable isotopes:coping with too many sources[J]. Oecologia, 2003, 136(2): 261-269. DOI:10.1007/s00442-003-1218-3 |
| [35] |
Post D M. Using stable isotopes to estimate trophic position:models, methods, and assumptions[J]. Ecology, 2002, 83(3): 703-718. DOI:10.1890/0012-9658(2002)083[0703:USITET]2.0.CO;2 |
| [36] |
Zanden M J V, Menno H, Mark S R, et al. Application of stable isotope techniques to trophic studies of age-0 smallmouth bass[J]. Transactions of the American Fisheries Society, 1998, 127(5): 729-739. DOI:10.1577/1548-8659(1998)127<0729:AOSITT>2.0.CO;2 |
| [37] |
Fry B. Natural stable carbon isotope tag traces Texas shrimp migrations[J]. Fishery Bulletin, 1981, 79(2): 337-345. |
| [38] |
Carassou L, Kulbicki M, Nicola T J R, et al. Assessment of fish trophic status and relationships by stable isotope data in the coral reef lagoon of New Caledonia, southwest Pacific[J]. Aquatic Living Resources, 2008, 21(1): 1-12. DOI:10.1051/alr:2008017 |
| [39] |
Wyatt A S J, Waite A M, Humphries S. Stable isotope analysis reveals community-level variation in fish trophodynamics across a fringing coral reef[J]. Coral Reefs, 2012, 31(4): 1029-1044. DOI:10.1007/s00338-012-0923-y |
| [40] |
Ho C T, Kao S J, Dai C F, et al. Dietary separation between two blennies and the Pacific Gregory in northern Taiwan:evidence from stomach content and stable isotope analyses[J]. Marine Biology, 2007, 151(2): 729-736. DOI:10.1007/s00227-006-0517-8 |
| [41] |
Adams J N, Brodeur R D, Daly E A, et al. Prey availability and feeding ecology of juvenile chinook (Oncorhynchus tshawytscha) and coho (O. kisutch) salmon in the northern California current ecosystem, based on stomach content and stable isotope analyses[J]. Marine Biology, 2017, 164(5): 1-14. |
| [42] |
Kolasinski J, Frouin P, Sallon A, et al. Feeding ecology and ontogenetic dietary shift of yellowstripe goatfish Mulloidichthys flavolineatus (Mullidae) at Reunion Island, SW Indian Ocean[J]. Marine Ecology Progress, 2009, 386(1): 181-195. |
| [43] |
Plass-Johnson J G, McQuaid C D, Hill J M. Stable isotope analysis indicates a lack of inter-and intra-specific dietary redundancy among ecologically important coral reef fishes[J]. Coral Reefs, 2013, 32(2): 429-440. DOI:10.1007/s00338-012-0988-7 |
| [44] |
Ho C T, Fu Y C, Sun C L, et al. Plasticity of feeding habits of two Plectroglyphidodon Damselfishes on coral reefs in southern Taiwan:Evidence from stomach content and stable isotope analyses[J]. Zoological Studies, 2009, 48(5): 649-656. |
| [45] |
Fenton G E. Diet and predators of Tenagomysis tasmaniae Fenton, Anisomysis mixta australis (Zimmer) and Paramesopodopsis rufa Fenton from south-eastern Tasmania (Crustacea:Mysidacea)[J]. Hydrobiologia, 1996, 323(1): 31-44. DOI:10.1007/BF00020545 |
| [46] |
Nunes A J P, Gesteira T C V, Goddard S. Food ingestion and assimilation by the southern brown shrimp Penaeus subtilis, under semi-intensive culture in NE Brazil[J]. Aquaculture, 1997, 149(1-2): 121-136. DOI:10.1016/S0044-8486(96)01433-0 |
| [47] |
John W H, Townsend C R, Kevin J C. Diet of the crayfish Paranephrops zealandicus in bush and pasture streams:insights from stable isotopes and stomach analysis[J]. New Zealand Journal of Marine & Freshwater Research, 2002, 36(1): 129-142. |
| [48] |
Li Z Y, Guo X P, Jin X S, et al. Feeding habit of Erisphex potti from Yangtze River Estuary and adjacent sea in spring[J]. Journal of Fisheries of China, 2006, 30(5): 654-661. [李忠义, 郭旭鹏, 金显仕, 等. 长江口及其邻近水域春季虻鲉的食性[J]. 水产学报, 2006, 30(5): 654-661.] |
| [49] |
Reñones O, Polunin N V C, Goni R. Size related dietary shifts of Epinephelus marginatus, in a western Mediterranean littoral ecosystem:an isotope and stomach content analysis[J]. Journal of Fish Biology, 2002, 61(1): 122-137. DOI:10.1111/jfb.2002.61.issue-1 |
| [50] |
Schwarcz H P. Some theoretical aspects of isotope paleodiet studies[J]. Journal of Archaeological Science, 1991, 18(3): 261-275. DOI:10.1016/0305-4403(91)90065-W |
| [51] |
Phillips D L, Gregg J W. Source partitioning using stable isotopes:coping with too many sources[J]. Oecologia, 2003, 136(2): 261-269. DOI:10.1007/s00442-003-1218-3 |
| [52] |
Zhang Y M, Dai C T, Yan Y R, et al. Feeding habits and trophic level of crimson sea bream, (Parargyrops edita Tanaka) in the Beibu Gulf[J]. Journal of Fisheries of China, 2014, 38(2): 265-273. [张宇美, 代春桃, 颜云榕, 等. 北部湾二长棘犁齿鲷摄食习性和营养级[J]. 水产学报, 2014, 38(2): 265-273.] |
| [53] |
Hernández-Aguilar S B, Escobar-Sánchez O, Galván-Magaña F, et al. Trophic ecology of the blue shark (Prionace glauca) based on stable isotopes (δ13C and δ15N) and stomach content[J]. Journal of the Marine Biological Association of the United Kingdom, 2016, 96(7): 1403-1410. DOI:10.1017/S0025315415001393 |
| [54] |
Preciado I, Cartes J E, Punzón A, et al. Food web functioning of the benthopelagic community in a deep-sea seamount based on diet and stable isotope analyses[J]. Deep Sea Research Part Ⅱ:Topical Studies in Oceanography, 2017, 137: 56-68. DOI:10.1016/j.dsr2.2016.07.013 |
| [55] |
Zanden M J V, Cabana G, Rasmussen J B. Comparing trophic position of freshwater fish calculated using stable nitrogen isotope ratios (δ15N) and literature dietary data[J]. Canadian Journal of Fisheries & Aquatic Sciences, 1997, 54(5): 1142-1158. |
| [56] |
Nakamura Y. Evidence of ontogenetic migration from mangroves to coral reefs by black-tail snapper Lutjanus fulvus:stable isotope approach[J]. Marine Ecology Progress, 2008, 355(1): 257-266. |
| [57] |
Power M, Power G, Caron F, et al. Growth and dietary niche in Salvelinus alpinus, and Salvelinus fontinalis, as revealed by stable isotope analysis[J]. Environmental Biology of Fishes, 2002, 64(1-3): 75-85. |
| [58] |
Morinière E C D L, Pollux B J A, Nagelkerken I, et al. Ontogenetic dietary changes of coral reef fishes in the mangrove-seagrass-reef continuum:stable isotopes and gut-content analysis[J]. Marine Ecology Progress, 2003, 246(1): 279-289. |
| [59] |
Bearhop S, Adams C E, Waldron S, et al. Determining trophic niche width:a novel approach using stable isotope analysis[J]. Journal of Animal Ecology, 2004, 73(5): 1007-1012. DOI:10.1111/j.0021-8790.2004.00861.x |
| [60] |
Ho C T, Kao S J, Dai C F, et al. Dietary separation between two blennies and the Pacific gregory in northern Taiwan:evidence from stomach content and stable isotope analyses[J]. Marine Biology, 2007, 151(2): 729-736. DOI:10.1007/s00227-006-0517-8 |
| [61] |
Layman C A, Arrington D A, Montana C G, et al. Can stable isotope ratios provide for community-wide measures of trophic structure?[J]. Ecology, 2007, 88(1): 42-48. DOI:10.1890/0012-9658(2007)88[42:CSIRPF]2.0.CO;2 |
| [62] |
Jackson A L, Inger R, Parnell A C, et al. Comparing isotopic niche widths among and within communities:SIBER-Stable Isotope Bayesian Ellipses in R[J]. Journal of Animal Ecology, 2011, 80(3): 595-602. DOI:10.1111/jane.2011.80.issue-3 |
| [63] |
Sorell J M, Varela J L, Goñi N, et al. Diet and consumption rate of Atlantic bluefin tuna (Thunnus thynnus) in the Strait of Gibraltar[J]. Fisheries Research, 2017, 188: 112-120. DOI:10.1016/j.fishres.2016.12.012 |
| [64] |
Krummen M, Hilkert A W, Juchelka D, et al. A new concept for isotope ratio monitoring liquid chromatography/mass spectrometry[J]. Rapid Communications in Mass Spectrometry, 2004, 18(19): 2260-2266. DOI:10.1002/rcm.v18:19 |
| [65] |
Fantle M S, Dittel A I, Schwalm S M, et al. A food web analysis of the juvenile blue crab, Callinectes sapidus, using stable isotopes in whole animals and individual amino acids[J]. Oecologia, 1999, 120(3): 416-426. DOI:10.1007/s004420050874 |
| [66] |
Hammer B T, Fogel M L, Hoering T C. Stable carbon isotope ratios of fatty acids in seagrass and redhead ducks[J]. Chemical Geology, 1998, 152(1-2): 29-41. DOI:10.1016/S0009-2541(98)00094-1 |
| [67] |
Mcclelland J W, Montoya J P. Trophic relationships and the nitrogen isotopic composition of amino acids in plankton[J]. Ecology, 2002, 83(8): 2173-2180. DOI:10.1890/0012-9658(2002)083[2173:TRATNI]2.0.CO;2 |
| [68] |
Boecklen W J, Yarnes C T, Cook B A, et al. On the use of stable isotopes in trophic ecology[J]. Annual Review of Ecology Evolution & Systematics, 2011, 42(1): 411-440. |
| [69] |
Olsen Y. Lipids and Essential Fatty Acids in Aquatic Food Webs:What Can Freshwater Ecologists Learn from Mariculture? Lipids in Freshwater Ecosystems[M]. New York: Springer, 1999: 161-202.
|
| [70] |
Connell J J. Advances in fish science and technology[M]. London: Fishing News Books Ltd., 1980.
|
| [71] |
Steven X C, Lisa A K, Stefano M. Stock Identification Methods (Second Edition)[M]. Elsevier, 2014: 235-256.
|
| [72] |
Sargent J R. Structure, metabolism and function of lipids in marine organisms[J]. Biochemical & Biophysical Perspectives in Marine Biology, 1976, 3(1): 149-212. |
| [73] |
Iverson S J, Field C, Bowen W D, et al. Quantitative fatty acid signature analysis:a new method of estimating predator diet[J]. Ecological Monographs, 2004, 74(2): 211-235. DOI:10.1890/02-4105 |
| [74] |
Xu Q, Yang H S. Fatty acid biomarker and its application in marine trophic relation studies[J]. Acta Oceanologica Sinica, 2011, 33(1): 1-6. [许强, 杨红生. 脂肪酸标志物在海洋生态系统营养关系研究中的应用[J]. 海洋学报, 2011, 33(1): 1-6. DOI:10.3969/j.issn.1009-5470.2011.01.001] |
| [75] |
Iverson S J, Frost K J, Lowry L F. Fatty acid signatures reveal fine scale structure of foraging distribution of harbor seals and their prey in Prince William Sound, Alaska[J]. Marine Ecology Progress, 1997, 151(1): 255-271. |
| [76] |
Bradshaw C J, Hindell M A, Best N J, et al. You are what you eat:describing the foraging ecology of southern elephant seals (Mirounga leonina) using blubber fatty acids[J]. Proceedings Biological Sciences, 2003, 270(1521): 1283-1292. DOI:10.1098/rspb.2003.2371 |
| [77] |
Bell J G, Ghioni C, Sargent J R. Fatty acid compositions of 10 freshwater invertebrates which are natural food organisms of Atlantic salmon parr (Salmo salar):a comparison with commercial diets[J]. Aquaculture, 1994, 128(3-4): 301-313. DOI:10.1016/0044-8486(94)90319-0 |
| [78] |
Pethybridge H, Daley R K, Nichols P D. Diet of demersal sharks and chimaeras inferred by fatty acid profiles and stomach content analysis[J]. Journal of Experimental Marine Biology & Ecology, 2011, 409(1-2): 290-299. |
| [79] |
Chavarie L, Howland K, Gallagher C, et al. Fatty acid signatures and stomach contents of four sympatric Lake Trout:assessment of trophic patterns among morphotypes in Great Bear Lake[J]. Ecology of Freshwater Fish, 2016, 25(1): 109-124. DOI:10.1111/eff.2016.25.issue-1 |
| [80] |
Raclot T, Groscolas R, Cherel Y. Fatty acid evidence for the importance of myctophid fishes in the diet of king penguins, Aptenodytes patagonicus[J]. Marine Biology, 1998, 132(3): 523-533. DOI:10.1007/s002270050418 |
| [81] |
Iverson S J, Springer A M, Kitaysky A S. Seabirds as indicators of food web structure and ecosystem variability:qualitative and quantitative diet analyses using fatty acids[J]. Marine Ecology Progress, 2007, 352(1): 235-244. |
| [82] |
Zhang X F, Liu Y, Li Y, et al. Identification of the geographical origins of sea cucumber (Apostichopus japonicus) in northern China by using stable isotope ratios and fatty acid profiles[J]. Food Chemistry, 2017, 218(1): 269-276. |
| [83] |
Every S L, Pethybridge H R, Crook D A, et al. Comparison of fin and muscle tissues for analysis of signature fatty acids in tropical euryhaline sharks[J]. Journal of Experimental Marine Biology and Ecology, 2016, 479: 46-53. DOI:10.1016/j.jembe.2016.02.011 |
| [84] |
Howell D L, Pond D W, Billett W, et al. Feeding ecology of deep-sea seastars (Echinodermata:Asteroidea):a fatty-acid biomarker approach[J]. Marine Ecology Progress Series, 2003, 255(1): 193-206. |
| [85] |
Xi X Q, Bao B L, Zhang S Y. Application of DNA barcoding in species analysis of fish stomach content[J]. Journal of Shanghai Ocean University, 2015, 24(2): 203-210. [席晓晴, 鲍宝龙, 章守宇. DNA条形码在鱼类胃含物种类鉴定中的应用[J]. 上海海洋大学学报, 2015, 24(2): 203-210.] |
| [86] |
Aguilar R, Ogburn M B, Driskell A C, et al. Gutsy genetics:identification of digested piscine prey items in the stomach contents of sympatric native and introduced warm water catfishes via DNA barcoding[J]. Environmental Biology of Fishes, 2017, 100(4): 325-336. DOI:10.1007/s10641-016-0523-8 |
| [87] |
Sakaguchi S O, Shimamura S, Shimizu Y, et al. Comparison of morphological and DNA-based techniques for stomach content analyses in juvenile chum salmon Oncorhynchus keta:a case study on diet richness of juvenile fishes[J]. Fisheries Science, 2017, 83(1): 47-56. DOI:10.1007/s12562-016-1040-6 |
| [88] |
Pompanon F, Deagle B E, Symondson W O C, et al. Who is eating what:diet assessment using next generation sequencing[J]. Molecular Ecology, 2012, 21(8): 1931-1950. DOI:10.1111/j.1365-294X.2011.05403.x |
| [89] |
Taberlet P, Coissac E, Pompanon F, et al. Towards next-generation biodiversity assessment using DNA metabarcoding[J]. Molecular Ecology, 2012, 21(8): 2045-2050. DOI:10.1111/j.1365-294X.2012.05470.x |
| [90] |
Harms-Tuohy C A, Schizas N V, Appeldoorn R S. Use of DNA metabarcoding for stomach content analysis in the invasive lionfish (Pterois volitans) in Puerto Rico[J]. Marine Ecology Progress, 2016, 558(1): 181-191. |
| [91] |
Kear A J. The diet of Antarctic squid:comparison of conventional and serological gut contents analyses[J]. Journal of Experimental Marine Biology & Ecology, 1992, 156(2): 161-178. |
| [92] |
Bowen W D, Iverson S J. Methods of estimating marine mammal diets:A review of validation experiments and sources of bias and uncertainty[J]. Marine Mammal Science, 2013, 29(4): 719-754. |
 2018, Vol. 25
2018, Vol. 25

