2. 青岛海洋科学与技术国家实验室, 海洋渔业科学与食物产出过程功能实验室, 山东 青岛 266071
2. Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071, China
近岸海洋系统是全球最重要的生态系统之一, 具有极高的经济价值和生态价值[1], 拥有众多水产养殖品种。盐度的季节性波动以及潮汐波动是该地区的一个重要特征[2]。同时由于近十年来, 全球变暖导致海洋环境急剧变化, 冰川加速消融, 雨季降水增多导致沿岸带海水盐度显著降低[3]。生活于该地区的海洋生物对于该地区不同盐度的适应机制以及适应能力有所不同[1]。对于鱼类, 甲壳类等渗透调节者(osmoregulator)而言, 可以通过活跃的离子转运机制维持细胞外液(ECF)的渗透压在一个稳定的范围[4-5], 而对于牡蛎等无脊椎动物而言, 它们缺乏渗透压调节能力, 其渗透压只能与外界环境渗透压保持一致, 又被称为渗透压随变者(osmoconformer)[6]。低盐会影响牡蛎的正常生理代谢包括摄食[7]、免疫活性[8]及生长[9], 甚至导致大规模死亡[10]。
岩牡蛎自然分布于日本、韩国和中国沿海, 附着生活在低潮线至水深20 m处的岩礁上[11]。与生活在潮间带的主要养殖种类长牡蛎(Crassostrea gigas)相比, 对盐度变化更加敏感[12]。牡蛎抵抗外界盐度变化主要采取两种策略, 在可耐受的范围内通过游离氨基酸等渗透调节因子将自身渗透压维持与周围海水水平一致, 超过耐受范围后会通过闭壳活动断绝与周围海水的交换来抵抗低盐环境, 与之相关的还会触发相关免疫反应, 热休克蛋白等抗凋亡因子的表达升高[11-13]。
实时荧光定量PCR (quantitative real-time PCR, qRT-PCR)是一种常用的检测基因表达的方法, 其灵敏度高、灵活性强、重复性好, 已被广泛应用于分子医学、生物科学、微生物学以及诊断学等领域[22]。然而, 由于RNA的质量和完整性和引物的特异性等差异会影响实验的准确性, 选择在不同组织及不同处理下具有相对高且稳定表达的合适的参考基因能够减少样本间的系统误差[16]。内参基因如肌动蛋白β-actin (ACT), 延长因子elongation factor-1α (EF1Α), 微管蛋白β-tubulin (TUB)已被广泛应用于长牡蛎的研究中[17]。然而, 目前国内外关于岩牡蛎的研究仅仅局限在生物学, 形态学等基础层面[11], 关于岩牡蛎低盐胁迫应答相关分子机制的研究几乎空白。而内参基因的筛选验证更是挖掘相关功能基因等研究的首要基础工作。
本研究评估了5个内参基因在正常生理状况下的4个组织和低盐胁迫下的鳃中的稳定性, 确定了合适的参考基因, 并通过qRT-PCR方法使岩牡蛎靶基因的表达正常化。这是首次针对岩牡蛎进行的内参基因筛选工作, 这些内参基因将为进一步研究与低盐胁迫相关的功能基因的表达模式提供有效信息, 并将有助于研究该物种低盐胁迫应答相关分子机制。
1 材料与方法 1.1 实验材料岩牡蛎亲本选自日本佐渡岛, 于中国山东乳山海域进行子代的养殖, 实验所用个体为2016年采集的一龄岩牡蛎。运回实验室后, 立即取其外套膜、鳃、闭壳肌和内脏团置于液氮中, 后转移至‒80℃低温冰箱保存。
1.2 实验方法实验共设置3个盐度梯度, 即盐度10、20和30(对照), 每个梯度设3个平行组, 各盐度处理组均由自然砂滤海水和曝气24 h自来水(低盐组)配制而成。实验所用岩牡蛎先于70 L塑料桶中暂养一周, 暂养海水盐度为30。暂养结束后直接放置到设置的不同盐度梯度, 每个平行组随机放入3个体, 未发现死亡个体, 胁迫一周后取样。暂养及胁迫期间连续充氧, 每天投喂饵料, 吸底及换水一次。
1.3 RNA提取及cDNA合成各组织总RNA用TRIzol试剂盒提取, Nanodrop核酸分析仪测定总RNA的浓度和纯度。用1.2%的琼脂糖凝胶电泳检测总RNA的完整性。cDNA链的合成按照PrimerScript RT reagent kit (TaKaRa)说明书要求进行。1 µg RNA反转得到cDNA第一链并最终获得20 µL cDNA。
1.4 内参基因的选择和q-PCR选择延伸因子(EF1A)、甘油-3-磷酸脱氢酶基因(GAPDH)、核异质核糖核蛋白(RO21)、α微管蛋白(TUA)和β微管蛋白(TUB)5个候选的内参基因, 引物通过primer 5.0 (http://www.premierbiosoft.com/)对岩牡蛎的转录组数据(Genbank accession: GGUV00000000)进行设计, 通过琼脂糖凝胶电泳和熔解曲线分析检测引物特异性(表 1)。使用Roche480进行定量分析。10 µL体系为: 1 µL模板cDNA, 0.3 µL正反引物(10 µmol/L), 5µL 2× SYBR EVE PCR Mix (abm), 3.4 µL超纯水。扩增反应程序为: 95℃预变性5 min; 95℃变性5 s, 60℃(不同引物退火温度不同)退火30 s, 72℃延伸30 s, 40个循环, 每个样品重复3次。引物扩增效率E通过对5倍梯度稀释的cDNA进行q-PCR所得Ct值绘制标准曲线, 根据公式E=(10-1/slope-1)× 100%得到。
|
|
表 1 候选内参基因的引物序列、扩增片段长度及扩增效率 Tab.1 Primer sequences, product sizes and PCR efficiencies of the selected genes |
为了在相同的实验条件下选择最优的参考基因进行精确归一化, 使用geNorm、NormFinder和BestKeeper评估5个候选基因的表达稳定性, 并对参考基因进行排序。在geNorm和NormFinder中, 需要将扩增循环数(Ct)的原始数据转换为相对量后进行计算, BestKeeper直接对Ct值通过两两关联进行分析。
2 结果与分析 2.1 候选内参基因的普通PCR扩增、标准曲线绘制及引物的扩增效率以不同组织和不同盐度胁迫下的岩牡蛎的鳃的总RNA反转录成的cDNA为模板进行普通PCR扩增, 以1.5%的琼脂糖电泳检测。各候选内参基因的扩增产物片段大小与预期相同, 条带清晰、无杂带和引物二聚体, 特异性良好。对梯度稀释的cDNA进行qPCR绘制标准曲线, 并根据标准曲线的斜率值和计算公式获得各候选内参基因的扩增效率为99.99%~104.75% (表 1)。不同组织中各候选内参基因Ct值变化范围是12.65~ 28.88, 其中GAPDH变化最小, TUA变化最大, EF1A表达水平最高(Ct值最小), RO21表达水平最低(图 1)。这些候选内参基因Ct值在不同盐度胁迫下鳃中的变化趋势与前述大致相同, 变化更小, 表达更稳定。
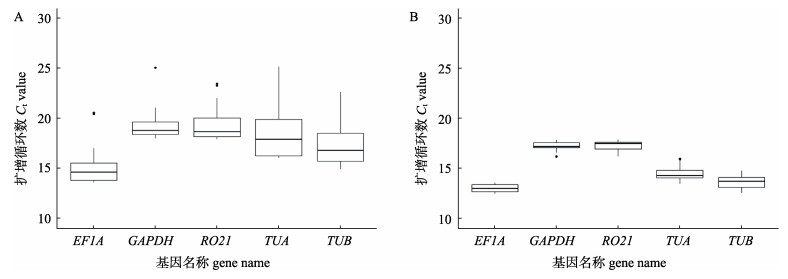
|
图 1 不同组织(A)和不同盐度胁迫(B)下候选内参基因的表达水平 Fig.1 Expression levels of candidate reference genes in different tissues (A) and salinity stress (B) |
geNorm软件利用表达稳定值M对候选内参基因的表达稳定性进行评估, M值为任意2个候选内参基因组合的表达水平比值的对数值, M值越小, 基因表达的稳定性越高。geNorm软件会依次排除最不稳定的基因, 直到仅剩2个为止。在不同组织中, 各候选内参基因的M值由大到小依次为GAPDH > TUA > TUB > RO21/EF1A, 说明在不同组织中表达水平最稳定的是RO21和EF1A (0.57)。而在不同盐度胁迫的鳃中, 各候选基因依M值从大到小排序为TUA > TUB > EF1A > RO21/ GAPDH, 说明在不同盐度胁迫下的鳃中表达水平最稳定的是RO21和GAPDH (0.15) (图 2)。
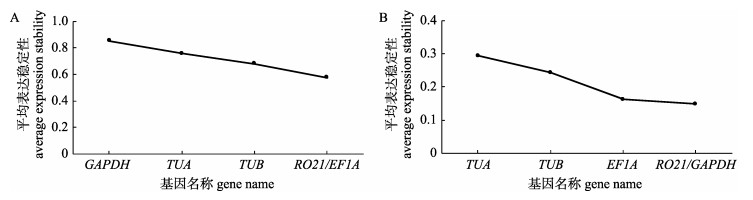
|
图 2 geNorm分析不同组织(A)和不同盐度胁迫(B)下候选内参基因的稳定性 Fig.2 Average expression stability values of the candidate reference genes in different tissues (A) and under salinity stress (B) analyzed by geNorm |
NormFinder软件同样利用表达稳定值M对基因进行排序, 不同于geNorm算法的是, 它是基于数学模型计算基因表达的稳定值。与geNorm的结果相似, 只是略有差异。在不同组织中, EF1A是最稳定的内参基因(0.22), GAPDH最不稳定(0.60)。在不同盐度胁迫下的鳃中, RO21是最稳定的内参基因(0.10), TUA最不稳定(0.23) (图 3)。
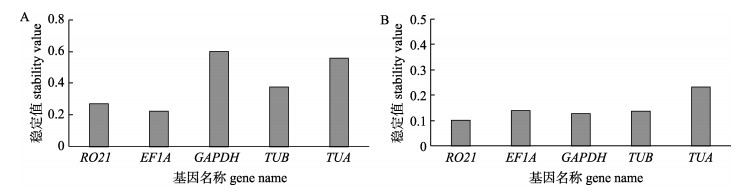
|
图 3 NormFinder分析不同组织(A)和不同盐度胁迫下候选内参基因的稳定性 Fig.3 Average expression stability values of the candidate reference genes in different tissues (A) and under salinity stress (B) analyzed by NormFinder |
BestKeeper算法通过计算和比较变异系数(CV)和标准差(SD)来估计候选基因的表达稳定性。根据SD值的大小选择最稳定的内参基因。候选内参基因的SD值与基因稳定性呈负相关关系, 说明SD值越低, 稳定性越高。与geNorm和NormFinder算法分析结果不同的是, 在不同组织中和不同盐度胁迫下的鳃中, BestKeeper均算出GAPDH为最稳定的基因, TUA最不稳定(表 2)。
|
|
表 2 BestKeeper分析候选内参基因的表达稳定性 Tab.2 Expression stability of reference genes analyzed by BestKeeper |
geNorm能够通过计算Vn/n+1分析最适内参基因数目。当Vn/n+1 < 0.15时, n即为所需的内参基因的数量。在不同组织中, 还需更多的内参基因才能获得理想的目标基因定量结果, 而在不同盐度胁迫下的鳃中, 1~2个内参基因便能获得理想结果(图 4)。
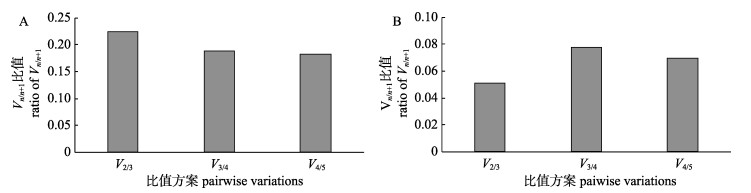
|
图 4 geNorm分析不同组织(A)和不同盐度胁迫(B)下合适内参基因数目 Fig.4 The number of reference genes calculated by geNorm in different tissues (A) and under salinity stress (B) analyzed by geNorm |
根据3种算法的排序结果, 得到参考基因的各自排序。计算每个参考基因排序的几何平均值[20]作为最后综合排序。在不同组织中, 各候选内参基因从最稳定到最不稳定依次为: EF1A > RO21 > GAPDH > TUB > TUA; 在不同盐度胁迫下的鳃中, GAPDH > RO21 > EF1A > TUB > TUA (表 3)。
|
|
表 3 候选内参基因在geNorm、NormFinder、BestKeeper 3种方法中的排名和综合排名 Tab.3 Ranking of reference genes by geNorm, NormFinder, BestKeeper and overall rank |
qRT-PCR是一种高灵敏度、高特异性和高重复性的基因表达分析方法。最佳的内参基因在不同的细胞、组织、物种和不同的实验条件下不断转录表达。然而, 在所有情况下满足所有条件的最稳定的参考基因几乎不存在。在软体动物中最常见的内参基因肌动蛋白actin (ACT)的表达稳定性受到贻贝发育阶段的很大影响[18], 许多内参基因在没有被评估其稳定性后便被直接使用, 影响实验结果的准确性。选择合适的内参基因是qRT-PCR中准确分析靶基因表达水平的前提。为了避免在基因表达谱分析中出现不必要的错误, 本文通过3种方法(geNorm、NormFinder和BestKeeper)分析了5个候选内参基因在不同组织和盐度胁迫下的表达稳定性。然而, 不同方法计算出的结果不尽相同, 这一现象在许多研究中同样发现[16-18], 这可能与这三种方法所应用的计算方式不同有关。目前, 并没有一致的意见认为哪个方法更加准确。
综合来看, 不同组织和与不同盐度处理下的鳃中5个候选内参基因的表达丰度大致相同且表达水平较高, 但不同组织中各内参基因的表达差异更大。这一现象在鲈的研究中也有报道[20], 表明与不同盐度处理相比, 不同组织对内参基因的稳定性的影响更大, 这可能与不同组织承担的生命活动以及各项生命活动的活跃程度, 基因的选择性表达有关。而对于不同盐度处理下的同一组织, 由于内参基因均是参与重要生命活动所必需的管家基因[21], 盐度处理对其的影响较小, 其稳定性更高。因此, 在对岩牡蛎分析不同组织中的功能基因的表达情况时, 需要选取数目更多的内参基因从而获得准确可靠的定量结果[20]。而在分析靶基因在不同处理下的单一组织的表达情况时, 1~2个内参基因便可以得到理想的结果。
本实验所选择的5个候选内参基因均为细胞中维持正常生命活动的管家基因, 作为候选内参基因在许多物种中检测其表达稳定性。各内参基因的表达稳定性在不同物种、不同组织、不同发育阶段及不同环境条件下的表现并不相同。GAPDH主要参与糖酵解过程, 并涉及核RNA输出、DNA复制、DNA修复、胞外膜融合等[22], 在太平洋牡蛎的不同组织[23]以及被寄生虫感染前后的机体中[24]、不同温度胁迫下的硅藻中[25]表达稳定。TUA和TUB编码微管蛋白中最常见的两个家族α-微管蛋白和β-微管蛋白, 是细胞骨架的基本组成成分。虽然在鲽[26]和草本植物[27]的不同发育阶段被发现表达稳定, 但在本研究中利用三种方法对其稳定性在不同组织, 不同盐度处理下进行评估时均发现其稳定性较差。研究发现该组分易受到盐度等环境因子的影响[28], 同时其含量在不同组织中也不尽相同[29]。EF1A是G蛋白家族中EF-1 (elongation factor-1)四个亚基中的一个, 在蛋白质翻译中起关键作用[30], 发现在太平洋牡蛎成体受病毒感染前后稳定性较好, 但在幼虫中较差[17]。RO21为异质性核糖核蛋白, 在mRNA相关过程的调控中发挥重要作用, 包括mRNA的激活, 定位等过程[31], 在软体动物的性腺、鳃、内脏团等组织中大量表达且表达稳定[32]。
本研究使用3个常用方法评估了岩牡蛎5个内参基因的稳定性, 发现GAPDH和RO21是在不同盐度下鳃组织中最合适的单一内参基因; 而在外套膜、鳃、闭壳肌和内脏团间, 则最好选择多个内参基因联合分析以获得较为准确的定量结果。本研究首次提供了岩牡蛎内参基因稳定性的基础数据, 可用于以后岩牡蛎功能基因的表达标准化分析。
| [1] |
Harley C. The impacts of climate change in coastal marine systems[J]. Ecology Letters, 2010, 9(2): 228-241. |
| [2] |
Drouin G, Himmelman J H, Pierre B. Impact of tidal salinity fluctuations on echinoderm and mollusc populations[J]. Canadian Journal of Zoology, 1985, 63(6): 1377-1387. DOI:10.1139/z85-207 |
| [3] |
Philippart C J M, Anadón R, Danovaro R, et al. Impacts of climate change on European marine ecosystems: Observations, expectations and indicators[J]. Journal of Experimental Marine Biology and Ecology, 2011, 400(1): 52-69. |
| [4] |
Bourque C W. Central mechanisms of osmosensation and systemic osmoregulation[J]. Nature Reviews Neuroscience, 2008, 9(7): 519-531. DOI:10.1038/nrn2400 |
| [5] |
Bruckner P, van der Rest M. Structure and function of cartilage collagens[J]. Microscopy Research & Technique, 2010, 28(5): 378-384. |
| [6] |
Mcallen R, Walker D, Taylor A. The environmental effects of salinity and temperature on the oxygen consumption and total body osmolality of the marine flatworm Procerodes littoralis[J]. Journal of Experimental Marine Biology and Ecology, 2002, 268(1): 103-113. DOI:10.1016/S0022-0981(01)00381-1 |
| [7] |
Ma H M, Liu X W, Mai K S, et al. Effects of abrupt salinity decline on anti-disease ability of scallop Chlamys farrer[J]. Chinese High Technology Letters, 2006, 16(7): 746-751. [马洪明, 刘晓伟, 麦康森, 等. 盐度突降对栉孔扇贝(Chlamys farreri)抗病力指标的影响[J]. 高技术通讯, 2006, 16(7): 746-751. DOI:10.3321/j.issn:1002-0470.2006.07.017] |
| [8] |
Butt D, Shaddick K, Raftos D, et al. The effect of low salinity on phenoloxidase activity in the Sydney rock oyster, Saccostrea glomerata[J]. Aquaculture, 2006, 251(2-4): 159-166. DOI:10.1016/j.aquaculture.2005.05.045 |
| [9] |
Navarro J M. The effects of salinity on the physiological ecology of Choromytilus chorus (Molina, 1782) (Bivalvia: Mytilidae)[J]. Journal of Experimental Marine Biology and Ecology, 1988, 122(1): 19-33. DOI:10.1016/0022-0981(88)90209-2 |
| [10] |
Meng X L, Dong Y W, Dong S L, et al. Mortality of the sea cucumber, Apostichopus japonicus Selenka, exposed to acute salinity decrease and related physiological responses: Osmoregulation and heat shock protein expression[J]. Aquaculture, 2011, 316(1-4): 88-92. DOI:10.1016/j.aquaculture.2011.03.003 |
| [11] |
Li W J. Biology and cultivation of oyster Crassostrea nippona[J]. Fisheries Science, 2007, 26(12): 689-690. [李文姬. 岩牡蛎的生物学及其养殖[J]. 水产科学, 2007, 26(12): 689-690. DOI:10.3969/j.issn.1003-1111.2007.12.013] |
| [12] |
Yang C, Su X R, Li T W. The cultural technique of Pacific oyster, Crassostrea gigas[J]. Fisheries Science, 2003, 22(5): 31-33. [杨春, 苏秀榕, 李太武. 牡蛎养殖技术[J]. 水产科学, 2003, 22(5): 31-33.] |
| [13] |
Berger V J, Kharazova A D. Mechanisms of salinity adaptations in marine molluscs[J]. Hydrobiologia, 1997, 355(1-3): 115-126. |
| [14] |
Zhao X L, Yu H, Kong L F, et al. Transcriptomic responses to salinity stress in the Pacific oyster Crassostrea gigas[J]. PLoS ONE, 2012, 7(9): e46244. DOI:10.1371/journal.pone.0046244 |
| [15] |
Meng J, Zhu Q H, Zhang L L, et al. Genome and transcriptome analyses provide insight into the euryhaline adaptation mechanism of Crassostrea gigas[J]. PLoS ONE, 2013, 8(3): e58563. DOI:10.1371/journal.pone.0058563 |
| [16] |
Dheda K, Huggett J F, Chang J S, et al. The implications of using an appropriate reference gene for real-time reverse transcription PCR data normalization[J]. Analytical Biochemistry, 2005, 344(1): 141-143. DOI:10.1016/j.ab.2005.05.022 |
| [17] |
Du Y S, Zhang L, Xu F, et al. Validation of housekeeping genes as internal controls for studying gene expression during Pacific oyster (Crassostrea gigas) development by quantitative real-time PCR[J]. Fish & Shellfish Immunology, 2013, 34(3): 939-945. |
| [18] |
Cubero-Leon E, Ciocan C M, Minier C, et al. Reference gene selection for qPCR in mussel, Mytilus edulis, during gametogenesis and exogenous estrogen exposure[J]. Environmental Science and Pollution Research, 2012, 19(7): 2728-2733. DOI:10.1007/s11356-012-0772-9 |
| [19] |
Sirakov M, Zarrella I, Borra M, et al. Selection and validation of a set of reliable reference genes for quantitative RT-PCR studies in the brain of the Cephalopod Mollusc Octopus vulgaris[J]. BMC Molecular Biology, 2009, 10: 70. DOI:10.1186/1471-2199-10-70 |
| [20] |
Wang H L, Wen H S, Li Y, et al. Evaluation of potential reference genes for quantitative RT-PCR analysis in spotted sea bass (Lateolabrax maculatus) under normal and salinity stress conditions[J]. PeerJ, 2018, 6: e5631. DOI:10.7717/peerj.5631 |
| [21] |
Zhang Y F, Zhao L J, Zeng Y L. Selection of reference genes in transcription analysis of gene expression[J]. Plant Physiology Journal, 2014, 50(8): 1119-1125. [张玉芳, 赵丽娟, 曾幼玲. 基因表达研究中内参基因的选择与应用[J]. 植物生理学报, 2014, 50(8): 1119-1125.] |
| [22] |
Bustin S A. Quantification of mRNA using real-time reverse transcription PCR (RT-PCR): Trends and problems[J]. Journal of Molecular Endocrinology, 2002, 29(1): 23-39. |
| [23] |
Dheilly N M, Lelong C, Huvet A, et al. Development of a Pacific oyster (Crassostrea gigas) 31, 918-feature microarray: identification of reference genes and tissue-enriched expression patterns[J]. BMC Genomics, 2011, 12: 468. DOI:10.1186/1471-2164-12-468 |
| [24] |
Morga B, Arzul I, Faury N, et al. Identification of genes from flat oyster Ostrea edulis as suitable housekeeping genes for quantitative real time PCR[J]. Fish & Shellfish Immunology, 2010, 29(6): 937-945. |
| [25] |
Guo R, Lee M A, Ki J S. Normalization genes for mRNA expression in the marine diatom Ditylum brightwellii following exposure to thermal and toxic chemical stresses[J]. Journal of Applied Phycology, 2010, 25(4): 1101-1109. |
| [26] |
Fernandes J M O, Mommens M, Hagen O, et al. Selection of suitable reference genes for real-time PCR studies of Atlantic halibut development[J]. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2008, 150(1): 23-32. DOI:10.1016/j.cbpb.2008.01.003 |
| [27] |
Fernández-Aparicio M, Huang K, Wafula E K, et al. Application of qRT-PCR and RNA-Seq analysis for the identification of housekeeping genes useful for normalization of gene expression values during Striga hermonthica development[J]. Molecular Biology Reports, 2013, 40(4): 3395-3407. DOI:10.1007/s11033-012-2417-y |
| [28] |
Temel A, Gozukirmizi N. Physiological and molecular changes in barley and wheat under salinity[J]. Applied Biochemistry and Biotechnology, 2015, 17(6): 2950-2960. |
| [29] |
Hiller G, Weber K. Radioimmunoassay for tubulin: a quantitative comparison of the tubulin content of different established tissue culture cells and tissues[J]. Cell, 1978, 14(4): 795-804. DOI:10.1016/0092-8674(78)90335-5 |
| [30] |
Browne G J, Proud C G. Regulation of peptide-chain elongation in mammalian cells[J]. European Journal of Biochemistry, 2002, 269: 5360-5368. DOI:10.1046/j.1432-1033.2002.03290.x |
| [31] |
Siomi H, Dreyfuss G. RNA-binding proteins as regulators of gene expression[J]. Current Opinion in Genetics and Development, 1997, 7(3): 345-353. DOI:10.1016/S0959-437X(97)80148-7 |
| [32] |
Xiong X H, Feng Q L, Xie L P, et al. Cloning and characterization of a heterogeneous nuclear ribonucleoprotein homolog from pearl oyster, Pinctada fucata[J]. Acta Biochimica et Biophysica Sinica, 2007, 39: 955-963. DOI:10.1111/j.1745-7270.2007.00341.x |
 2019, Vol. 26
2019, Vol. 26

