2. 中国水产科学研究院黄海水产研究所, 山东 青岛 266071
2. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071, China
DNA条形码是生物体内一段标准的、有足够变异的、易扩增且相对较短的DNA片段。由于该DNA片段与物种存在唯一对应关系, 因而该片段可以像商品的条形码一样标识对应的物种信息, 故称为DNA条形码。按照国际生命条形码协会建议, 动物DNA条形码通常选择COI (CO1或者COX1)基因为候选基因, 植物一般采取matK、rbcL、trnH-psbA、ITS基因为候选基因[1]。研究人员通过分子检测技术, 快速获得未知物种的DNA条形码信息, 比较已知的DNA条形码-物种对应关系, 能准确鉴定未知物种的信息。与传统的形态学分类相比, 条形码技术具有不受样品性别、发育阶段、形态学变化限制的优点, 因此在生命科学、法医学、药学、食品学和检验检疫等领域均有广泛的应用。
在获得DNA条形码与物种分类信息的对应关系后, 如何有效管理DNA条形码、物种分类信息及其二者之间的对应关系信息, 并能方便相关研究人员使用这些数据信息, 即数据管理和信息共享问题, 是限制DNA条形码应用的主要问题。目前, 构建DNA条形码数据信息系统是最有效的解决方案。结合数据库技术和网络技术构建DNA条形码信息平台, 一方面能规范存储和查询存储样品信息和DNA条形码序列, 另一方面能对外提供物种鉴定分析的生物信息学工具, 对推动DNA条形码研究和应用具有重要意义[1]。第一个国际DNA条形码数据系统-生命条形码数据库系统(Barcode of Life Database Systems, BOLD systems), 由国际生命条形码联盟生命条形码协会(Consortium for the Barcode of Life, CBOL)于2007年建立[2]。迄今, BOLD系统内包含超过18万种动物、6.6万种植物和2.1万种真菌等物种的566.5万条DNA条形码序列[3]。CBOL还针对特定生物类群设立了单独的条形码数据库, 其中就有针对全球鱼类的条形码数据库Fish-BOL(Fish Barcode of Life, http://fishbol.org/)。2011年, 应加入国际生命条形码计划(international Barcode of Life, iBOL)的需求, 我国也建立起中国生命条形码数据门户(http://www.barcodeoflife.cn/)。该数据库包含64176个标本的77957条序列[4]。另外, 国内各行业有针对性的逐步建立了一系列条形码数据库, 例如中药材DNA条形码鉴定系统(http://www.tcmbarcode.cn/), 中国珍稀濒危植物DNA条形码鉴定平台(http://www.brep.ac.cn/)以及中国检疫性有害生物DNA条形码鉴定系统(http://www.qbol.org.cn/)等。
我国渔业生物资源丰富, 海洋生物种类高达20278种, 其中鱼类3032种[5]。仅在渔业统计和市场上列名的渔业对象也有200多种, 意味着我国海洋水产生物拥有大量潜在的生物信息数据。尽管Fish BOLD系统和BOLD系统收录全球鱼类和其他海洋生物的DNA条形码数据, 但这两个数据库主要采集欧、美和澳等西方国家的渔业物种信息。鉴于针对中国渔业生物的DNA条形码信息收录较少的现状, 科技部分别于2013年启动科技基础性工作专项“我国重要渔业生物DNA条形码信息采集及其数据库构建”重点项目, 于2014年启动“我国近海海洋生物DNA条形码资源库构建”基础性工作专项。此外, 国内其他科研机构也采集了一些特殊生境下渔业生物或者特定物种的DNA条形码信息。譬如, 沈彦君等[6]采集获得华中地区和长江中下游区域近100种重要渔业生物的DNA条形码序列。唐伟等[7]采集中华鳖6个地理群体的111个个体DNA条形码序列。在获得DNA条形码序列后, 有必要建立相关的信息平台, 有效地管理数据和共享信息, 以解决限制DNA条形码应用的主要问题。
综上所述, 本项目在采集我国重要渔业生物的凭证标本及相应DNA条形码的基础上, 拟通过建立中国渔业生物DNA条形码信息平台, 在网络环境下实现对我国渔业生物DNA条形码的物种鉴定、信息浏览及查询功能, 为我国渔业生物DNA条形码数据管理及快速鉴定提供服务平台, 为物种鉴定、资源保护和质量安全科研人员提供技术支持。
1 材料与方法 1.1 实验材料本项目选取COI、ITS和rbcL序列为DNA条形码位点, 其中鱼类和甲壳类选择COI, 贝类选择COI和ITS, 藻类主要选择ITS和rbcL为检测位点。
本平台的DNA条形码数据有两部分来源, 一部分来源是项目组采集的渔业生物凭证标本和对应的DNA条形码序列。该部分数据经过实验验证, 有可追溯的凭证标本, 因此数据质量高, 归为标准数据集, 即“标准库”。为保证条形码序列和物种信息可靠性, 对于标准库的序列信息, 利用BLAST比对到NCBI NR数据库, 检测提交的DNA条形码序列是否污染序列以及同源序列是否为上述三个基因。不同于常用的核酸数据库, 除了DNA条形码信息外, 本信息平台还要求提交对应的物种信息、凭证标本号、标本信息、采集信息等信息。这是保证DNA条形码序列质量的可追溯信息, 也是了解对应物种特征的重要信息。其中, 凭证标本是DNA条形码序列信息的实物载体, 是溯源的唯一实物标识。
另一部分来源于GenBank数据库, 这部分数据未经过实验验证, 归为参考数据集, 即“参考库”。选择基因名称为“COI”、“CO1”、“COX1”、“rbcL”和“ITS”, 且物种门类为“Actinopteri”、“Bivalvia”、“Malacostraca”和“Rhodophyta”的基因记录。利用BioPerl进行解析, 保留物种分类信息、物种名称、条形码序列信息。由于GenBank中序列存在冗余, 对于碱基信息一致、物种一致的序列, 仅保留最长序列作为唯一记录。
1.2 数据库设计及实现本信息平台由物种分类数据库、凭证标本数据库和DNA条形码数据库三个子库组成。数据标准的不统一会在项目组内部和对外共享时造成壁垒, 因此本研究对三个子库的数据建立统一的规范格式, 利用SQL server存储和检索三个子库的数据。
物种分类数据库是利用NCBI Taxonomy数据库的“门-纲-目-科-属-种”关系构建的, 每个物种有且仅有一个明确的且唯一的分类关系。该数据库中的物种分为鱼类、甲壳类、贝类、藻类以及其他渔业生物五大门类。除物种的分类信息外, 数据库还包括拉丁名、英文名、中文名、同种异名、形态特征、地理分布和参考文献等。为丰富物种的形态特征和地理分布, 本平台还借鉴FishBase (http://fishbase.org/)和国家水产种质资源平台(http://zzzy.fishinfo.cn/)中渔业生物的物种信息。在物种分类数据库中, 每个物种仅有唯一的记录。在上述信息中, 物种的拉丁名和分类地位是必需信息。每条记录以物种拉丁名为主键, 以作为该物种的唯一标识。
凭证标本数据库是各项目组所采集样品的详细信息, 包含凭证标本对应物种的拉丁名、凭证编码、馆藏码、标本保存位置、鉴定者、鉴定日期、采集区域、采集方式、底质、采集日期、水深、样品照片、特征描述、提交者以及录入时间等。在凭证标本数据库中, 每个凭证标本也是唯一的。在上述信息中, 凭证编码、物种的拉丁名和馆藏码是必需信息。其中以凭证编码为主键, 作为该凭证信息的唯一标识; 以物种拉丁名为外键, 与物种分类数据库的物种拉丁名关联; 馆藏码是该标本对应在实体库中的凭证编码。对于每个凭证标本, 仅有唯一对应的物种信息。
DNA条形码数据库是各项目组采集的凭证标本所对应的DNA条形码, 包含物种拉丁名、英文名、中文名、DNA编码、序列长度、录入时间、基因位点、序列提交者、基因类型(COI、ITS或者rbcL序列)、提交机构、Fasta格式序列、PCR扩增引物、实验人员、信息审核者、取样部位、样品提供者、样品来源和采集依据等。在DNA条形码数据库中, 每条DNA条形码也是非冗余的。在上述信息中, 物种的拉丁名、DNA编码和基因类型是必需信息。其中DNA编码为主键, 作为该条形码的唯一标识; 以物种拉丁名为外键, 与物种分类数据库的拉丁名关联。每个DNA条形码仅有唯一对应的物种信息。
1.3 共享平台的模块设计门户网站是国际上常用的数据共享方式。本信息平台包括以下三个模块:
数据门户模块:在网页门户提供项目进展、数据统计和信息。允许用户以某些关键词查询标准数据集中物种分类、凭证标本和DNA序列三个数据库的信息。也支持通过上述五种大门类浏览各个物种名录, 选择感兴趣的物种, 查询对应的信息。
物种鉴定模块:基于本地构建的BLASTN程序, 将需要鉴定的序列与DNA条形码数据库进行同源比对, 根据最佳比对结果进行物种鉴定。
管理模块:面向项目成员的可视化数据管理, 包括数据查询、导入、增删改、用户角色管理、数据备份和日志维护等功能。
1.4 平台框架的实现上述三个模块布置在Windows Server操作系统上。为实现数据门户和物种鉴定的两个模块, 我们采用“浏览器+服务器端”的架构。用户通过浏览器访问服务器, 检索查询信息和鉴定物种。服务器端以表现层、数据访问层和数据库的设计模式进行开发(图 1)。这两个模块的表现层包括信息检索、物种鉴定和数据统计等功能。本研究组利用ASP.NET技术开发网站实现表现层功能。在数据访问层实现方面, 数据门户模块通过SQLdb连接服务器端的SQL Server数据库, 允许以物种拉丁名、标本编码和DNA条形码编码为关键词查询标准数据集信息; 物种鉴定模块调动服务器端的BLAST程序, 将用户提交的未知生物DNA条形码序列与DNA条形码数据库进行比对, 查询最近缘物种。在数据库方面, 利用SQL server实现数据库标准管理。
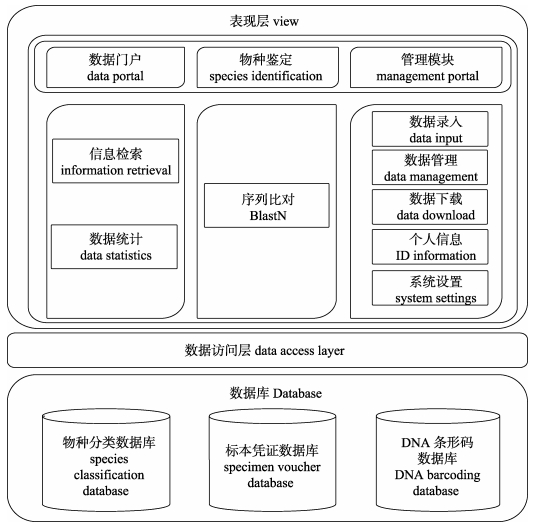
|
图 1 信息平台框架 Fig.1 System framework |
管理模块面向项目成员用于数据递交、管理和分析。为实现管理模块, 本研究采用“客户端+服务器端”的架构。用户通过客户端访问服务器端。客户端不仅具有上述两个模块的表现层所有功能, 而且还具有上传、编辑和下载数据的作用。本研究组利用C#语言编程, 使用Microsoft Visual Studio工具开发客户端。服务器端以数据访问层和数据库的设计模式进行开发(图 1)。对于服务器端的数据访问层, 一方面通过SQLdb连接服务器端的SQL Server数据库, 另一方面通过模块调动服务端的BLAST程序以进行物种鉴定。在服务器端的数据库方面, 同样利用SQL server实现数据库标准管理。
2 结果与讨论 2.1 数据统一规范管理中国渔业生物DNA条形码信息平台包括标准库和参考库。其中, 标准库包含物种分类数据库、凭证标本数据库和DNA条形码数据库三个子库。三个子库是以物种信息关联在一起。物种与凭证之间为“一对多”映射关系。由于采集区域、时间、取样部位和标本形态等差异, 一个物种可以对应多个凭证标本。凭证标本与条形码之间为“多对一”映射关系。由于同一物种不同个体之间遗传信息的保守性, 多个凭证标本可对应到相同的条形码序列。最终, 通过建立“物种-凭证-DNA条形码”三者的对应关系, 实现利用DNA条形码信息体现物种信息的目的。目前标准库已收录6020个物种的11262条DNA条形码序列(表 1)。
|
|
表 1 标准库已收录的条形码序列数 Tab.1 Data statistics in standard DNA barcoding dataset |
而参考库仅包含物种分类数据库和DNA条形码数据库两个子库。参考库的物种分类数据库的数据规范同标准数据集的物种分类数据库一致, 也按照鱼类、甲壳类、贝类、藻类以及其他渔业生物分成5个大类。参考库和标准库以物种信息关联在一起。目前参考数据库中收录了61159条DNA条形码序列, 包括54960条COI序列、5740条ITS序列和459条rbcL序列。虽然标准数据库的数据质量高, 但是由于样品采集困难和实验周期长等因素影响, 数据量较少。利用参考数据库信息能极大扩充渔业生物DNA条形码信息平台信息, 有效弥补标准数据库数据量不足的缺陷。
2.2 数据查询和浏览本平台有两种访问模式。第一种模式是网页访问模式。广大有DNA条形码使用需求的用户可以访问www.fishery-barcode.cn, 该网站对应平台的数据门户模块和物种鉴定模块, 提供所有信息查询和检索功能。为查询物种分类、凭证标本和DNA条形码序列信息, 可以分别在“物种名录”、“凭证标本”和“DNA条形码”三个页面下, 以感兴趣物种的拉丁名进行检索, 网页返回详细的信息, 并且提供三种信息之间的关联链接(图 2)。用户在三个页面之间通过链接切换。
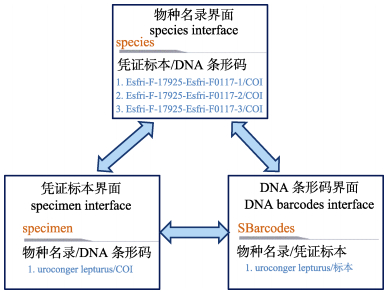
|
图 2 “物种名录”、“凭证标本”和“DNA条形码”三种信息链接关联 Fig.2 The linkage of interfaces among the "Species", "Specimen" and "DNA barcodes" |
物种鉴定功能是DNA条形码信息平台的最终目标。有物种鉴定需求的用户, 通过网页模式访问三种DNA条形码“在线BlastN”页面。在确定对应的DNA条形码类型后, 选择标准库或者参考库进行比对。提交未知物种Fasta格式的序列信息(序列长度介于20个碱基和1200个碱基)到物种鉴定模块的查询框, 然后点击“BlastN匹配”进行测试比对(图 3)。程序将返回同源序列, 并给出数据库中每条同源序列对应的物种分类信息。比对结果按照同源性从高到低排序。同源性越高的结果, 表明未知物种的DNA条形码与该物种的DNA条形码越相似, 提示未知物种与该物种越相似。为方便了解, 同源物种的各类信息, 比对结果还提供物种详细信息的超链接。系统默认给出同源性最高的物种作为推测的可能物种。
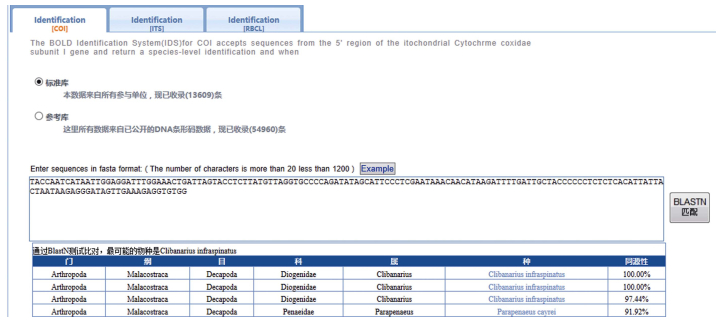
|
图 3 物种鉴定在线BlastN界面 Fig.3 The interface of species identification by BlastN |
为验证本信息平台的可用性和可靠性, 以7种常见鲟的COI序列作为实际案例, 利用本平台的物种鉴定模块进行识别对应的物种。结果显示鉴定的物种准确且可靠(表 2), 本平台在水产品的物种分类和来源鉴定等方面应该能够发挥重要作用。
|
|
表 2 7种常见鲟的COI序列比对结果 Tab.2 The alignment results of COI sequences from seven common sturgeons |
客户端访问是第二种访问模式, 面向项目成员实现数据上传、分析和管理功能, 对应到平台的管理模块。项目成员可以随时通过客户端上传和更新本平台的物种信息和条形码信息。为保障数据安全, 本平台的数据每3个月备份一次。相比网页访问模式, 除了物种信息管理、凭证标本管理、条形码管理和物种鉴定外, 客户端访问模式增加上传和编辑数据的功能。可上传的信息具体包括物种分类信息、物种信息、标本信息和条形码, 用户还可以通过客户端编辑和修订每条信息, 以保证数据的准确性(图 4)。
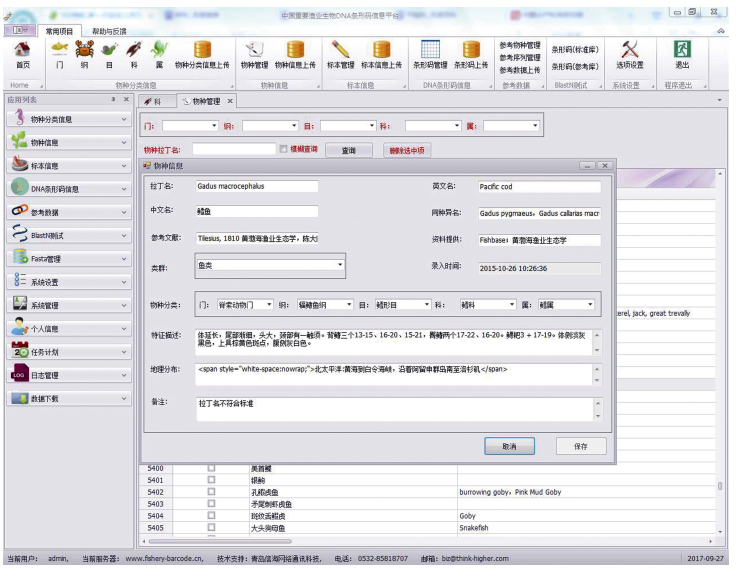
|
图 4 DNA条形码信息平台客户端界面 Fig.4 The client interface of the DNA barcoding system |
建立渔业生物DNA条形码信息平台, 具有重要的科研意义和广泛的应用前景。首先, 能弥补我国渔业生物DNA条形码数据的缺失, 新鉴定的渔业生物信息及条形码数据将源源不断地加入数据库, 促进我国渔业生物分类学科的发展。其次, 有助于实现渔业生物种质鉴定的标准化。比较DNA条形码序列间差异, 确定近缘物种, 为渔业生物的准确鉴定提供了依据。谢小雪等[8]发现可以利用ITS基因对坛紫菜进行准确的品系间种质鉴定, 从而避免种质混淆实现良种化栽培。再次, 建立渔业生物DNA条形码信息平台对鱼类资源生物多样性保护有重要意义。渔业生物遗传多样性是渔业资源评价的重要指标之一, 种内DNA条形码的多态性能够反映物种的遗传多样性。在生物多样性评估过程中, 以DNA条形码为参考, 加快完成物种的准确区别和鉴定, 能区分争议种, 发现濒危种、新种、隐存种和外来种, 重建种间和种内的演化关系, 揭示渔业生物多样性。最后, 建立渔业生物DNA条形码信息平台有助于加强水产品来源地和加工流通管理。将本信息平台应用于水产品流通环节, 可监管水产品加工中可能存在的以次充好、以假乱真等现象的发生。邱德义等[9]和王敏等[10]分别利用DNA条形码技术对市场上鱼肉等水产制品进行了鉴定, 发现广泛存在产品标签与实际不符和以次充好的情况[9-10]。
现阶段本信息平台存储COI、ITS和rbcL三个基因的序列信息, 物种鉴定方法只支持三个基因的BlastN匹配。实际上, 只依靠单一的条形码基因往往不能准确鉴定区分一些特定物种。Kruck等[11]发现只有同时利用这三个基因的序列才能准确鉴别
新型信息技术将推动本信息平台更广泛的应用。近年来, 微信等通信方式成为各类信息服务类平台与用户交流的新渠道, 目前仅中国政务类微信公众号已逾10万[16]。各类博物馆、图书馆和学术数据库等非营利性信息服务平台也通过微信公众号这一创新、灵活和高效的传播方式, 从而提高自身的公众服务能力[17-19]。通过微信后台与平台网站接入, 用户在关注公众号后, 无需任何预装软件直接通过移动端进行信息检索。公众号的良好运营, 对平台宣传、提高网站流量、完成数据共享和信息服务, 也有巨大的推动作用, 这些技术优势是网站访问模式无法比拟的。目前其他国内DNA条形码数据库平台或系统仅支持网页检索和客户端接入, 未见支持微信公众号类访问模式。本平台下一步计划支持微信公众号的访问模式, 及时向用户推送平台研究进展, 同时将项目发现的新物种、土著种、珍稀种介绍给公众, 促进本平台在渔业生物鉴定、资源可持续利用和濒危物种保护中的广泛应用。
DNA条形码技术在进出口检验检疫、外来入侵物种监管和食品鉴定等方面正发挥越来越重要的作用, 本平台将持续为国内水产科学研究和渔业产业发展提供准确可靠的DNA条形码信息服务。
| [1] |
Chen L, Wu L, Wang Q F, et al. Application of DNA barcoding in biodiversity[J]. Sichuan Journal of Zoology, 2016, 35(6): 942-949. [陈炼, 吴琳, 王启菲, 等. DNA条形码及其在生物多样性研究中的应用[J]. 四川动物, 2016, 35(6): 942-949.] |
| [2] |
international Barcode of Life, iBOL[OL]. http://ibol.org/. [国际生命条形码计划[OL]. http://ibol.org/.]
|
| [3] |
Barcode of Life Database Systems, BOLD systems[DB/OL]. http://boldsystems.org/. [生命条形码数据库系统[DB/OL]. http://boldsystems.org/.]
|
| [4] |
China Barcode of Life[DB/OL]. http://www.barcodeoflife.cn/. [中国生命条形码数据门户[DB/OL]. http://www.barcodeoflife.cn/.]
|
| [5] |
Du B L. The role of marine resources development in sustainable development in China[J]. Ocean Development and Management, 1997, 14(3): 40-43. [杜碧兰. 海洋资源开发对我国可持续发展的作用[J]. 海洋开发与管理, 1997, 14(3): 40-43.] |
| [6] |
Shen Y J, Wang X Z, Gan X N. DNA barcoding promotes informatization of Chinese fishery in the food safety of important fishery species[J]. Management and Research on Scientific & Technological Achievements, 2016(7): 56-58. [沈彦君, 王绪祯, 甘小妮. DNA条形码技术促进我国水产领域信息化进程——重要水产动物DNA条形码及其在食品安全领域的应用[J]. 科技成果管理与研究, 2016(7): 56-58. DOI:10.3772/j.issn.1673-6516.2016.07.020] |
| [7] |
Tang W, Zhu Z R, Wang C S, et al. Identification of Trionyx sinensis Using DNA barcoding based on COI gene[J]. Jiangsu Agriculture Science, 2017, 45(6): 30-36. [唐伟, 朱治任, 汪财生, 等. 基于COI基因的DNA条形码在鳖科动物鉴定上的应用[J]. 江苏农业科学, 2017, 45(6): 30-36.] |
| [8] |
Xie X X, Chen C S, Xu Y, et al. Comparative analysis on applicability of DNA sequence markers for identification of Pyropia haitanensis gemplasm[J]. Journal of Applied Oceanography, 2013, 32(3): 404-410. [谢小雪, 陈昌生, 徐燕, 等. DNA序列标记在坛紫菜种质鉴定中适用性的比较分析[J]. 应用海洋学学报, 2013, 32(3): 404-410. DOI:10.3969/J.ISSN.2095-4972.2013.03.014] |
| [9] |
Qiu D Y, Hu J, Liu D X, et al. Application of DNA barcoding in anti-fraud identification of aquatic products[J]. Meat Research, 2013, 27(4): 40-43. [邱德义, 胡佳, 刘德星, 等. DNA条形码技术在肉品防欺诈鉴别中的应用[J]. 肉类研究, 2013, 27(4): 40-43.] |
| [10] |
Wang M, Liu H, Huang H, et al. Identifying fish products in Shenzhen through DNA barcoding[J]. Food Science, 2015, 36(20): 247-251. [王敏, 刘荭, 黄海, 等. DNA条形码技术在深圳鱼肉制品鉴定中的应用[J]. 食品科学, 2015, 36(20): 247-251. DOI:10.7506/spkx1002-6630-201520048] |
| [11] |
Krück N C, Tibbetts I R, Ward R D, et al. Multi-gene barcoding to discriminate sibling species within a morphologically difficult fish genus (Sillago)[J]. Fisheries Research, 2013, 143(1): 39-46. |
| [12] |
Chen W B, Miao T Y, Peng J, et al. Identification of Anguilla rostrata, Anguilla anguilla and Anguilla japonica using DNA barcoding based on 16S rRNA gene[J]. Food Science, 2017, 38(4): 283-289. [陈文炳, 缪婷玉, 彭娟, 等. 基于16S rRNA基因DNA条形码鉴定美洲鳗、欧洲鳗、日本鳗[J]. 食品科学, 2017, 38(4): 283-289.] |
| [13] |
Wu F F. DNA barcoding assisting identification of Rhodophyta in the intertidal zone of Shandong Peninsula[D]. Qingdao: Ocean University of China, 2013. [吴菲菲.山东半岛潮间带红藻分子条形码鉴定[D].青岛: 中国海洋大学, 2013.]
|
| [14] |
Chen X Z, Guo S L, Gong Y Q, et al. 16S rRNA and COI Gene Barcodes Applied for Identification of Ornamental Fishes[J]. Fujian Journal of Agricultural Sciences, 2016, 31(12): 1267-1272. [陈信忠, 郭书林, 龚艳清, 等. 16S rRNA和COI基因条形码在12种观赏鱼种类鉴定中的应用[J]. 福建农业学报, 2016, 31(12): 1267-1272.] |
| [15] |
Chesters D, Zheng W, Zhu C. A DNA barcoding system integrating multigene sequence data[J]. Methods in Ecology & Evolution, 2015, 6(8): 930-937. |
| [16] |
微信公众号, 百度百科[OL]. https://baike.baidu.com/item/微信公众号/4916400?fr=aladdin.
|
| [17] |
Lu J M. Analysis of the use of WeChat APP platform to enhance the museum's public cutural sevices[J]. Innovation Science and Technology, 2016(6): 76-78. [鹿继敏. 利用微信APP平台提升博物馆公共文化服务的探析[J]. 创新科技, 2016(6): 76-78. DOI:10.3969/j.issn.1671-0037.2016.06.021] |
| [18] |
Li W J, Chen S Q. WeChat as library public service platform for the APP client[J]. New Technology of Library and Information Service, 2014, 30(7/8): 133-138. [李文江, 陈诗琴. 微信作为APP客户端的图书馆公共服务平台[J]. 现代图书情报技术, 2014, 30(7/8): 133-138.] |
| [19] |
Chen W. Problems and srategies of WeChat public account usage in academic database[J]. Publishing Journal, 2016, 24(4): 83-87. [陈维. 学术数据库微信公众号运营现状调查与评析[J]. 出版科学, 2016, 24(4): 83-87. DOI:10.3969/j.issn.1009-5853.2016.04.021] |
 2018, Vol. 25
2018, Vol. 25

