2. 南京农业大学信息科学技术学院, 江苏 南京 210014;
3. 中国水产科学研究院淡水渔业研究中心, 农业农村部淡水渔业与种质资源利用重点实验室, 江苏 无锡 214081;
4. 山西农业大学动物科技学院, 山西 晋中 030801
2. Information Science and Technology College, Nanjing Agricultural University, Nanjing 210014, China;
3. Key Laboratory of Freshwater Fisheries and Germplasm Resources Utilization, Ministry of Agriculture and Rural Affairs; Freshwater Fisheries Research Center, Chinese Academy of Fishery Sciences, Wuxi 214081, China;
4. Animal Science and Veterinary Medicine College, Shanxi Agricultural University, Jinzhong 030801, China
鲤是世界上最早养殖的鱼类之一, 在中国有着悠久的历史。至今在淡水养殖鱼类中, 鲤依然占据举足轻重的地位。2017年淡水养殖产量共为3179.26万t, 其中鲤位居第三, 占淡水水产养殖产量的11%[1]。在众多养殖的鲤品种中, 黄河鲤具有丰富的文化底蕴, 在沿黄河地区具有较大的养殖面积。项目组在河南省水产科学研究院培育的豫选黄河鲤群体第9代的基础上, 通过最佳线性无偏预测(best linear unbiased prediction, BLUP)、家系选育、分子辅助育种等手段对其开展相关选育工作, 经过3个世代的选择育种, 项目组成功获得了体重提高20.5%, 不饱和脂肪酸提高了9.5%的黄河鲤新品系[2]。为了明确选育系生长优势的机制, 项目组应用比较基因组的方法, 以黄河鲤新品系选育系和对照系共240尾鱼为研究对象, 通过genome-wide association study (GWAS)分析, 筛选出与鲤生长相关的功能基因, 如IGF2、bone morphogenetic protein (BMP4), Neurexin-1a- like (NRXN1a)等。
IGF2具有促进细胞分裂的功能, 其结构与胰岛素类似[3], 在胰岛素的靶细胞中发挥类胰岛素的功能。此外, 也是生长激素发挥作用的中间信使, 传递生长激素, 对机体的生长和发育起作用[4-7]。1986年, Andersson等[8]提出IGF2可以调节猪体内生长激素与代谢水平, 刺激肌纤维的增殖和分化, 从而影响生长发育。研究表明正常范围内IGF2水平与许多大型哺乳动物从胎儿期到青年期的生长率成正相关[9-11]。Chen等[12]使用外源性的重组IGF2载体腹腔注射罗非鱼5周后, 与对照组相比体重和体长分别增加了72%和34%, 验证了其表达量的增加对罗非鱼生长具有促进作用。IGF2在哺乳动物中只存在单个基因, 在鲤中存在IGF2a和IGF2b两个同源基因, 且鲤IGF2b与哺乳动物中的IGF2基因同源性更高, 但IGF2b基因是否影响黄河鲤新品系的生长尚不清楚。
在多态位点层面, 以往的研究多从进食状态、营养物质的转运等方面研究IGF2基因的功能。Bachner-Melman等[13]在以家庭为单位的饮食失调症调查中发现, IGF2 ApaI A/G位点与进食态度测试分数、身体质量指数显著关联。Petry等[14]通过对1160个家庭(每个家系包括父母本和子代)的研究发现, 父源IGF2基因两个多态位点与妊娠晚期母体的葡萄糖浓度和出生时胎盘IGF2浓度呈正显著相关。Van Laere等[15]的研究证明, 位于IGF2内含子3上的G/A多态使猪的瘦肉率增加。后来该位点扩展到了外三元的研究分析中, 通过研究大白、长白和杜洛克猪中出生重和早期生长速度的关系, 证实了该位点对早期生长速度、饲料转化率等的关联性[16-17], 即影响肉质的IGF2基因SNP位点与该基因的表达有关。那么本文拟从重测序的角度获得与黄河鲤新品系IGF2相关的SNPs位点, 并通过分段检测该基因在黄河鲤新品系育种群体中的SNPs和对应基因表达量, 来研究其多态位点和基因表达量的关系。
从上述描述可以看出, IGF2基因可通过其表达量、多态性及它们的相互联系来影响动物机体的生长性能。而在鲤和斑马鱼上均发现了IGF2基因存在两个基因[18-19], 因此, 本研究以与哺乳动物相似度更高的IGF2b基因为研究对象, 从基因组DNA水平上分段获取IGF2b基因的SNPs信息, 并以不同的鲤品种验证其适用性, 同时在黄河鲤新品系中检测其应用, 为分子辅助育种、基因组育种提供研究基础。
1 材料与方法 1.1 实验试剂RNA提取试剂RNAiso Plus、cDNA反转录试剂盒、DL2000 Marker、TaKaRa全血基因组试剂盒均购自Takara宝生物工程(大连)有限公司; 琼脂糖等购自上海申能博彩生物科技公司; DYY-7C型电泳仪购自北京市六一仪器厂; TaKaRa梯度PCR扩增仪由日本TaKaRa公司生产; UVP GelDoc-ItTM Imaging System由美国UVP凝胶成像系统公司生产, 4℃冷冻离心机、实时定量荧光PCR仪由美国Bio-Rad生产。
1.2 实验动物从中国水产科学研究院淡水渔业研究中心南泉试验基地随机选取黄河鲤、建鲤以及二者的正反交后代各5尾, 体重约200 g (用于引物条件摸索和测序验证)、黄河鲤新品系同一世代二龄的选育群体中随机选取60尾(用于分子标记和基因表达量检测)。实验鱼体况良好, 称量体重并记录。宰杀取肌肉组织。部分组织在‒20℃保存, 用于提取DNA, 部分组织用液氮处理后于‒80℃冻存, 用于总RNA的提取。
1.3 数据来源从NCBI数据库(https://www.ncbi.nlm.nih.gov/)下载33条10个不同品种鲤的重测序序列(具体序列号参考Xu等[20]的文章)
1.4 重测序数据组装及IGF2b的所有有关SNPs的获取通过fastq-dump v2.2.2软件将上述下载的序列从SRA格式转换为FASTQ格式。再以上述组装的序列作为参考序列, 运用软件BWA进行匹配。给参考基因组构建索引, 生成以参考基因组为基础的amb、ann、bwt、pac、sa格式的文件。寻找输入读长的SA坐标生成pair-end测序对应2个文件的sai文件。运用BWA生成sam格式的比对文件并对其进行重排序, 然后将排序后的sam文件转换为bam文件, 再对bam文件进行sort排序处理和加头处理。如果一个样本分为多个通道进行测序, 应先将各个通道的bam文件合并。使用picard tools去掉序列重复的部分, 去掉重复后的文件通过samtools形成对应的索引文件, 格式为bai文件。使用GATK中的RealignerTargetCreator和IndelRealigner对由indel导致错配的区域进行重新比对, 将indel附近的比对错误率降到最低。使用BaseRecalibrator和PrintReads将经过质量值校正的数据输出到新的bam文件中, 用于后续的变异检测。使用ReduceReads将bam文件进行压缩, 生成新的bam文件; 使用samtools形成VCF文件。获得原始VCF文件后, 通过以下标准进行分子标记的筛选:所有个体的单个位点覆盖度必须是10×以上; 读长大于10, 质量分数大于20, 最小基因频率大于0.01;所有的多态位点每个个体均应有对应的碱基, 不存在缺失情况。在获得筛选过的VCF文件后, 根据基因组的注释文件找到所有有关IGF2b基因的SNPs。
1.5 DNA提取和PCR扩增测序采用TaKaRa试剂盒提取黄河鲤、建鲤、黄河鲤和建鲤的正反交后代以及黄河鲤新品系肌肉组织总DNA, 凝胶电泳检测所提取DNA的质量, 并用分光光度计检测DNA浓度。其中黄河鲤、建鲤、黄河鲤和建鲤的正反交后代DNA用于IGF2b基因的分段克隆, 黄河鲤新品系DNA用于已筛选SNPs的应用及与该基因表达量相关程度的检测。
根据基因库中鲤IGF2b基因的cDNA序列(AF402958)和基因组DNA序列(HM755899)设计引物(表 1)分段扩增该基因的DNA序列。引物由上海英骏生物技术有限公司合成。本文对IGF2b3#采用的是直接测序法, 测序的结果使用DNAMAN9确定SNPs, 然后对照测序峰图确定实验结果的准确性。
|
|
表 1 鲤IGF2b DNA扩增所需引物 Tab.1 Primers for IGF2b DNA amplified |
PCR反应体系为25 μL, 包括10×buffer 15 μL, Mg2+ (25 mmol/L) 1 μL, dNTPs (2 mmol/L) 1 μL, 上下游引物(10 mmol/L)各1 μL, 模板DNA 1 ng, Taq DNA聚合酶(Promega)1 U, ddH2O补足。扩增反应在TaKaRa公司生产的梯度PCR仪上完成。PCR反应程序设置为: 94℃预变性3 min, 94℃变性20 s, 56℃退火20 s, 72℃延伸30 s, 28个循环; 72℃延伸10 min。8%非变性聚丙烯酰胺凝胶电泳结合goldview显色进行扩增片段检测, 由UVP成像系统(UVP gel docTM Imaging System, UVP Inc)拍照, 上海英骏生物技术有限公司测序。
1.6 总RNA的提取及其反转录、qPCR (quantitative real-time PCR, qPCR)实验按照RNAiso Plus操作说明书对黄河鲤新品系肌肉组织进行总RNA的提取, 采用琼脂糖凝胶电泳检测RNA的提取情况, 用分光光度法检测RNA的浓度, RNA反转录成cDNA用于后续的PCR反应, 其余的RNA于-80℃保存。
用实时荧光定量PCR仪(MJ Research)检测测序结果中随机选取差异基因的表达水平, 使用的IGF2b基因以及内参hACTB引物见文献[21]。PCR反应体系20 μL, 包括RT产物1.0 μL, dNTP 200 μmol/L, 目的基因上、下游引物各10 pmol, Taq DNA聚合酶3 U, 荧光探针10 pmol, 10×PCR Buffer (含100 mmol/L Tris-HCl pH 8.3, 500 mmol/L KCl) 1 μL。扩增条件为94℃预变性3 min, 然后按94℃变性45 s, 58℃退火30 s, 72℃延伸30 s, 共40个循环。反应结束后由电脑自动分析出定量结果。
1.7 数据分析在NCBI数据库中采用Blast进行序列比对, 使用Python2自写脚本完成对VCF文件中数据的提取、图的制作。实验数据在Microsoft Excel中初步整理后, 使用SAS8.0分析。实时荧光定量PCR中基因表达量采用ΔΔCt法进行计算, 采用t检验进行统计, 显著水平设定为0.05。
2 结果与分析 2.1 基于鲤重测序数据的IGF2b基因SNPs的获取从10个鲤品种重测序数据和基因组注释发现IGF2b基因共有两个转录本, 且两个转录本所在的区域80%交叉重合。筛选重测序数据发现, 转录本Ⅰ和转录本Ⅱ分别有107个和72个该基因的SNPs, 其中转录本Ⅰ中外显子占20个, 内含子占87个, 转录本Ⅱ中外显子占18个, 内含子占54个, 相较于转录本Ⅱ, 转录本Ⅰ多了2个外显子。且在2个转录本中内含子区域大小都远高于外显子区, 这些SNPs在两个转录本上的分布情况见图 1。相比外显子区域, 内含子区含有较多的SNPs, 同时转录本Ⅰ的SNPs数量高于转录本Ⅱ。
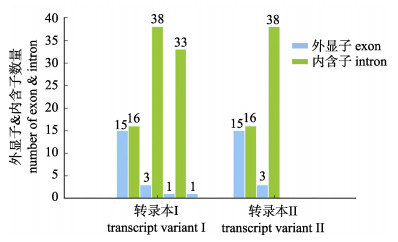
|
图 1 鲤全基因组IGF2b基因所有SNPs在两个转录本上的分布情况 Fig.1 Statistical graph of exon & intron of IGF2b in Cyprinus carpio whole-genome |
为了分析IGF2b基因的SNPs与鲤生产性能的关系, 利用8对PCR引物鲤IGF2b基因分别进行内含子区的分段克隆和测序获取相应的SNPs, 并对获得的分段克隆片段采用直接测序进行验证。然后分别对黄河鲤、建鲤、黄河鲤和建鲤的正反交后代进行PCR检测以验证其通用性, 扩增结果见图 2。根据片段的大小和序列特征, 选择直接测序和限制性片段长度多态性(PCR-restriction fragment length polymorphism, PCR-RFLP)结合进行。对于IGF2b基因克隆后SNP的检测, PCR检测发现条带单一, 目的片段大小正确。
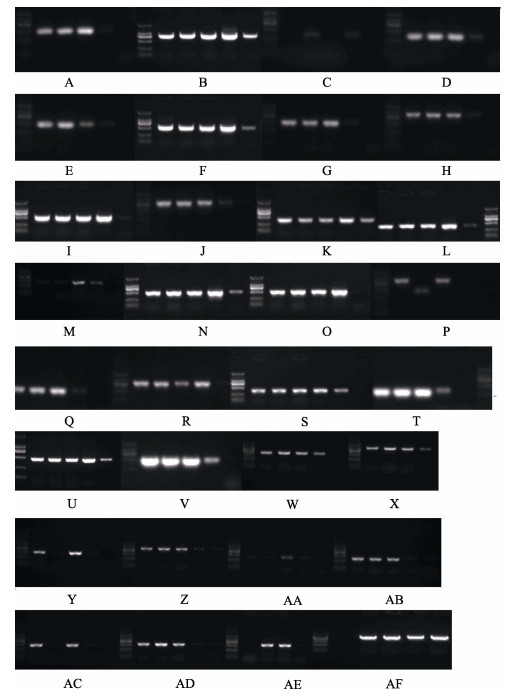
|
图 2 IGF2b产物的扩增结果 Marker: DL 2000 DNA分子标记; A~H:在黄河鲤中对IGF2b基因进行SNP分析的8对引物; I~P:在建鲤中对IGF2b基因进行SNP分析的8对引物; Q~X:在建鲤黄河鲤正交个体中对IGF2b基因进行SNP分析的8对引物; Y~AF:在建鲤黄河鲤反交个体中对IGF2b基因进行SNP分析的8对引物. Fig.2 Products of IGF2b SNP amplification Marker: DL 2000 DNA marker; A–H: 8 primer pairs were used to analysis of IGF2b SNP in Huanghe carp; I–P: 8 primer pairs were used to analysis of IGF2b SNP in Jian carp; Q–X: 8 primer pairs were used to analysis of IGF2b SNP in direct cross individuals of Jian carp and Huanghe carp; Y–AF: 8 primer pairs were used to analysis of IGF2b SNP in reciprocal cross individuals of Jian carp and Huanghe carp. |
利用IGF2b基因3#引物对黄河鲤新品系DNA进行扩增、测序, 发现227位点处存在A变C的基因多态性(图 3), 其中AA的基因型频率为0.75, AC的基因型频率为0.25, 并且两个基因型之间体重差异显著, 纯合型体重较大, 杂合子体重较小(图 4)。
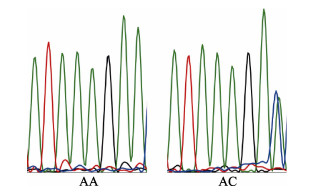
|
图 3 IGF2b3#引物227号位点出现的基因型 Fig.3 Genotypes of IGF2b 3# on 227 locus |
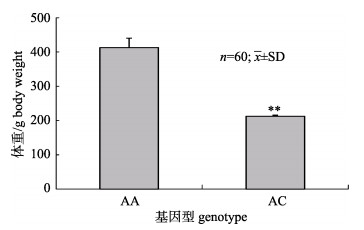
|
图 4 IGF2b3#引物227号位点出现的基因型对体重的影响 **表示P < 0.01. Fig.4 Effect on body weight of genotypes IGF2b3# on 227 locus **means P < 0.01. |
采用qPCR对IGF2b基因3#引物进行黄河鲤新品系基因型检测, 检测结果见图 5。根据测序图可看出, 该片段存在AA纯合子变为AT杂合子的现象, 经计算发现AA的基因型频率为0.33, AT的基因型频率为0.67。在分型的基础上, 对所研究的黄河鲤新品系的cDNA进行表达量的检测, 得到的扩增曲线和熔解曲线, 如图 6所示。从图中可以看出扩增曲线和熔解曲线都符合要求, 可用于表达量的检测。统计表明IGF2b3#不同基因型之间存在着表达量差异, 杂合子具有较大的基因表达量, 且差异显著, 见图 7。
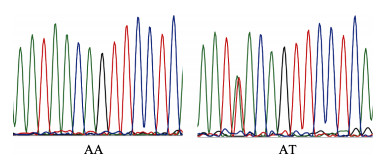
|
图 5 IGF2b基因表达量有差异的该基因基因型测序图 Fig.5 Sequence graph of IGF2b's different genotypes which had significant transcription expression level |
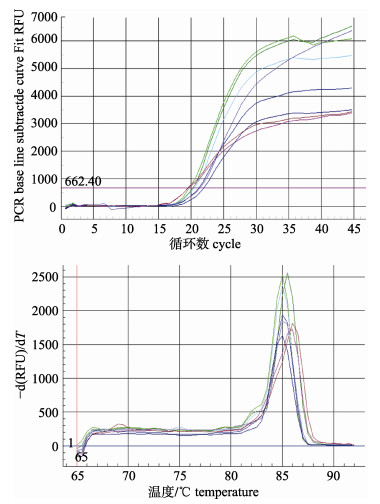
|
图 6 用于IGF2b基因表达检测的扩增曲线和熔解曲线 Fig.6 Amplification and melting curves of IGF2b realtime PCR |
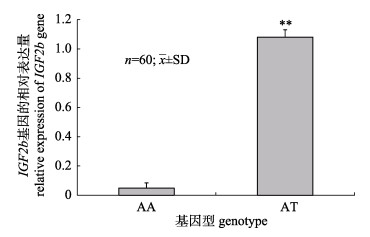
|
图 7 用于IGF2b基因3#引物不同基因型之间该基因表达量的差异 **表示P < 0.01. Fig.7 Expression difference in different genotypes for IGF2b 3# **means P < 0.01. |
本文通过基因库获取已有的重测序数据, 从整体上把握与目的性状相关的目标基因的所有SNPs及分布情况, 然后分段克隆获取目标SNPs。在思路上不同于传统的方法, 即通过基因克隆结合一代测序的路径查找潜在的SNPs, 然后通过酶切等手段获取多个个体的SNPs[11, 15, 22-23]来研究单个基因SNPs与选育性状的关系。另外本文从全基因组重测序角度, 从10个中外鲤品种共计33个个体获得IGF2b基因SNPs的分布情况, 同时分8个基因段, 对10个品种中的2个品种及其正反交后代做了可行性验证, 结果具有可靠性。这将为其他物种、其他功能基因的多态性分子标记研究提供思路。
3.2 IGF2b基因SNPs多态性、基因型表达量与黄河鲤新品系体重的关联研究结果显示黄河鲤新品系IGF2b3#引物227号位点处, 出现突变位点, 使得其体重降低。IGF2基因突变是否会通过表达量的变化影响生产性能的变化?查阅资料发现, 张宁等[24]通过构建鲤IGF2b载体并注射幼体鲤证实了该基因表达量与体重显著相关。因此本文基于此推断IGF2b3#引物227号位点与该基因表达量有关, 从而影响了黄河鲤新品系的体重。Younis等[25]通过转基因和基因敲除手段证实了IGF2基因突变通过扰乱该基因与转录因子ZBED6的结合而影响该基因的表达量, 进而影响家猪的瘦肉率。这与本文的结果类似。类似的, Msalya等[26]研究证明PRNP基因del/del基因型可调控该基因有23 bp长度RNA的表达。Helmig等[27]同样报道了TNFα (tumor necrosis factor-α)基因308野生型和杂合型个体血液白细胞中该基因表达有显著差异, Gossner等[28] Prnp (prion protein gene)基因型可影响Prnp基因的表达量。近年来, 更有研究报道从基因组水平上研究不同基因型之间的表达差异[29]。可以看出, 靶基因型和表达量均可能与所研究的目标性状有关。但是它们之间并不相关的情况也有报道。比如, Olsson等[30]研究发现SELS (selenoprotein S)基因基因型和表达量均与风险因子有关, 但是不同基因型和表达量的关系没有检测到。同样的报道显示, 与热耐受力有关的GSTP1 (glutathione S-transferase pi-1)基因表达量与基因型都与耐热性有关, 但是也没有发现二者的相关性[31]。这与本研究中IGF2b基因的SNP报道不一致, 这说明影响表型的SNP不一定通过表达量来影响生产性能。
通过对鲤的重测序数据及其IGF2b基因SNPs的分析, 并将其应用于黄河鲤新品系中进行验证, 发现黄河鲤新品系IGF2b3#引物227号位点处, 出现的突变位点使体重降低, 且有一处SNPs与其表达量有关。此方法已能够在该基因全基因组范围内开展鲤分子育种中SNPs的检测。
| [1] |
Fisheries Bureau of Ministry of Agriculture. China Fisheries Statistical Yearbook. 2017[M]. Beijing: China Agriculture Press, 2017.
|
| [2] |
Su S Y, Zhang C F, Dong Z J, et al. The breeding gain of Huanghe carp (Cyprinus carpio hacmalopterus Temminck et Schlegel) new strain G3—The effect of higher carbohydrate diet on growth and fatty acid profile of Huanghe carp new strain[J]. Journal of Yangzhou University (Agricultural and Life Science Edition), 2018, 39(2): 63-66. [苏胜彦, 张成锋, 董在杰, 等. 黄河鲤新品系G3代选育进展——饲喂高碳水化合物饲料对其生长和脂肪酸组分的影响[J]. 扬州大学学报(农业与生命科学版), 2018, 39(2): 63-66.] |
| [3] |
Rinderknecht E, Humbel R E. The amino acid sequence of human insulin-like growth factor I and its structural homology with proinsulin[J]. Journal of Biological Chemistry, 1978, 253(8): 2769-2776. |
| [4] |
Vasilatosyounken R, Scanes C G. Growth hormone and insulin-like growth factors in poultry growth: Required, optimal, or ineffective?[J]. Poultry Science, 1991, 70(8): 1764-1780. DOI:10.3382/ps.0701764 |
| [5] |
Kouros-Mehr H, Werb Z. Candidate regulators of mammary branching morphogenesis identified by genome-wide transcript analysis[J]. Developmental Dynamics, 2006, 235(12): 3404-3412. DOI:10.1002/dvdy.v235:12 |
| [6] |
Su S Y, Dong Z J, Qu J Q, et al. Molecular cloning and single, nucleotide polymorphism analysis of IGF2a genes in the common carp (Cyprinus carpio)[J]. Genetics and Molecular Research, 2012, 11(2): 1327-1340. DOI:10.4238/2012.May.15.3 |
| [7] |
Yuan Y, Hong Y. Medaka insulin-like growth factor-2 supports self-renewal of the embryonic stem cell line and blastomeres in vitro[J]. Scientific Reports, 2017, 7: Article No. 78. DOI:10.1038/s41598-017-00094-y |
| [8] |
Andersson L, Haley C S, Ellegren H, et al. Genetic mapping of quantitative trait loci for growth and fatness in pigs[J]. Science, 1994, 263(5154): 1771-1774. DOI:10.1126/science.8134840 |
| [9] |
Liu G L, Jiang S W, Xiong Y, et al. Association of PCR- RFLP polymorphisms of IGF2 gene with fat deposit related traits in pig resource family[J]. Acta Genetica Sinica, 2003, 30(12): 1107-1112. [刘桂兰, 蒋思文, 熊远, 等. IGF2基因PCR-RFLP多态性与脂肪沉积相关性状的关联分析[J]. 遗传学报, 2003, 30(12): 1107-1112.] |
| [10] |
Knoll A, Putnová L, Dvořák J, et al. A Ncii PCR-RFLP within intron 2 of the porcine insulin-like growth factor 2b (IGF2) gene[J]. Animal Genetics, 2000, 31(2): 150. DOI:10.1046/j.1365-2052.2000.00583.x |
| [11] |
Wang G Y, Yan B X, Deng X M, et al. Effects of IGF2 gene on growth and carcass traits of chickens and their imprinting status[J]. Science in China Ser. C: Life Sciences, 2004, 34(5): 429-435. [王根宇, 颜炳学, 邓学梅, 等. IGF2基因对鸡生长及屠体性状的影响及印记状况的研究[J]. 中国科学: C辑, 生命科学, 2004, 34(5): 429-435.] |
| [12] |
Chen J Y, Chen J C, Chang C Y, et al. Expression of recombinant tilapia insulin-like growth factor-I and stimulation of juvenile tilapia growth by injection of recombinant IGFs polypeptides[J]. Aquaculture, 2000, 181(3-4): 347-360. DOI:10.1016/S0044-8486(99)00239-2 |
| [13] |
Bachner-Melman R, Zohar A H, Nemanov L, et al. Association between the insulin-like growth factor 2 gene (IGF2) and scores on the Eating Attitudes Test in nonclinical subjects: A family-based study[J]. American Journal of Psychiatry, 2005, 162: 2256-2262. DOI:10.1176/appi.ajp.162.12.2256 |
| [14] |
Petry C J, Seear R V, Wingate D L, et al. Associations between paternally transmitted fetal IGF2 variants and maternal circulating glucose concentrations in pregnancy[J]. Diabetes, 2011, 60(11): 3090-3096. DOI:10.2337/db11-0689 |
| [15] |
Van Laere A S, Nguyen M, Braunschweig M, et al. A regulatory mutation in IGF2 causes a major QTL effect on muscle growth in the pig[J]. Nature, 2003, 425(6960): 832-836. DOI:10.1038/nature02064 |
| [16] |
Sun Y, Fei S Q, Tang H, et al. Polymorphism of G3072A in IGF2 gene and its association with birth weight and early growth traits in Yorkshire pigs[J]. Acta Veterinaria et Zootechnica Sinica, 2007, 38(12): 1306-1310. [孙亿, 费思清, 唐辉, 等. 猪IGF2基因G3072A位点多态性及其与大白猪初生重和早期生长的关系[J]. 畜牧兽医学报, 2007, 38(12): 1306-1310. DOI:10.3321/j.issn:0366-6964.2007.12.006] |
| [17] |
Fassa V B, Carden T R, Marini S J, et al. Analysis of the effects of five genes (IGF2, CTSD, TBC1D1, MC4R and FABP3) on feed conversion, growth rate and the content of subcutaneous fat in pigs of the Landrace breed[J]. RIA, Revista de Investigaciones Agropecuarias, 2015, 41(3): 282-288. |
| [18] |
Zou S M, Kamei H, Modi Z, et al. Zebrafish IGF genes: gene duplication, conservation and divergence, and novel roles in midline and notochord development[J]. PLoS ONE, 2009, 4(9): e7026. DOI:10.1371/journal.pone.0007026 |
| [19] |
Su S Y, Dong Z J, Qu J Q, et al. Molecular cloning and single nucleotide polymorphism analysis of IGF2a genes in the common carp (Cyprinus carpio)[J]. Genetics and Molecular Research, 2012, 11(2): 1327-1340. DOI:10.4238/2012.May.15.3 |
| [20] |
Xu P, Zhang X F, Wang X M, et al. Genome sequence and genetic diversity of the common carp, Cyprinus carpio[J]. Nature Genetics, 2014, 46: 1212-1219. DOI:10.1038/ng.3098 |
| [21] |
Su S Y, Dong Z J, Yuan X H, et al. Clone of IGF2b introns, analysis of its genomic sequence and. construction of lentiviral vector (Lenti-IGF2-IRES-EGF) in common carp (Cyprinus carpio L.)[J]. Acta Scientiarum Naturalium Universitatis Sunyatseni, 2012, 51(2): 77-85. [苏胜彦, 董在杰, 袁新华, 等. 鲤IGF2b基因内含子的克隆, 基因组序列分析及慢病毒载体构建[J]. 中山大学学报(自然科学版), 2012, 51(2): 77-85.] |
| [22] |
Li Z H, Wang Q G, Zhao J G, et al. Study on correlation analysis of single nucleotide polymorphism of IGF2 gene and body fatness traits in chicken[J]. Scientia Agricultura Sinica, 2004, 37(4): 600-604. [李志辉, 王启贵, 赵建国, 等. 类胰岛素生长因子Ⅱ (IGF2)基因多态性与鸡体脂性状的相关研究[J]. 中国农业科学, 2004, 37(4): 600-604. DOI:10.3321/j.issn:0578-1752.2004.04.024] |
| [23] |
Yu D B, He Z L, Zhang W F, et al. The genetic effects of IGF2 gene intron3 variance in pigs[J]. Hereditas, 2008, 30(1): 87-93. [虞德兵, 何宗亮, 张伟峰, 等. 猪IGF2基因内含子3变异的遗传效应分析[J]. 遗传, 2008, 30(1): 87-93.] |
| [24] |
Zhang N, Su S Y, Yuan X H, et al. The effect of overexpression of IGF2b gene mediated by lentiviral vector in dorsal muscle of common carp on growth of common carp: Cyprinus carpio L.)[J]. Journal of Yangzhou University (Agricultural and Life Science Edition), 2015, 36(3): 31-35. [张宁, 苏胜彦, 袁新华, 等. 慢病毒载体介导的IGF2b基因在鲤鱼背部肌肉组织中过表达对鲤鱼生长的影响[J]. 扬州大学学报:农业与生命科学版, 2015, 36(3): 31-35.] |
| [25] |
Younis S, Schönke M, Massart J, et al. The ZBED6–IGF2 axis has a major effect on growth of skeletal muscle and internal organs in placental mammals[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(9): E2048-E2057. DOI:10.1073/pnas.1719278115 |
| [26] |
Msalya G, Shimogiri T, Ohno S, et al. Evaluation of PRNP expression based on genotypes and alleles of two indel loci in the medulla oblongata of Japanese Black and Japanese Brown cattle[J]. PLoS ONE, 2011, 6(5): e18787. DOI:10.1371/journal.pone.0018787 |
| [27] |
Helmig S, Aliahmadi N, Stephan P, et al. TNF-α−308 genotypes are associated with TNF-α and TGF-β1 mRNA expression in blood leucocytes of humans[J]. Cytokine, 2011, 53(3): 306-310. DOI:10.1016/j.cyto.2010.11.018 |
| [28] |
Gossner A G, Bennet N, Hunter N, et al. Differential expression of Prnp and Sprn in scrapie infected sheep also reveals Prnp genotype specific differences[J]. Biochemical and Biophysical Research Communications, 2009, 378(4): 862-866. DOI:10.1016/j.bbrc.2008.12.002 |
| [29] |
Lee E, Cho S, Kim K, et al. An integrated approach to infer causal associations among gene expression, genotype variation, and disease[J]. Genomics, 2009, 94(4): 269-277. DOI:10.1016/j.ygeno.2009.06.002 |
| [30] |
Olsson M, Olsson B, Jacobson P, et al. Expression of the selenoprotein S (SELS) gene in subcutaneous adipose tissue and SELS genotype are associated with metabolic risk factors[J]. Metabolism, 2011, 60(1): 114-120. DOI:10.1016/j.metabol.2010.05.011 |
| [31] |
Yao J, Liao K, Li Q L, et al. mRNA expression of glutathione S-transferase Pi (GSTP1) under heat stress and association of genotypes with heat tolerance ability in Holstein[J]. Journal of Animal and Veterinary Advances, 2011, 10(6): 757-763. DOI:10.3923/javaa.2011.757.763 |
 2019, Vol. 26
2019, Vol. 26

