2. 中国水产科学研究院淡水渔业研究中心, 江苏 无锡 214081
2. Freshwater Fisheries Research Center, Chinese Academy of Fishery Sciences, Wuxi 214081, China
白细胞介素17 (Interleukin-17, IL-17)是一种T细胞来源细胞因子, 最初被定义为细胞毒性T淋巴细胞抗原8 (cytotoxic T lymphocytes antigen 8, CTLA-8), 主要由单核细胞、CD+记忆T淋巴细胞分泌, 可通过激活中性粒细胞吸引趋化因子(如IL-8), 诱导相关促炎细胞因子和抗菌肽(AMPs)等而产生强烈的炎症反应[1-3]。哺乳动物IL-17家族有IL-17A~IL-17F共6个成员[4], 其中, IL-17A与IL-17F不仅介导IL-6、IL-8、粒细胞集落刺激因子等炎性因子, 还可以与TNF-α、IL-1等细胞因子协同作用, 增强炎症反应[5-6], 并与宿主防御[7-8]、自身免疫性疾病[9-10]和癌症[11]等相关。
Gunimaladevi等[12]于2006年首先在斑马鱼(Danio rerio)中鉴定克隆了IL-17家族成员基因IL-17A/F1、IL-17A/F2、IL-17A/F3、IL-17C和IL-17D, 分析了其结构, 其中因为鱼类IL-17A/F与哺乳动物IL-17A和IL-17F有着相近的相似性而命名为A/F, 测定了正常组织和刺激组织中各成员的表达水平。随后, 在东方红鳍鲀(Takifugu rubripes)[13]、虹鳟(Oncorhynchus mykiss)[14-15]、鮸(miiuy croaker)[16]、青鳉(Oryzias latipes)[17]、欧洲鲈(Dicentrarchus labrax)[18]中也鉴定到了IL-17家族成员。至今除了哺乳动物中IL-17E的同源基因在硬骨鱼类中没被发现外, 其余的均被找到, 且在硬骨鱼类发现了一个IL-17家族新成员IL-17N[18-20]。Wang等[20]对IL-17N的来源进行了分析, 认为IL-17N属于IL-17A/F亚家族。硬骨鱼类IL-17N的相关研究主要集中在基因克隆和组织表达方面, 对于其功能研究相对较少。
本研究使用同源搜索和基因克隆的方法鉴定了鲤(Cyprinus carpio)的两个IL-17N基因, 通过鱼类IL-17N基因的线性分析、鱼类IL-17N同源性分析和系统树构建, 揭示了IL-17N的进化地位; 使用实时定量PCR测定CcIL-17Ns (Cyprinus carpio IL-17Ns)在鲤的胚胎发育阶段、早期发育阶段、夏花和成鱼不同组织中的表达模式以及感染嗜水气单胞菌(Aeromonas hydrophila)对CcIL-17Ns的表达影响; 利用原核表达的方法获得可溶的鲤IL-17N重组蛋白, 通过测定鲤IL-17N重组蛋白孵育后鲤肾组织中促炎细胞因子(IL-1β、IFN-γ和IL-6)、趋化因子CCL20及NF-κB、TRAF6 (TNF receptor associated factor 6)基因的表达量的变化, 进一步验证鲤IL-17N的促炎功能。
1 材料与方法 1.1 实验鱼鲤早期胚胎样品:水温22 ℃下, 授精后0.5 h (胚盘出现)、12 h (原肠中期)、25 h (肌节出现)、35 h (脑泡出现)、60 h (心、眼点出现)、120 h (即将破膜)的受精卵, 每次取样20~30枚受精卵并保存于RNA sample protector中。鲤仔鱼样品:水温22 ℃, 受精卵出膜后1 d、4 d、7 d、14 d、26 d的仔鱼, 每次取样20~30尾仔鱼并保存于RNA sample protector中。鲤夏花样品:采集出膜后培育35 d, 体长约3 cm的鲤夏花心、肝、脾、肾、鳃、皮肤、脑、肠和肌肉等9个组织, 取20尾鱼, 每种组织做5份混合样并保存于RNA sample protector中。取鲤成鱼(4尾, 平均体重500 g)心、肝、脾、体肾、头肾、肠、脑、鳃、皮肤和肌肉10个组织, 于–80 ℃保存。感染嗜水气单胞菌[1.4×108 colony-forming units (CFU)/mL]前两周, 将平均体重500 g的鲤暂养于两个装有1000 L水的鱼缸中(每个鱼缸15尾), 1周适应后, 实验组每尾鱼腹腔注射0.1 mL嗜水气单胞菌, 而对照组的鱼注射等量的生理盐水溶液。感染组和对照组分别于感染后第1、3、7天采集鲤的脑、肝、心、体肾、肌肉、肠、皮肤、脾、鳃和头肾等组织, –80 ℃保存。所有鱼均采自中国水产科学研究院淡水渔业研究中心宜兴养殖基地。
1.2 实验引物根据Blast和线性分析在鲤基因组中挖掘到的2个CcIL-17N基因序列, 设计引物, P1-P2、P1-P3分别用于克隆验证CcIL-17Na和CcIL-17Nb全长cDNA。由于CcIL-17Na和CcIL-17Nb cDNA序列一致性较高, 设计一对共用引物P4-P5测定CcIL-17Ns的表达水平。在NCBI数据库(https://www.ncbi.nlm.nih.gov)搜索鲤NF-κB (登录号: a为GU064560.2, b为HM535646.1)、TRAF6 (登录号: LHQP01010838.1)、CCL20 (登录号: AJ245635)、IL-1β (登录号: a: KC858890.1、b: KC858889.1)、IFN-γ (登录号: NW_017537846.1)、IL-6 (登录号: XM_026248516.1、LHQP01001186.1), 由于鲤为四倍体, 为保证设计引物能测定所有旁基因的表达, 把鲤已有基因与斑马鱼等鱼的同源基因进行比对, 寻找基因的保守部位, 设计定量引物对, P6-P7、P8-P9、P10-P11、P12-P13、P14-P15、P16-P17分别用于TRAF6、NF-κB、IL-1β、IL-6、IFN-γ、CCL20基因的定量表达测定。P18-P19是根据鲤β-actin基因(登录号: M24113)设计的定量引物, 作为内参基因。P20-P21为原核表达扩增CcIL-17N片段引物。引物均由苏州金唯智生物科技有限公司合成, 详细内容见表 1。
|
|
表 1 序列验证和qPCR所用引物 Tab.1 Primers used for sequences validation and qPCR |
鲤IL-17N基因所在连锁群信息来自NCBI (http://www.ncbi.nlm.nih.gov), 雀鳝(Lepisosteus oculatus)、斑马鱼、三刺鱼(Gasterosteus aculeatus)等鱼类的IL-17N线性分析使用Genomicus (v87.01)[21]。使用MatGAT软件分析鲤及其他鱼类IL-17家族氨基酸序列的一致性和相似性。在MEGA 7.0软件[22]中进行鲤及其他鱼类IL-17家族多序列比对, 比对结果用邻接法, 以1000次自检率构建N-J系统进化树。使用protparam在线工具对鲤IL-17N基因的相关理化性质进行预测分析(http://web.expasy.org/cgi-bin/protparam/protparam), SignalP 4.1在线工具[23] (http://www.cbs.dtu.dk/services/SignalP/)预测信号肽。
1.4 提取总RNA和实时荧光定量PCR (qPCR)分析使用总RNA提取试剂盒(Omega Bio-Tek, 上海)提取各组织总RNA并测定OD260/OD280 (1.8~2.0), 并于1%琼脂糖凝胶电泳, 检测RNA完整性。采用Prime ScriptTM RT Master Mix (Perfect Real Time)(TaKaRa, 大连)试剂盒进行反转录, 按照SYBR® Premix Ex Taq IITM (TaKaRa, 大连)说明书进行实时定量PCR, 测定CCIL-17Ns的表达水平, 使用β-actin作为内参。
实验结果采用2–∆∆Ct法[24]进行计算, 随后使用SPSS 20.0软件中的Turkey多重比较法和t检验进行差异显著性分析, P < 0.05为差异显著, P < 0.01为差异极显著。
1.5 原核重组表达质粒的构建及表达纯化根据鲤IL-17N基因序列, 设计扩增成熟蛋白引物。在正、反向引物的5′端分别添加BamH I和Sal I的酶切位点及保护碱基(表 1), 以CcIL-17N基因的T-克隆质粒为模板, PCR扩增目的序列。使用胶回收试剂盒(上海博彩生物技术公司)回收PCR产物, 经BamH I和Sal I限制性内切酶(TaKaRa, 大连)酶切后, 与被同样酶切过的pMAL-c2X (杭州研真科技有限公司)连接, 构建CcIL-17N表达载体pMAL-c2X-17N。将连接产物转化至大肠杆菌DH5α敏感态细胞(北京全式金生物有限公司), 使用菌液PCR和双酶切鉴定筛选阳性单克隆, 并送至苏州金唯智生物科技有限公司测序。
将测序准确的重组质粒pMAL-c2X-17N和pMAL-c2X空载分别转入大肠杆菌(Escherichia coli) Transetta (DE3)感受态细胞(北京全式金生物有限公司), 挑取单克隆于10 mL含氨苄青霉素的LB液体培养基中, 37 ℃, 200 r/min培养过夜。次日, 以1 : 100在含氨苄青霉素的LB液体培养基中扩大培养, 37 ℃, 200 r/min培养, OD600值0.5~0.8, 加入终浓度为0.5 mmol/L的IPTG于25 ℃, 诱导8~12 h。离心收集细菌, 超声后取总菌和上清蛋白进行SDS-PAGE电泳。
上清裂解液用Ni-NTA (无锡天演生物技术科技有限公司)亲和层析, 使用SCGTMUV-VIS Detector (苏州赛普仪器有限公司)分离纯化。
1.6 鲤IL-17N重组蛋白(MBP-17N)对鲤炎症因子、趋化因子以及NF-κB、TRAF6的表达影响纯化后的重组蛋白MBP-17N和MBP用多粘菌素B树脂(G-Biosciences, 美国)去除内毒素, 并用鲎试剂(厦门鲎试剂生物科技股份有限公司)检测内毒素残留, 将去除内毒素的蛋白超滤浓缩(10 kD)(Millipore, 美国)于无内毒素的PBS中, 考虑到镍柱纯化蛋白总存在杂蛋白, 参照王钦等[25]使用与已知浓度的BSA蛋白标准品(Sigma, 美国)进行SDS-PAGE电泳, 根据目的带与牛血清白蛋白(BSA)条带灰度(Tanon GIS imagine), 计算纯化后MBP-17N和MBP蛋白的浓度。
取新鲜的鲤体肾, 放入一个含有RPMI-1640 (Thermo Scientific, 美国)的培养皿中, 切成1~ 3 mm的小块。将组织块分装到含400 µL RPMI- 1640 (含10%链青霉素)的六孔板中, 每孔5~6块, 约2 h后, 待组织块贴于六孔板壁上, 将培养液换成400 µL含10%胎牛血清和10%链青霉素的RPMI-1640, 28 ℃, 5% CO2孵育过夜后, 用不同浓度的MBP-17N (0.1 ng/mL, 1 ng/mL, 10 ng/mL, 100 ng/mL)、MBP蛋白对照组(100 ng/mL的MBP蛋白)和N83对照组(83 ℃失活的MBP-17N蛋白)分别培养孵育鲤体肾, 8 h后从组织中提取总RNA。使用实时荧光定量PCR法测定样品中NF-κB、TRAF6、CCL20、IL-1β、IFN-γ、IL-6的表达。
2 结果与分析 2.1 鲤IL-17N基因序列使用基因同线性分析和氨基酸序列同源搜索, 在NCBI中鉴定到2个鲤IL-17N基因, 分别位于LHQP01016936, LHQP01050094连锁群上, 由连锁群上TOP3B基因内含子2的互补序列编码, 命名为CcIL17Na和CcIL17Nb。使用RT-PCR和PCR验证, 测序表明只存在个别碱基差异, 可能是SNP位点。CcIL17Na和CcIL17Nb结构一致, 均由3个外显子和两个内含子组成, 外显子长度相同, 均为15 bp、176 bp、220 bp, 编码136个氨基酸; 但内含子存在差异, 分别为1439 bp、257 bp和1044 bp、284 bp。使用SignalP 4.1分析, N端起始的22个氨基酸为信号肽, 成熟蛋白均含有IL-17家族保守的4个半胱氨酸, 构成2个二硫键, 分子量分别为15.41 kD和15.33 kD, 等电点分别为5.74和5.47。CcIL-17Ns与其他硬骨鱼类IL-17N以及鲤IL-17家族其他成员间同源性比较, 结果, CcIL-17Na、CcIL-17Nb与草鱼(Ctenopharyngodon idellus)、斑马鱼、尼罗罗非鱼(Oreochromis niloticus)、红鳍东方鲀和青鳉、虹鳟等鱼IL-17N的相似性分别为65.3%~97.1%、64.7%~ 96.3%, 与鲤IL-17家族其他成员的相似性分别为32.9%~51.4%、31.4%~50.7% (表 2)。
|
|
表 2 鲤与其他鱼类IL-17N以及鲤IL-17家族成员氨基酸的一致性和相似性百分比 Tab.2 Percentage of amino acid similarity and identity of CcIL-17N with other known IL-17N and CcIL-17 members |
比较鱼类IL-17N附近基因, 发现除了东方红鳍鲀的相邻基因是AEBP1和NR5A1外, 在雀鳝、腔棘鱼(Coelacanth)、斑马鱼、棘鱼(bristling)、尼罗罗非鱼、青鳉、鲤、金线鲃(Sinocyclocheilus grahami)中, IL-17N相邻基因均为SDF2L1 (stromal cell derived factor 2 like 1)和PPM1F (Protein phosphatase 1F) (图 1)。
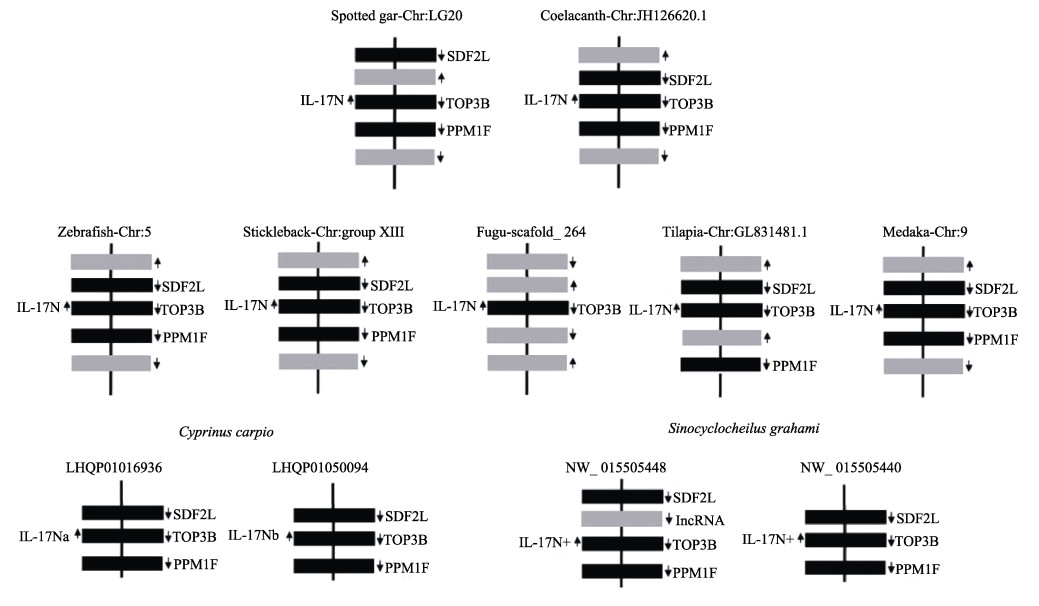
|
图 1 硬骨鱼类及其祖先IL-17N共线性分析示意图
PPM1F: Protein phosphatase 1F; SDF2L1: stromal cell derived factor 2 like 1; TOP3B: DNA topoisomerase 3-beta-1. 箭头方向表示转录方向. Fig.1 Schematic diagram of synteny of IL-17N in teleosts and their ancestors PPM1F: Protein phosphatase 1F; SDF2L1: stromal cell derived factor 2 like 1; TOP3B: DNA topoisomerase 3-beta-1. Arrows indicate the transcriptional orientation. |
鱼类IL-17家族成员N-J系统进化树显示鱼类IL-17家族7个成员形成6支, 除了IL-17C一支置信值为63%外, 其余均大于70%。其中, IL-17A/F1、IL-17A/F3先各自聚在一起, 然后再以76%置信度聚为一支。鲤IL-17N与同属鲤科鱼类的斑马鱼、草鱼先以94%置信值聚在一起, 然后与鲑鳟鱼类的虹鳟和大西洋鲑、以及鳉、东方红鳍鲀和尼罗罗非鱼等以85%的置信值聚在一起, 再以71%的置信值与硬骨鱼类二倍体祖先雀鳝的IL-17N聚为一支, 最后与腔棘鱼以95%置信值聚为可靠的一支, IL-17N支与IL-17A/F2支聚成一支的置信值只有43% (图 2)。
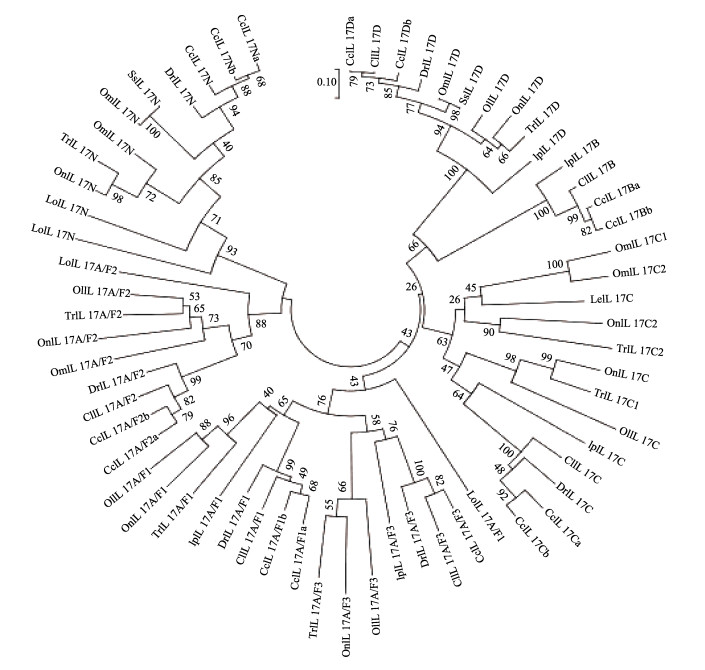
|
图 2 鱼类IL-17家族成员系统进化树 将氨基酸序列与ClustalOmega程序比对, 并使用MEGA 7软件中的邻接法构建无根系统发育树。节点处数字代表10000次自举检验置信度。各氨基酸序列的GenBank登录号:草鱼(Ctenopharyngodon idella, Ci): IL-17A/F1 KC978892, A/F2 KP412312, A/F3 AKM20919, IL-17C AKM20917, IL-17-D AGW43284;斑马鱼(Danio rerio, Dr): IL-17A/F1 NM_001020787, A/F2 NP_001018634, A/F3 NP_001018626, IL-17C NP_001018624, IL-17D NM_001020789;斑秘鳉(Aphanius punctatus, Ip): IL-17A/F1 XP_017331302, A/F3 XP_017310921, IL-17B XP_017341329, IL-17C1 XP_017320727, IL-17C2 XP_017320728, IL-17D XP_017326425;尼罗罗非鱼(Oreochromis niloticus, On): IL-17A/F1 XP_003437645, A/F2 XP_003437800, A/F3 XP_003440884, IL-17C1 XP_003449294, IL-17C2 XP_005447868, IL-17D XP_003456654, IL-17N LG12 (347611152:426406-433928);东方红鳍鲀(Takifugu rubripes, Tr): IL-17A/F1 AB522594, A/F2 BAI82579, A/F3 AB522596, IL-17C1 BAI82581, IL-17C2 BAI82582, IL-17D XP_003962280, IL-17N AB522600;青鳉(Oryzias latipes, Ol): IL-17A/F1 AB567679, A/F2 NP_001191713, A/F3 AB567681, IL-17C NP_001191723, IL-17-D BAJ41375, IL-17N NP_001191717, 虹鳟(Oncorhynchus mykiss, Om): IL-17A/F2NP_001118091, IL-17C1 CAW30792, IL17C2 CAW30793, IL-17D CAE45584, IL-17N KJ921981, 大西洋鲑(Salmo salar, Ss): IL-17D NP_001134365, IL-17N KJ921975;雀鳝(Lepisosteus oculatus, Lo): IL-17A/F1 ENSLOCG00000015957, IL-17A/F2 ENSLOCG00000015959;腔棘鱼(Latimeria chalumnae, Lc): IL-17C XP_006008330, IL-17N ENSLACG00000013517. Fig.2 Phylogenetic tree of fish IL-17 family members Amino acid sequences are aligned with the ClustalOmega program and an unrooted phylogenetic tree is constructed with the neighbor-joining method in the MEGA 7 software. Node values represent percent bootstrap confidence derived from 10, 000 replicates. The accession numbers of the molecules used are as follows: Ctenopharyngodon idella (Ci): IL-17A/F1 KC978892, A/F2 KP412312, A/F3 AKM20919, IL-17C AKM20917, IL-17-D AGW43284; Danio rerio (Dr): IL-17A/F1 NM_001020787, A/F2 NP_001018634, A/F3 NP_001018626, IL-17C NP_001018624, IL-17D NM_001020789; Aphanius punctatus (Ip): IL-17A/F1 XP_017331302, A/F3 XP_017310921, IL-17B XP_017341329, IL-17C1 XP_017320727, IL-17C2 XP_017320728, IL-17D XP_017326425; Oreochromis niloticus (On): IL-17A/F1 XP_003437645, A/F2 XP_003437800, A/F3 XP_003440884, IL-17C1 XP_003449294, IL-17C2 XP_005447868, IL-17D XP_003456654, IL-17N LG12 (347611152:426406-433928); Takifugu rubripes (Tr): IL-17A/F1 AB522594, A/F2 BAI82579, A/F3 AB522596, IL-17C1 BAI82581, IL-17C2 BAI82582, IL-17D XP_003962280, IL-17N AB522600; Oryzias latipes (Ol): IL-17A/F1 AB567679, A/F2 NP_001191713, A/F3 AB567681, IL-17C NP_001191723, IL-17-D BAJ41375, IL-17N NP_001191717; Oncorhynchus mykiss (Om): IL-17A/F2NP_001118091, IL-17C1 CAW30792, IL17C2 CAW30793, IL-17D CAE45584, IL-17N KJ921981, Salmo salar (Ss): IL-17D NP_001134365, IL-17N KJ921975; Lepisosteus oculatus (Lo): IL-17A/F1 ENSLOCG00000015957, IL-17A/F2 ENSLOCG00000015959; Latimeria chalumnae (Lc): IL-17C XP_006008330, IL-17N ENSLACG00000013517. |
使用qPCR测定了鲤胚胎发育阶段CcIL-17Ns的表达, 结果显示, 受精后0.5 h和12 h CcIL-17Ns表达量最高, 极显著高于25 h、35 h、60 h和120 h (P < 0.01);受精后35 h表达下降到最低点, 然后开始上升, 但表达量差异不显著(P > 0.05)。仔鱼(出膜后1—26 d) CcIL-17Ns的表达稳定与受精后25 h后各点无显著差异, 均低于受精后0.5 h和12 h (图 3A)。
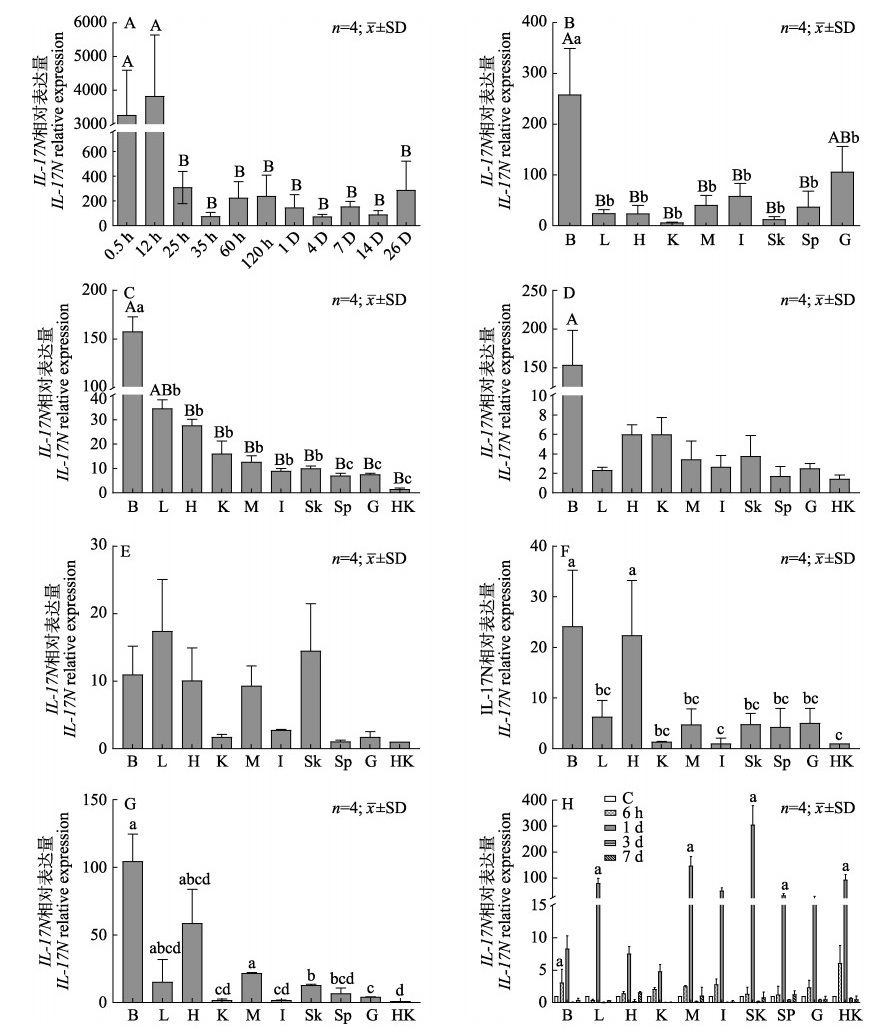
|
图 3 CcIL-17Ns在鲤不同发育阶段以及嗜水气单胞菌感染后各组织的表达量
A.鲤胚胎及仔鱼中CcIL-17Ns的表达水平, 以受精后120 h的相对表达量为1; B. CcIL-17Ns在鲤夏花不同组织中的表达, 肾的相对表达量为1; C. CcIL-17Ns在成鱼各组织中的表达; D~G分别为嗜水气单胞菌感染后6 d、1 d、3 d和7 d CcIL-17Ns的表达, C~G中各组织CcIL-17Ns的相对表达量以CcIL-17Ns在成鱼体肾中的相对表达量为1; H.嗜水气单胞菌感染后CcIL-17Ns的表达变化, 对照组为1. 不同小写字母表示不同组织间差异显著(P < 0.05), 不同的大写字母表示不同组织间差异极显著(P < 0.01). B:脑; L:肝; H:心; K:体肾; M:肌肉; I:肠; Sk:皮肤; Sp:脾; G:鳃; HK:头肾. Fig.3 Expression of CcIL-17Ns in different development and various tissues of healthy and Aeromonas hydrophila infected individuals Expression of CcIL-17Ns in embryo and larval carp. Date in A is normalized to the expression level of CcIL-17Ns at 120 h after fertilization. B. Expression of CcIL-17Ns in tissues of fingerling. Data in B is normalized to the expression level of the fingerling's kidney value of CcIL-17Ns. C. Expression of CcIL-17Ns in adult fish tissues. D-G. Expression of CcIL-17Ns on 6 h, 1 d, 3 d and 7 d after A. hydrophila infection, respectively. Data in C-G are expressed relative to the HK value.H.Changes in expression of CcIL-17Ns at different time after A. hydrophila infection. Data in H are expressed relative to the corresponding control group value. Different lowercase letters indicate significant difference at P < 0.05 and different capital letters indicate highly significant difference at P < 0.01. B: brain; L: liver; H: heart; K: trunk kidney; M: muscle; I: intestine; Sk: skin; Sp: spleen; G: gill; HK: head kidney. |
CcIL-17Ns在鲤夏花脑中的表达量最高, 显著高于鳃(P < 0.05), 极显著高于肝、心、体肾、肌肉、肠、皮肤和脾脏中的表达量(P < 0.01);在鳃中的表达量显著高于肾和肌肉中的表达量(P < 0.05) (图 3B)。
CcIL-17Ns在成鱼脑中的表达最高, 其次是肝, 均极显著高于其他组织(P < 0.01), 其余组织间没有明显差异(P > 0.05) (图 3C)。
2.2.3 嗜水气单胞菌刺激对CcIL-17Ns表达的影响实时定量PCR结果显示, 嗜水气单胞菌感染6 h, 脑中CcIL-17Ns的表达显著高于肝、心、体肾、肌肉、肠、皮肤、脾、鳃和头肾中的表达(P < 0.05);感染1 d, CcIL-17Ns的表达量从高到低依次为肌肉、肝、皮肤、脑、心、体肾、肠、鳃和头肾, 但差异不显著(P > 0.05); 3 d时, 脑和心的CcIL-17Ns表达显著高于肝、体肾、肌肉、肠、皮肤、脾、鳃和头肾(P < 0.05); 7 d时, 脑、心、肝和肌肉中CcIL-17Ns的表达显著高于体肾、肠、皮肤、脾、鳃和头肾(P < 0.05) (图 3D~图 3G)。
嗜水气单胞菌感染6 h, 脑中CcIL-17Ns基因表达量显著高于对照组(P < 0.05);感染1 d, 肝、心、体肾、肌肉、肠、皮肤、脾、鳃和头肾中CcIL-17Ns也显著上调(P < 0.05), 感染3 d和7 d, 各组织中CcIL-17Ns的表达均回落, 与对照组无显著性差异(P > 0.05) (图 3H)。
2.3 重组蛋白的表达与纯化将测序验证过的重组载体pMAL-c2X-17N和对照载体pMAL-c2X分别转化至大肠杆菌Transetta (DE3)敏感态细胞中, 25 ℃, 0.5 mmol/L IPTG, 诱导培养8~12 h, 离心收集菌体并超声裂解, SDS-PAGE电泳检测总菌和上清液中鲤IL-17N重组蛋白(MBP-17N)的表达情况。结果显示, 在分子量58 kD处出现一特殊条带, 其分子量与预期重组目的蛋白大小一致, 且在上清也有明显表达(图 5A)。使用镍柱纯化上清蛋白中重组蛋白MBP-17N和MBP蛋白, SDS-PAGE电泳检测, 得到单一条带与预期结果一致。每克总菌约获得0.09 mg MBP-17N蛋白, 每克总菌约获得0.46 mg MBP蛋白(图 5B)。
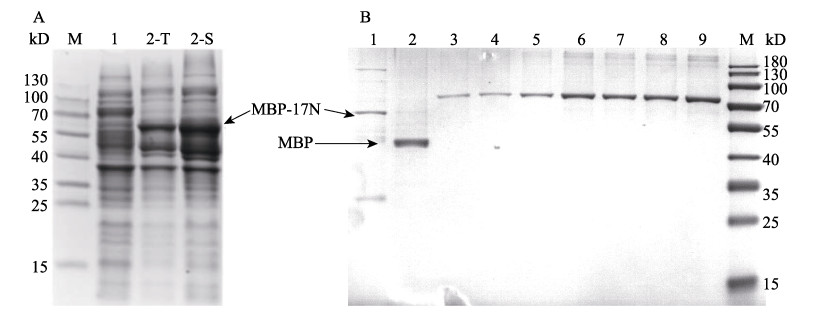
|
图 4 MBP-17N的表达及蛋白浓度的确定
A. M:预染蛋白marker; 1:对照; 2: MBP-17N; T:总菌; S:上清. B. M:预染蛋白marker; 1: MBP-17N蛋白; 2: MBP蛋白; 3~9:分别为0.250 μg、0.375 μg、0.50 μg、1.0 μg、1.5 μg、2.0 μg、2.5 μg牛血清白蛋白.箭头所指为目的条带. Fig.4 Expression of MBP-17N and the correction of protein concentration A. M: prestained protein marker; 1: control; 2: MBP-17N; T: total protein; S: supernatant protein. B. M: prestained protein marker; 1: MBP-17N protein; 2: MBP protein; 3~9: 0.250 μg, 0.375 μg, 0.50 μg, 1.0 μg, 1.5 μg, 2.0 μg, 2.5 μg BSA. The arrow points to the destination protein. |
|
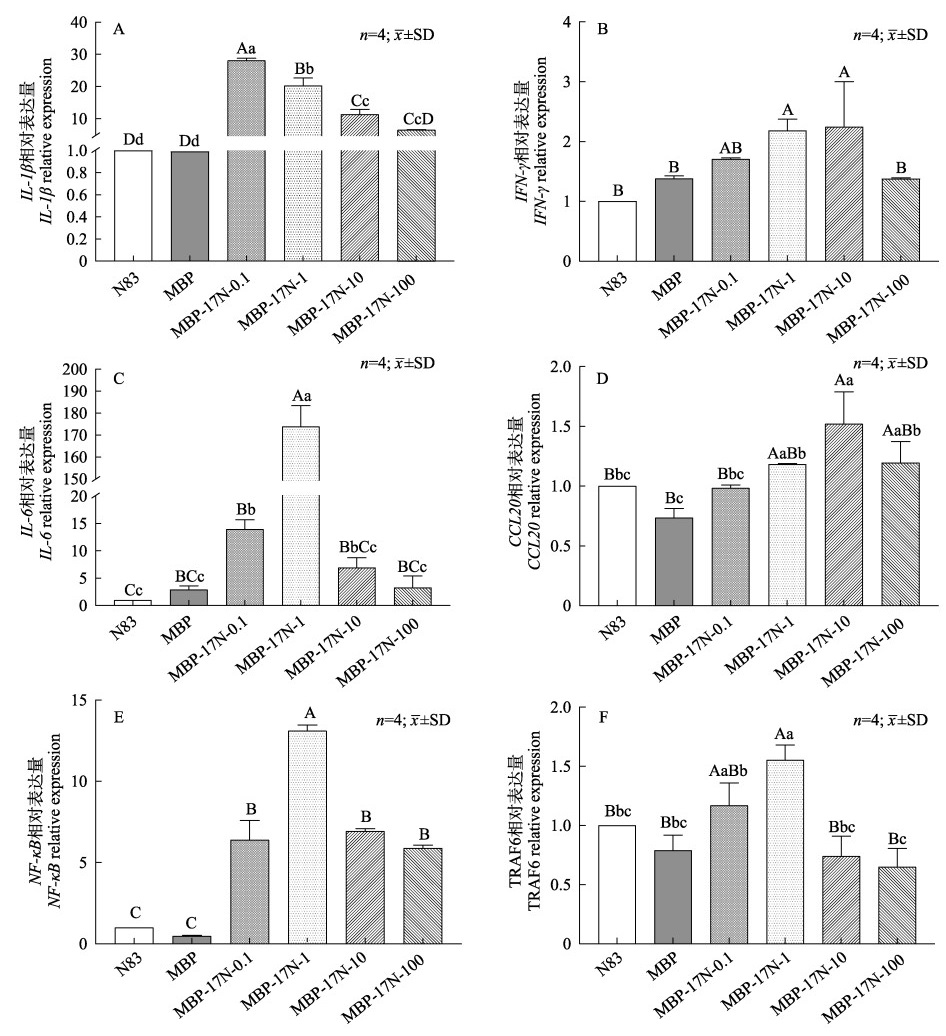
图 5 不同浓度的MBP-17蛋白对IL-1β、IFN-γ、IL-6、CCL20、NF-κB、TRAF6的表达影响 A: IL-1β; B: IFN-γ; C: IL-6; D: CCL20; E: NF-κB; F: TRAF6; N83: 83 ℃失活的MBP-17N蛋白; MBP:相同体积的100 ng/mL的MBP蛋白; MBP-17N-0.1、1、10、100分别表示0.1 ng/mL、1 ng/mL、10 ng/mL和100 ng/mL的MBP-17N.以N83组为空白对照, 对照组为1, 柱高为平均值; 不同小写字母表示不同组织间差异显著(P < 0.05), 不同的大写字母表示不同组织间差异极显著(P < 0.01). Fig.5 Effects of different concentrations of MBP-17N on IL-1β, IFN-γ, IL-6, CCL20, NF-κB and TRAF6 expression A: IL-1β; B: IFN-γ; C: IL-6; D: CCL20; E: NF-κB; F: TRAF6; N83: inactive MBP-17N at 83 ℃; MBP: 100 ng/mL of MBP protein; MBP-17N-0.1, 1, 10, and 100 represent MBP-17N protein with concentrations of 0.1 ng/mL, 1 ng/mL, 10 ng/mL and 100 ng/mL, respectively. Data is normalized to the expression level of the N83. Different lowercase letters indicate significant difference at P < 0.05; different capital letters indicate significant difference at P < 0.01. |
与MBP蛋白对照组相比, 所有基因的N83对照组均无显著性差异(P > 0.05), 下文对照组皆以MBP蛋白对照组为准。
使用不同浓度的MBP-17N蛋白(0.1 ng/mL、1 ng/mL、10 ng/mL)孵育肾组织8 h, 结果肾组织中的IL-1β的表达量均极显著上调(P < 0.01), 分别是对照组的28.11倍、18.98倍和9.59倍, 100 ng/mL时IL-1β显著上调, 是对照组的6.52倍(P < 0.05) (图 5A)。1 ng/mL和10 ng/mL的MBP-17N极显著上调IFN-γ的表达(P < 0.01), 100 ng/mL时, 与MBP蛋白对照组相比, IFN-γ的表达水平已无显著性差异(P > 0.05) (图 5B)。IL-6的表达呈现先升高后降低的趋势, 浓度1 ng/mL时达到峰值, 且极显著高于对照组(P < 0.01) (图 5C)。
MBP-17N的浓度为1 ng/mL、100 ng/mL时, CCL20的表达显著上调, 分别是对照组的1.21倍和1.22倍(P < 0.05), 在10 ng/mL时达到峰值, 且极显著高于对照组(P < 0.01)(图 5D)。
在MBP-17N蛋白的刺激下, NF-κB的表达极显著高于对照组, 表达水平随着浓度的升高先增加后下降, 在1 ng/mL时达到峰值(P < 0.01) (图 5E); 类似地, TRAF6的表达趋势与NF-κB相似, 也在1 ng/mL时显著高于对照组(P < 0.05) (图 5F)。
3 讨论本研究挖掘到两个鲤的IL-17N基因CcIL-17Na和CcIL-17Nb, 从线性分析、同源比对和系统树, 可以看出, IL-17N在鱼类二倍体祖先雀鳝和陆生动物祖先腔棘鱼中均存在且有一定的保守性, 而在哺乳动物中没找到该基因[17, 27], 应该是在进化过程中丢失所致。本研究中系统进化树的分析结果显示IL-17N和IL-17A/F2聚成一支的置信值只有43%, 不支持Wang等[20]推测的该基因为IL-17A/F亚家族成员的观点, 这可能与当时鱼类IL-17家族基因挖掘工作还处于起始阶段, 不能很好地揭示IL-17N的系统进化关系有关。鱼类IL-17家族成员中IL-17C一支置信值小于70%, 表明该基因在进化中变异比较大。
本研究组织表达分析结果表明, 无论是夏花还是成鱼CcIL-17Ns在脑中表达量均最高。这与青鳉和鲑[20]的IL-17N在脑中有高表达一致, 但该基因在神经系统中为何高表达, 其作用是什么还有待揭示; IL-17N在河鲀的头肾中表达量最高[13], 而鮸中则是在肾脏和鳃中有高表达[16], 说明了IL-17N的表达模式存在物种差异。此外, CcIL- 17Ns与CcIL-17A/F2的表达存在差异, CcIL-17A/F2在鳃的表达水平最高[3], 说明IL-17家族不同成员的表达存在差异, 这与斑马鱼的研究结果一致[12]。
有研究表明, IL-17家族参与了炎症反应。Kumari等[26]报道, LPS刺激大西洋鲑头肾, AsIL- 17D (Atlantic salmon IL-17D)在1 d时表达显著上调, 4 d和7 d时, AsIL-17D表达水平均降低。在Li等[3]的研究中, 嗜水气单胞菌感染后的前3 d, 鲤IL-17A/F2的表达在黏膜组织, 如头肾、体肾和脾中显著上调, 第7天表达水平降低。Korenaga等[13]证实, 草鱼IL-17N的表达水平在LPS刺激后4 h达到顶峰, 第2天表达水平下调。和上述结果类似, 鲤感染嗜水气单胞菌后, 6 h~1 d CcIL-17Ns在各组织的表达量均显著增加, 3 d和7 dCcIL-17Ns的表达均下调。这与Korenaga等[13]发现LPS能够上调河豚头肾细胞中的IL-17N的研究一致, 而与Wang等[20]LPS、poly I : C、PHA和PMA对大西洋鲑头肾细胞中IL-17N的表达没有影响的研究结果不一致, 说明IL-17N可能参与机体的炎症反应。嗜水气单胞菌诱导的CcIL-17Ns参与炎症反应, 但与CcIL-17A/F2表达模式不同, 暗示它们在炎症过程中具有不同的生物学功能。
研究表明哺乳动物IL-17A和F通过NF-κB信号通路的激活, 刺激炎症因子、趋化因子等的表达而参与炎症反应[27, 32]。转染IL-17基因入小鼠胃癌细胞, 结果趋化因子CCL20、CXCL2及免疫调节因子VEGF、MMP-9、IL-6的表达均增加[28]。至今研究表明, 鱼类IL-17家族成员也参与了免疫反应, 如鲑IL-17A/F2重组蛋白能够上调脾细胞中IL-6和IL-8的表达[29]; 草鱼IL-17D重组蛋白能刺激肾细胞炎症因子IL-1β、CXCL-8的表达[30]; 草鱼IL-17A/F1重组蛋白可上调促炎细胞因子IL-1β、TNF-α和IL-6的表达[31], 且两者均可通过经典的NF-κB炎症信号通路发挥作用; 鲤IL-17A/F2重组蛋白也可诱导肾组织中炎症因子IL-1β的表达[3]。本研究结果表明MBP-17N也能诱导炎症因子IL-1β、IFN-γ、IL-6以及趋化因子CCL20的表达, 表明鲤IL-17N也可通过NF-κB炎症信号通路参与炎症反应。本研究结果还显示1 ng/mL MBP-17N重组蛋白对所检测的因子均有显著的刺激作用, 而高浓度100 ng/mL则刺激作用减弱, 推测高浓度对细胞活性有影响[3]。
综上所述, 本研究分离并鉴定了鲤IL-17N基因, 对鱼类IL-17N的系统发育进行了比较全面的分析, 测定了在CcIL-17Ns的时空表达模式; 嗜水气单胞菌能够刺激CcIL-17Ns的显著上调, 以及鲤IL-17N重组蛋白能够诱导促炎细胞因子(IL-1β、IFN-γ 和IL-6)、趋化因子CCL20及NF-κB、TRAF6的上调, 表明IL-17N能通过NF-κB途径, 通过诱导趋化因子和炎症因子达到促炎作用。
| [1] |
Yao Z B, Fanslow W C, Seldin M F, et al. Herpesvirus saimiri encodes a new cytokine, IL-17, which binds to a novel cytokine receptor[J]. Immunity, 1995, 3(6): 811-821. DOI:10.1016/1074-7613(95)90070-5 |
| [2] |
Rouvier E, Luciani M F, Mattéi M G, et al. CTLA-8, cloned from an activated T cell, bearing AU-rich messenger RNA instability sequences, and homologous to a Herpesvirus saimiri gene[J]. Journal of Immunology, 1993, 150(12): 5445-5456. |
| [3] |
Li H X, Yu J H, Li J L, et al. Cloning and characterization of two duplicated interleukin-17A/F2 genes in common carp (Cyprinus carpio L.):Transcripts expression and bioactivity of recombinant IL-17A/F2[J]. Fish & Shellfish Immunology, 2016, 51: 303-312. |
| [4] |
Kolls J K, Lindén A. Interleukin-17 family members and inflammation[J]. Immunity, 2004, 21(4): 467-476. DOI:10.1016/j.immuni.2004.08.018 |
| [5] |
Gaffen S L. Structure and signalling in the IL-17 receptor family[J]. Nature Reviews Immunology, 2009, 9(8): 556-567. |
| [6] |
Weaver C T, Hatton R D, Mangan P R, et al. IL-17 family cytokines and the expanding diversity of effector T cell lineages[J]. Annual Review of Immunology, 2007, 25: 821-852. DOI:10.1146/annurev.immunol.25.022106.141557 |
| [7] |
Liang S C, Tan X Y, Luxenberg D P, et al. Interleukin (IL)-22 and IL-17 are coexpressed by Th17 cells and cooperatively enhance expression of antimicrobial peptides[J]. The Journal of Experimental Medicine, 2006, 203(10): 2271-2279. DOI:10.1084/jem.20061308 |
| [8] |
Onishi R M, Gaffen S L. Interleukin-17 and its target genes:Mechanisms of interleukin-17 function in disease[J]. Immunology, 2010, 129(3): 311-321. DOI:10.1111/j.1365-2567.2009.03240.x |
| [9] |
Ogura H, Murakami M, Okuyama Y, et al. Interleukin-17 promotes autoimmunity by triggering a positive-feedback loop via interleukin-6 induction[J]. Immunity, 2008, 29(4): 628-636. DOI:10.1016/j.immuni.2008.07.018 |
| [10] |
Boggio E, Clemente N, Mondino A, et al. IL-17 protects T cells from apoptosis and contributes to development of ALPS-like phenotypes[J]. Blood, 2014, 123(8): 1178-1186. DOI:10.1182/blood-2013-07-518167 |
| [11] |
Zou W, Restifo N P. TH17 cells in tumour immunity and immunotherapy[J]. Nature Reviews Immunology, 2010, 10(4): 248-256. DOI:10.1038/nri2742 |
| [12] |
Gunimaladevi I, Savan R, Sakai M. Identification, cloning and characterization of interleukin-17 and its family from zebrafish[J]. Fish & Shellfish Immunology, 2006, 21(4): 393-403. |
| [13] |
Korenaga H, Kono T, Sakai M. Isolation of seven IL-17 family genes from the Japanese pufferfish Takifugu rubripes[J]. Fish & Shellfish Immunology, 2010, 28(5-6): 809-818. |
| [14] |
Wang T H, Martin S A M, Secombes C J. Two interleukin-17C-like genes exist in rainbow trout Oncorhynchus mykiss that are differentially expressed and modulated[J]. Developmental & Comparative Immunology, 2010, 34(5): 491-500. |
| [15] |
Secombes C J, Wang T, Bird S. The interleukins of fish[J]. Developmental & Comparative Immunology, 2011, 35(12): 1336-1345. |
| [16] |
Yang Q, Sun Y N, Su X R, et al. Characterization of six IL-17 family genes in miiuy croaker and evolution analysis of vertebrate IL-17 family[J]. Fish & Shellfish Immunology, 2016, 49: 243-251. |
| [17] |
Kono T, Korenaga H, Sakai M. Genomics of fish IL-17 ligand and receptors:A review[J]. Fish & Shellfish Immunology, 2011, 31(5): 635-643. |
| [18] |
González-Fernández C, Chaves-Pozo E, Cuesta A. Identification and regulation of interleukin-17(IL-17) family ligands in the teleost fish European sea bass[J]. International Journal of Molecular Sciences, 2020, 21(7): 2439. DOI:10.3390/ijms21072439 |
| [19] |
Yang Q. Characterization, expression, and evolution analysis of miiuy croaker IL-1 and IL-17 family genes[D]. Zhoushan: Zhejiang Ocean University, 2017. [杨琼.鮸鱼IL-1及IL-17家族成员特征、表达及进化分析[D].舟山: 浙江海洋大学, 2017.]
|
| [20] |
Wang T H, Jiang Y S, Wang A, et al. Identification of the salmonid IL-17A/F1a/b, IL-17A/F2b, IL-17A/F3 and IL-17N genes and analysis of their expression following in vitro stimulation and infection[J]. Immunogenetics, 2015, 67(7): 395-412. DOI:10.1007/s00251-015-0838-1 |
| [21] |
Louis A, Muffato M, Roest Crollius H. Genomicus:five genome browsers for comparative genomics in eukaryota[J]. Nucleic Acids Research, 2012, 41(D1): D700-D705. DOI:10.1093/nar/gks1156 |
| [22] |
Kumar S, Stecher G, Tamura K. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [23] |
Krogh A, Larsson B R, von Heijne G, et al. Predicting transmembrane protein topology with a hidden Markov model:Application to complete genomes[J]. Journal of Molecular Biology, 2001, 305(3): 567-580. DOI:10.1006/jmbi.2000.4315 |
| [24] |
Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCt method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [25] |
Wang Q, Li J L, Tang Y K, et al. Prokaryotic expression and activity analysis of catalytic domain of Cyprinus carpio PLA2g3a1[J]. Journal of Nanjing Agricultural University, 2020, 43(2): 339-346. [王钦, 李建林, 唐永凯, 等. 鲤鱼PLA2g3a1催化活性区的原核表达及酶活性分析[J]. 南京农业大学学报, 2020, 43(2): 339-346.] |
| [26] |
Kumari J, Larsen A N, Bogwald J, et al. Interleukin-17D in Atlantic salmon (Salmo salar):Molecular characterization, 3D modelling and promoter analysis[J]. Fish & Shellfish Immunology, 2009, 27(5): 647-659. |
| [27] |
Shi P Q, Zhu S, Qian Y C. IL-17 signaling and function[J]. Chinese Journal of Cell Biology, 2011, 33(4): 345-357. [施沛青, 朱书, 钱友存. IL-17的信号传导及功能研究[J]. 中国细胞生物学学报, 2011, 33(4): 345-357.] |
| [28] |
Zhang Z P, Li Q X, Shan B E. Effects of IL-17 gene transfection on the biological characteristics of gastric cancer cells in mice[C]//Proceedings of the 9th National Congress of Immunology. Beijing: Chinese Society for Immunology, 2014: 559-560. [张智萍, 李巧霞, 单保恩.转染IL-17基因对小鼠胃癌细胞生物学特性的影响[C]//第九届全国免疫学学术大会论文集.北京: 中国免疫学会, 2014: 559-560.]
|
| [29] |
Monte M M, Wang T H, Holland J W, et al. Cloning and characterization of rainbow trout interleukin-17A/F2(IL-17A/F2) and IL-17 receptor A:Expression during infection and bioactivity of recombinant IL-17A/F2[J]. Infection and Immunity, 2013, 81(1): 340-353. DOI:10.1128/IAI.00599-12 |
| [30] |
Du L Y, Qin L, Wang X Y, et al. Characterization of grass carp (Ctenopharyngodon idella) IL-17D:Molecular cloning, functional implication and signal transduction[J]. Developmental & Comparative Immunology, 2014, 42(2): 220-228. |
| [31] |
Du L Y, Feng S Y, Yin L C, et al. Identification and functional characterization of grass carp IL-17A/F1:An evaluation of the immunoregulatory role of teleost IL-17A/F1[J]. Developmental & Comparative Immunology, 2015, 51(1): 202-211. |
| [32] |
Iwakura Y, Ishigame H, Saijo S, et al. Functional specialization of interleukin-17 family members[J]. Immunity, 2011, 34(2): 149-162. DOI:10.1016/j.immuni.2011.02.012 |
 2020, Vol. 27
2020, Vol. 27

