2. 浙江大学动物科学学院, 浙江 杭州 310058;
3. 上海实验动物研究中心, 上海 201203
2. College of Animal Sciences, Zhejiang University, Hangzhou 310058, China;
3. Shanghai Laboratory Animal Research Center, Shanghai 201203, China
肠道微生物对宿主食物消化、代谢和病害防御产生重要的影响[1]。鱼类肠道细菌与其消化、营养、生长和繁殖密切相关[2], 查明肠道细菌组成有助于深入了解鱼类生长和健康的机理。迄今已报道了多种鱼类的肠道细菌组成[3-24], 如大西洋鲑(Salmo sa-lar)[3, 17-19]、大西洋鳕(Gadus morhus)[4]、橙点石斑鱼(Epinephelus bleekeri)[5-6]、草鱼(Ctenopharyngodon idellus)[7, 11, 13, 16, 20]、银鲫(Carassius auratus gibe-lio)[8]、虹鳟(Oncorhynchus mykiss)[9, 21]、斑点叉尾鮰(Ictalurus punctatus)[10]、大口黑鲈(Micropterus salmoides)[10], 蓝鳃太阳鱼(Lepomis macroc-hirus)[10]、团头鲂(Megalobrama amblycephala)[11]、鲫(Carassius auratus)[11]、鲤(Cyprinus carpio)[11]、鲢(Hypophthalmichthys molitrix)[11]、黄颡鱼(Pelteo-bagrus fulvidraco)[11]、鳙(Aristichthys nobilis)[11-12]、匙吻鲟(Polyodon spathula)[12]、尖吻鲈(Lates cal-carifer)[14]、三刺鱼(Gasterosteus aculeatus)[15]、黄尾鰤(Seriola lalandei)[22]、紫红笛鲷(Lutjanus ar-gentimaculatus)[23]和齿鲷(Dentex dentex)[24]等。由于鱼类种类繁多, 同一种类不同个体之间甚至同一个体不同肠道部位之间细菌组成或多样性往往存在较大的差异[25-27], 已有的资料尚不足以反映鱼类肠道细菌的多样性以及分析决定肠道细菌多样性的因素。有研究表明, 宿主的遗传背景、免疫状况以及食物和环境条件等多种因素决定其肠道细菌的组成[28], 而宿主免疫系统与肠道微生物之间存在相互影响[29]。鱼类肠道细菌除了受宿主因素(如遗传、性别、体重、年龄、免疫力和肠动力)、环境因素(如水、食物、药物或抗生素)、微生物因素(黏附力、酶和代谢能力)、宿主个体差异与日常波动等影响[30]外, 还与宿主的发育有关[31-32]。此外, 在鱼类肠道的不同部位, 细菌多样性也存在变化[26-27]。研究发现, 草鱼中肠的细菌多样性高于前肠和后肠[33], 真鰶(Dorosoma cepedianum)后肠细菌多样性高于前肠[34], 鲢前肠细菌多样性高于后肠[11]。
花鲈(Lateolabrax japonicus)和日本黄姑鱼(Nibea japonica)分别隶属于鲈形目的鮨科和石首鱼科, 是亚洲, 特别是中国和韩国沿海地区重要的海水养殖鱼类种类[35-36]。有关花鲈[37]、加利福尼亚湾石首鱼(Totoaba macdonaldi)[38]、眼斑拟石首鱼(Sciaenops ocellatus)[39]、大黄鱼(Larimichthys cro-cea)[40-41]和舌齿鲈(Dicentrarchus labrax)[42]等鮨科鱼类和石首鱼科鱼类的肠道细菌已有一些报道。潘艳艳等[37]比较了投喂冰鲜鱼的花鲈在饥饿和恢复喂食期间肠道细菌多样性的变化, 但其研究中没有分析花鲈肠道细菌的空间异质性, 也没有比较花鲈肠道细菌组成与食物(冰鲜鱼)和养殖环境(海水)中细菌组成的差异。有关日本黄姑鱼的肠道细菌组成尚未见报道。本研究初步分析了舟山近岸海水网箱养殖花鲈和日本黄姑鱼的前肠、中肠和后肠的细菌多样性及其与食物(配合饲料)和养殖环境的关系, 目的是揭示近岸网箱养殖条件下海水鱼类肠道细菌多样性及其空间异质性的特征, 为开发新型饲料添加剂提供科学依据。
1 材料与方法 1.1 鱼类肠道、饲料和海水的采样2017年4月28日9:00~12:00采集舟山登步岛(29°52′N, 122°18′E)近岸网箱中养殖的日本黄姑鱼肠道细菌样品。日本黄姑鱼投喂配合饲料(粗蛋白含量为48.5%, 粗脂肪含量为11.0%, 德清县鸿利饲料有限公司生产)。鱼类肠道采样步骤如下:将鱼从网箱中捕出, 放入预先加入丁香油(浓度120 mg/L)的海水水槽中麻醉, 待鱼完全失去平衡后捞出。用75%酒精消毒鱼体表, 再用经酒精消毒的剪刀将鱼腹部剪开, 用灭菌镊子取出鱼内脏团放置在用酒精消毒的冰板上, 分离出肠道并去除肠道内容物。按预先确定的位置分别采集前肠(胃末端至第一个肠回转处)、中肠(前肠和后肠之间)和后肠(肠道后部具有明显环节处至泄殖腔)样品, 将每个肠道样品放入灭菌离心管(2 mL)中, 迅速投入液氮罐内冷冻。所采集的日本黄姑鱼体重为(275±44) g, 体长为(25.5±0.5) cm (n=3)。采集肠道样品后随机从饲料袋中取约100 g配合饲料, 保存在灭菌的样品袋中。用5 L采水器在养殖网箱中间水面下50 cm处采水样, 保存在1000 mL灭菌玻璃瓶中。饲料样品和水样保存在盛有碎冰的便携式保温箱中。现场用表面温度计测定网箱中水温(15.2 ℃), 用Mettler-Toledo pH计(Mettler- Toledo International Inc., Leicester, UK)测定pH (7.42), 用IS/Mill-E盐度计(AS ONE Corporation, Osaka, Japan)测定盐度(27)。将采集的日本黄姑鱼肠道、配合饲料和海水样品在采样后5 h内运回位于浙江大学舟山校区的实验室。
2017年11月3日12:00~13:30采集舟山朱家尖(N29°53′, E122°21′)近岸网箱中养殖的花鲈肠道细菌样品。花鲈投喂冰鲜鱼。按照日本黄姑鱼的采样方法和步骤采集花鲈肠道(前肠、中肠和后肠)和养殖网箱内海水样品。考虑到花鲈养殖过程中每天投喂的冰鲜鱼来源不同, 一次取样缺乏代表性, 故未对冰鲜鱼取样。所采集的花鲈体重为(133±37) g, 体长为(19.7±1.8) cm (n=3)。采样当天花鲈网箱中水温、pH和盐度分别为21 ℃、7.71和26。采样后在5 h内将花鲈肠道和海水样品运回浙江大学舟山校区实验室。
1.2 细菌基因组DNA的提取将鱼类肠道样品置于超净工作台(苏州安泰空气技术有限公司, 苏州, 中国)上的灭菌冰板上, 用灭菌手术刀将其切碎并转入灭菌离心管(10 mL)中, 用E.Z.N.A.® Soil DNA试剂盒(Omega Bio- Tek, Norcross, USA)提取肠道细菌DNA。配合饲料中细菌DNA的提取方法同肠道细菌。取100 mL海水水样, 用灭菌的玻璃滤器(Whatman 111106滤膜, 孔径0.22 μm, Whatman, Maidstone, UK)过滤, 再用E.Z.N.A.® Water DNA试剂盒(Omega Bio-Tek, Norcross, USA)提取滤膜上细菌的DNA。鱼类肠道、配合饲料和海水中细菌DNA保存在–20 ℃下。
1.3 PCR扩增和16S rDNA测序利用16S rDNA基因的可变区V3~V4通用引物338F (5′-ACTCCTACGGGAGGCAGCA-3′)和806R (5′-GGACTACHVGGGTWTCTAAT-3′)[43]进行PCR (ABI GeneAmp® 9700, ABI, USA)扩增。所用20 μL反应体系中包含4 μL 5×FastPfu缓冲液、2 μL 2.5 mmol/L dNTP、0.8 μL 5 μmol/L引物(正向和反向)、0.4 μL FastPfu聚合酶、0.2 μL BSA(牛血清白蛋白)和10 ng DNA模板。日本黄姑鱼肠道细菌、配合饲料和海水中细菌DNA扩增步骤如下:初始变性循环(95 ℃ 3 min)、29个变性循环(每个循环95 ℃, 30 s)、退火(55 ℃, 30 s)、延伸(72 ℃, 45 s)和最后延伸(72 ℃, 10 min)。花鲈肠道细菌和海水细菌DNA扩增步骤如下:初始变性循环(95 ℃ 3min)、28个变性循环(每个循环95 ℃, 30 s)、退火(53 ℃, 30 s)、延伸(72 ℃, 45 s)和最后延伸(72 ℃, 10 min)。对每个样品的细菌DNA扩增3份, 将PCR产物合并。用AxyPrepDNA凝胶提取试剂盒(Axygen, CA, USA)纯化PCR产物, 用Tris HCl洗脱后再用2%琼脂糖凝胶检测纯化后的PCR产物。利用Illumina MiSeq平台(上海美吉生物医药科技有限公司, 上海, 中国)进行细菌16S rDNA测序。
1.4 数据分析用FLASH和Trimmomatic对数据进行质控。用Usearch (版本7.0 http://drive5.com/uparse/)从优化序列中提取非重复序列。用RDP分类器贝叶斯算法将代表性序列分类为97%相似性水平的OTU, 置信度阈值为0.7 (Qiime平台, RDP分类器)。用Mothur计算基于随机样本的多样性指数, 包括Chao1指数、ACE指数、Shannon指数和Simpson指数。用单因素方差分析(one-way ANOVA)和邓肯检验(Duncan’s test)比较鱼类前肠、中肠和后肠细菌多样性指数的差异, 用配对数据t检验比较花鲈和日本黄姑鱼相同肠段位置(前肠、中肠或后肠)细菌多样性的差异。用主成分分析(PCoA)检验花鲈肠道细菌与养殖网箱内海水中细菌的关系, 以及日本黄姑鱼肠道细菌与配合饲料、海水中细菌间的关系。鱼类肠道细菌多样性指数表示为平均值±标准差(x±SD)。利用SPSS软件(版本21.0)进行邓肯检验和t检验, 设0.05为差异显著性水平。
2 结果与分析 2.1 鱼类肠道、配合饲料和海水中的细菌组成从花鲈和日本黄姑鱼肠道中共鉴定出732451条序列, 其中从花鲈肠道中鉴定出390494条序列, 从日本黄姑鱼肠道中鉴定出341957条序列。从配合饲料中鉴定出37529条序列。从养殖网箱海水中共鉴定出86523条序列, 其中从花鲈养殖网箱中鉴定出45418条序列, 从日本黄姑鱼养殖网箱中鉴定出41105序列。所有测序直至序列数不再随测序深度增加而明显增加为止, 故测序深度足以反映在97%相似度水平下的细菌多度。从鱼类肠道、配合饲料和养殖网箱内海水中测出的序列聚为886个OTU, 其中花鲈肠道测出374个OTU, 日本黄姑鱼肠道测出360个OTU, 日本黄姑鱼配合饲料测出155个OTU, 养殖网箱海水中测出463个OTU(花鲈网箱中测出429个OTU, 日本黄姑鱼网箱中测出191个OTU)。相对丰度较高的50个OTU见图 1。
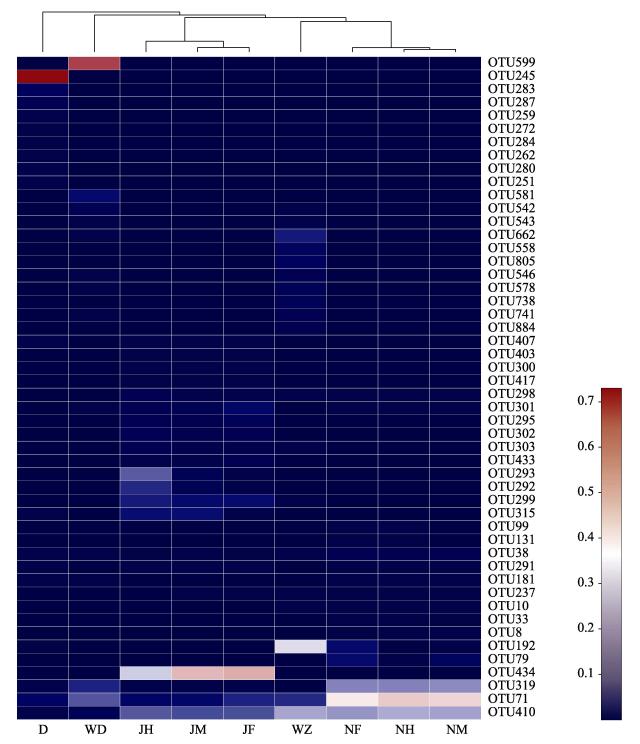
|
图 1 花鲈和日本黄姑鱼肠道、配合饲料和养殖海水中主要细菌OTU的相对丰度 OTU:可操作分类单元; JF:花鲈前肠; JM:花鲈中肠; JH:花鲈后肠; NF:日本黄姑鱼前肠; NM:日本黄姑鱼中肠; NH:日本黄姑鱼后肠; D:配合饲料; WD:日本黄姑鱼网箱中的海水; WZ:花鲈网箱中的海水. Fig.1 Relative abundance of bacterial OUT in Lateolabrax japonicus gut, Nibea japonica gut, formulated feed and seawater OTU: operational taxonomic units; JF: foregut of L. japonicus; JM: midgut of L. japonicus; JH: hindgut of L. japonicus; NF: foregut of N. japonica; NM: midgut of N. japonica; NH: hindgut of N. japonica; D: formulated feed; WD: seawater in the net pen for N. japonica farming; WZ: seawater in the net pen for L. japonicus farming. |
从花鲈和日本黄姑鱼肠道、配合饲料以及养殖网箱内的海水中鉴定出的优势菌分别隶属8个门、40个属。从图 2a可见, 厚壁菌门(Firmicutes, 71.6%)、变形菌门(Proteobacteria, 25.5%)、放线菌门(Actinobacteria, 1.7%)和拟杆菌门(Bacteroidetes, 0.7%)在花鲈肠道中占优势。厚壁菌门(91.3%)、变形菌门(4.2%)、软壁菌门(Tenericutes, 3.0%)和放线菌门(1.0%)在日本黄姑鱼的肠道中占优势。蓝细菌门(Cyanobacteria, 70.1%)、变形菌门(14.9%)、厚壁菌门(11.2%)、拟杆菌门(2.0%)和放线菌门(1.6%)在配合饲料中占优势。变形菌门(77.1%)、厚壁菌门(20.4%)和拟杆菌门(1.2%)在日本黄姑鱼养殖网箱海水中占优势。变形菌门(55.1%)、厚壁菌门(24.3%)、放线菌门(6.2%)、蓝细菌门(4.2%)、拟杆菌门(2.6%)、Marinimicrobia_SAR406_clade_ (2.2%)和绿弯菌门(Chloroflexi, 1.8%)在花鲈养殖网箱海水中占优势。
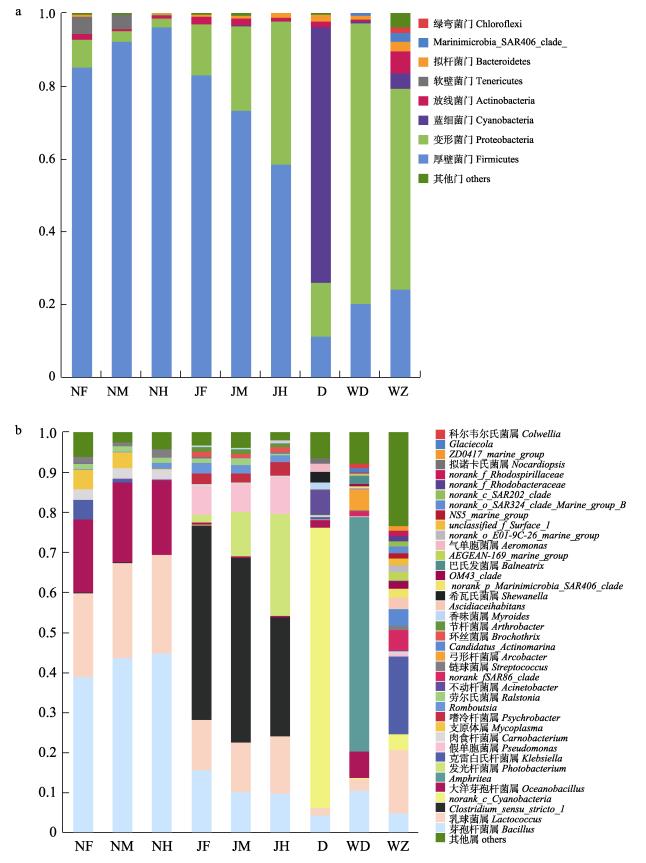
|
图 2 花鲈和日本黄姑鱼肠道、配合饲料和海水中的优势菌门(a)和属(b) JF:花鲈前肠; JM:花鲈中肠; JH:花鲈后肠; NF:日本黄姑鱼前肠; NM:日本黄姑鱼中肠; NH日本黄姑鱼后肠; D:配合饲料; WD:日本黄姑鱼养殖网箱内的海水; WZ:花鲈养殖网箱内的海水. Fig.2 Dominant bacteria in the Lateolabrax japonicus gut, Nibea japonica gut, formulated feed and seawater at the phylum (a) and genus (b) levels JF: foregut of L. japonicus; JM: midgut of L. japonicus; JH: hindgut of L. japonicus; NF: foregut of N. japonica; NM: midgut of N. japonica; NH: hindgut of N. japonica; D: formulated feed; WD: seawater in the net pen for N. japonica farming; WZ: seawater in the net pen for L. japonicus farming. |
从图 2b可见, 梭菌属(Clostridium_sensu_ stricto_1, 41.6%)、乳球菌属(Lactococcus, 13.1%)、发光杆菌属(Photobacterium, 12.9%)、芽孢杆菌属(Bacillus, 11.9%)和假单胞菌属(Pseudomonas, 7.7%)在花鲈肠道中占优势。芽孢杆菌属(42.5%)、乳球菌属(23.2%)、大洋芽孢杆菌属(Oceanobacillus, 19.0%)、支原体属(Mycoplasma, 3.0%)和肉食杆菌属(Carnobacterium, 2.5%)在日本黄姑鱼肠道中占优势。蓝细菌属(Norank_c_Cyanobacteria, 70.1%)、不动杆菌属(Acinetobacter, 6.2%)、芽孢杆菌属(4.2%)、希瓦氏菌属(Shewanella, 2.7%)、气单胞菌属(Aeromonas, 1.9%)、大洋芽孢杆菌属(1.9%)、乳球菌属(1.9%)、香味菌属(Myroides, 1.6%)和拟诺卡氏菌属(Nocardiopsis, 1.3%)在配合饲料中占优势。克雷白氏杆菌属(Klebsiella, 19.5%)、乳球菌属(15.8%)、Norank_f_SAR86_clade (5.1%)、芽孢杆菌属(4.9%)、Candidatus_Actinomarina (4.0%)、蓝细菌属(3.8%)、Ascidiaceihabitans (2.8%)、norank_p_Marinimicrobia_SAR406_clade_(2.2%)、AEGEAN-169_marine_group (2.1%)、OM43_clade (1.9%)、norank_o_E01-9C-26_marine_group (1.8%)、norank_o_SAR324_clade_Marine_group_B_(1.6%)、unclassified_f_Surface_1(1.5%)、NS5_marine_group (1.5%)、norank_c_SAR202_clade (1.4%)、norank_ f_Rhodobacteraceae (1.3%)、ZD0417_marine_ group (1.1%)和链球菌属(Streptococcus, 1.1%)在花鲈养殖网箱海水中占优势。Amphritea (58.5%)、芽孢杆菌属(10.6%)、大洋芽孢杆菌属(6.5%)、弓形杆菌属(Arcobacter, 5.3%)、乳球菌属(2.8%)、巴氏发菌属(Balneatrix, 2.2%)、Norank_f_SAR86_clade (1.3%)、Glaciecola (1.1%)和科尔韦尔氏菌属(Colwellia, 1.1%)在日本黄姑鱼养殖网箱海水中占优势。
2.2 鱼类肠道、配合饲料和海水中的细菌多样性从表 1可见, 花鲈和日本黄姑鱼肠道细菌之间ACE、Chao1指数、Shannon多样性指数和Simpson多样性指数无显著差异(配对数据t检验, P> 0.05)。在相同肠道部位(前肠、中肠或后肠), 花鲈和日本黄姑鱼之间的ACE、Chao1指数、Shannon多样性指数和Simpson多样性指数无显著差异(配对数据t检验, P>0.05)。
|
|
表 1 花鲈和日本黄姑鱼肠道、配合饲料和海水中的细菌多样性 Tab.1 Bacterial diversity in Lateolabrax japonicus gut, Nibea japonica gut, formulated feed and seawater |
从图 2a可见, 在门的水平上, 花鲈前肠的优势菌包括厚壁菌门(83.1%)、变形菌门(14.1%)和放线菌门(2.0%); 中肠的优势菌包括厚壁菌门(73.4%)、变形菌门(23.3%)和放线菌门(2.0%); 后肠的优势菌包括厚壁菌门(58.5%)、变形菌门(39.3%)和放线菌门(1.1%)。日本黄姑鱼前肠的优势菌包括厚壁菌门(85.3%)、变形菌门(7.5%)、软壁菌门(5.0%)和放线菌门(1.5%); 中肠的优势菌包括厚壁菌门(92.4%)、软壁菌门(4.0%)、变形菌门(2.7%)和放线菌门(0.5%); 后肠的优势菌包括厚壁菌门(96.2%)、变形菌门(2.5%)和放线菌门(0.8%)。
从图 2b可见, 在属的水平上, 花鲈前肠的优势菌包括Clostridium_sensu_stricto_1 (48.7%)、芽孢杆菌属(15.5%)、乳球菌属(12.7%)、假单胞菌属(7.2%)、Romboutsia(2.7%)、嗜冷杆菌属(Psychrob-acter, 2.6%)、发光杆菌属(2.0%)、环丝菌属(Bro¬c-hothrix, 1.4%)、劳尔氏菌属(Ralstonia, 1.2%)和节杆菌属(Arthrobacter, 1.1%); 中肠的优势菌包括Clostridium_sensu_stricto_1 (46.3%)、乳球菌属(12.3%)、发光杆菌属(11.1%)、芽孢杆菌属(10.2%)、假单胞菌属(6.8%)、嗜冷杆菌属(2.4%)、Rombo¬utsia (2.0%)、劳尔氏菌属(1.7%)、节杆菌属(1.0%)和环丝菌属(1.0%); 后肠的优势菌包括Clostridium_ sensu_ stricto_1 (29.8%)、发光杆菌属(25.5%)、乳球菌属(14.2%)、芽孢杆菌属(9.8%)、假单胞菌属(9.1%)、嗜冷杆菌属(3.4%)、Romboutsia (1.8%)、环丝菌属(1.3%)和节杆菌属(1.0%)。日本黄姑鱼前肠的优势菌包括芽孢杆菌属(39.0%)、乳球菌属(21.0%)、大洋芽孢杆菌属(18.3%)、支原体属(4.9%)、克雷白氏杆菌属(4.8%)、肉食杆菌属(2.5%)、链球菌属(1.4%)和劳尔氏菌属(1.3%); 中肠的优势菌包括芽孢杆菌属(43.7%)、乳球菌属(23.8%)、大洋芽孢杆菌属(20.1%)、支原体属(4.0%)、肉食杆菌属(2.5%)、劳尔氏菌属(1.3%)、链球菌属(1.0%)和克雷白氏杆菌属(0.8%); 后肠的优势菌包括芽孢杆菌属(44.7%)、乳球菌属(24.8%)、大洋芽孢杆菌属(18.7%)、肉食杆菌属(2.3%)、链球菌属(2.0%)、Romboutsia (1.5%)和劳尔氏菌属(1.3%)。从表 2可见, 同一种鱼类前肠、中肠和后肠的细菌多样性指数之间无显著差异(邓肯检验, P>0.05)。
|
|
表 2 花鲈和日本黄姑鱼前肠、中肠和后肠的细菌多样性 Tab.2 Bacterial diversity in foregut, midgut and hindgut of Lateolabrax japonicus and Nibea japonica |
从图 3可见, 花鲈前肠、中肠和后肠与其养殖网箱海水中共有的细菌OTU数分别为85、95和88。日本黄姑鱼前肠、中肠和后肠与配合饲料中共有的细菌OTU数分别为100、100和94, 与养殖网箱海水中共有的细菌OTU数分别为34、39和38。日本黄姑鱼肠道(前肠、中肠和后肠)、配合饲料和养殖网箱海水中共有的细菌OTU数为36个, 其中肠道与配合饲料中共有的细菌OTU数(72)多于肠道与养殖网箱海水中共有的细菌OTU数(32)。
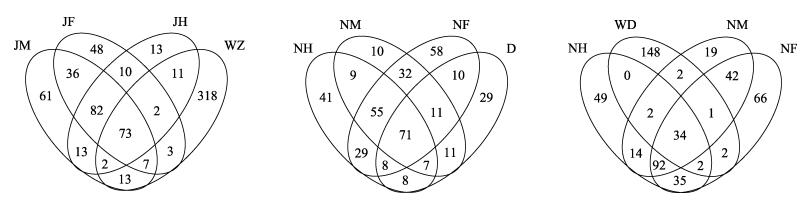
|
图 3 花鲈和日本黄姑鱼肠道、配合饲料和养殖网箱海水中共有和独有的细菌OTU JF:花鲈前肠; JM:花鲈中肠; JH:花鲈后肠; NF:日本黄姑鱼前肠; NM:日本黄姑鱼中肠; NH:日本黄姑鱼后肠; D:配合饲料; WD:日本黄姑鱼养殖网箱内的海水; WZ:花鲈养殖网箱内的海水. Fig.3 The number of common and unique OTUs between the gut of Lateolabrax japonicus and Nibea japonica, formulated feed and seawater JF: foregut of L. japonicus; JM: midgut of L. japonicus; JH: hindgut of L. japonicus; NF: foregut of N. japonica; NM: midgut of N. japonica; NH: hindgut of N. japonica; D: formulated feed; WD: seawater in the net pen for N. japonica farming; WZ: seawater in the net pen for L. japonica farming. |
从图 4可见, 花鲈肠道细菌OTU与日本黄姑鱼肠道细菌OTU明显分离(PCoA)。花鲈和日本黄姑鱼肠道细菌OTU与其各自养殖网箱内海水中的细菌OTU明显分离。花鲈与日本黄姑鱼肠道细菌群落的相似性分别高于每一种鱼类肠道细菌与其养殖网箱内海水中细菌之间的相似性。
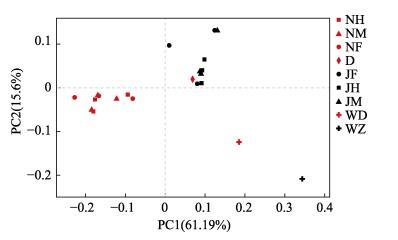
|
图 4 日本黄姑鱼和花鲈肠道、配合饲料和养殖网箱海水中细菌组成的差异 JF:花鲈前肠; JM:花鲈中肠; JH:花鲈后肠; NF:日本黄姑鱼前肠; NM:日本黄姑鱼中肠; NH:日本黄姑鱼后肠; D:配合饲料; WD:日本黄姑鱼养殖网箱内的海水; WZ:花鲈养殖网箱内的海水. Fig.4 The dissimilarity in bacterial community between the gut of Lateolabrax japonicus and Nibea japonica, formulated feed and seawater JF: foregut of L. japonicus; JM: midgut of L. japonicus; JH: hindgut of L. japonicus; NF: foregut of N. japonicus; NM: midgut of N. japonicus; NH: hindgut of N. japonicus; D: formulated feed; WD: seawater in the net pen for N. japonicus farming; WZ: seawater in the net pen for l. japonicus farming. |
据不完全统计, 迄今已报道了超过90种鱼类的肠道细菌组成[44]。有关鱼类肠道中的核心微生物种类仍有待确定[25]。在门水平上, 变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、厚壁菌门(Firmicutes)和梭杆菌门(Fusobacterium)通常在鱼类肠道中占优势[4, 9, 11-12, 33, 36-37]。在属水平上, 弧菌属(Vibrio)、发光杆菌属(Photobacterium)和梭菌属(Clostridium)通常占优势[43]。石首鱼科中, 加利福尼亚湾石首鱼肠道优势菌属包括发光杆菌属、弧菌属、气单胞菌属(Aeromonas)和希瓦氏菌属(Shewanella)[38]; 大黄鱼优势菌属包括芽孢杆菌属、乳球菌属、大洋芽孢杆菌属(Oceanobacillus)和Tyzzerella_3[41]。鮨科中, 舌齿鲈肠道细菌包括乳杆菌属(Lactobacillus)、假单胞菌属(Pseudomonas)、弧菌属(Vibrio)和伯克氏菌属(Burkholderia)[42]。本研究发现, 在门水平上, 花鲈肠道中厚壁菌门、变形菌门、放线菌门和拟杆菌门占优势; 日本黄姑鱼肠道中厚壁菌门、变形菌门、软壁菌门(Tenericutes)和放线菌门占优势。在属水平上, 花鲈肠道中梭菌属(Clost-ridium_sensu_stricto_1)、乳球菌属(Lactococcus)、发光杆菌属、芽孢杆菌属(Bacillus)和假单胞菌属占优势; 日本黄姑鱼肠道中芽孢杆菌属、乳球菌属、大洋芽孢杆菌属(Oceanobacillus)、支原体属(Mycoplasma)和肉食杆菌属(Carnobacterium)占优势。相比之下, 花鲈和日本黄姑鱼肠道优势菌在属水平上存在的差异大于其在门水平上表现出的差异, 这一结果表明, 在属水平上比在门水平上能够更确切地比较鱼类肠道细菌组成的差异, 同时这一结果也支持宿主遗传背景决定其肠道细菌组成的观点[28, 32]。本研究中, 在日本黄姑鱼肠道中发现了部分在大黄鱼肠道中出现的优势菌属(如芽孢杆菌属、乳球菌属和大洋芽孢杆菌属)[41], 但未发现加利福尼亚湾石首鱼肠道中出现的优势菌属[38]。在花鲈肠道中发现了部分齿鲈肠道中出现的优势菌属(如乳球菌属和假单胞菌属)[42]。鉴于不同研究之间在研究对象(鱼类种类)、研究方法(包括样品采集到细菌鉴定方法)和环境背景往往存在较大的差异[25-27], 尚不能通过简单比较不同的研究结果得出鱼类分类地位影响其肠道细菌群落结构的结论, 有关分类地位接近的鱼类种类肠道细菌是否相似性较高, 尚需直接的实验检验。
本研究发现, 日本黄姑鱼肠道细菌的ACE、Chao1指数和Simpson指数均低于配合饲料和养殖网箱海水中的细菌; 花鲈肠道细菌的ACE和Chao1指数低于其养殖网箱海水中的细菌, 但Simpson指数高于后者。Zhang等[41]报道大黄鱼和黑鲷肠道细菌的ACE、Chao1指数、Shannon指数和Simpson多样性均低于配合饲料和养殖网箱海水中细菌的相应指标, 但Wei等[40]认为大黄鱼肠道细菌的Shannon指数相对高于海水中细菌的Shannon指数。综合本研究和早期研究结果, 初步认为海水养殖鱼类肠道细菌多样性通常低于其所生活的海水中的细菌多样性。
有关鱼类肠道细菌群落的空间异质性已有一些研究报道[26-27, 34, 41]。例如, 生活在密西西比河流域的真鰶和鲢从前肠到后肠细菌多样性表现出不同的变化特点, 真鰶后肠细菌多样性高于前肠, 而鲢前肠细菌多样性高于后肠[34]。大黄鱼中肠细菌OTU数目和Chao1指数高于前肠, 而黑鲷前、中、后肠细菌OTU数目、ACE、Chao1指数、Shannon指数和Simpson指数均无显著差异[41]。本研究中, 同一种鱼类(花鲈或日本黄姑鱼)前肠、中肠和后肠间细菌多样性未表现出显著差异, 反映出两种鱼类肠道细菌空间异质性特点均不明显。这与对黑鲷肠道细菌空间异质性的研究结果[41]一致。在门的水平上, 从花鲈前肠到中肠和后肠, 变形菌门和发光杆菌属的相对丰度呈逐渐增加的趋势, 而厚壁菌门的相对丰度逐渐降低。相反, 从日本黄姑鱼前肠到中肠和后肠厚壁菌门的相对丰度呈逐渐增加的趋势。这与Ye等[34]对真鰶和鲢肠道细菌的研究结果类似。在属的水平上, Clostridium_ sensu_stricto_1在花鲈前肠、中肠和后肠的相对丰度均最高, 而在日本黄姑鱼肠道中的相对丰度较低(>0.01%); 大洋芽孢杆菌属在花鲈肠道中的相对丰度较低(1%), 但在日本黄姑鱼的前肠、中肠和后肠中相对丰度均较高(约20%)。这些结果表明花鲈和日本黄姑鱼前、中、后肠细菌群落具有不同的特点, 进一步证明遗传因素可能是决定鱼类肠道细菌(在属的水平上)差异的重要原因[30]。
研究表明饮食和环境条件可影响宿主肠道细菌组成[28]。食物条件是影响鱼类肠道细菌组成的重要因素[3-4, 14, 21, 34, 45]。花鲈饥饿前、后其肠道壁细菌群落发生明显变化[38]。摄食配合饲料的草鱼肠道细菌组成与饲料中细菌的相似性低于与养鱼池塘水和底泥中细菌的相似性[8], 然而大黄鱼和黑鲷肠道与其饲料中共有的细菌OTU数多于肠道与其养殖网箱内海水中共有的细菌OTU数[41]。本研究中, 日本黄姑鱼肠道细菌与配合饲料中共有的OTU数多于鱼类肠道细菌与养殖网箱海水中细菌共有的OTU数, 表明配合饲料中细菌对鱼类肠道细菌的影响可能超过网箱海水中细菌的影响, 这与研究大黄鱼和黑鲷肠道细菌的结论一致[41]。此外, 乳球菌属和芽孢杆菌属在花鲈和日本黄姑鱼肠道中均占优势, 肉食杆菌属同时出现在花鲈和日本黄姑鱼肠道、日本黄姑鱼配合饲料以及两种鱼类养殖网箱海水中, 支原体属出现在日本黄姑鱼肠道中。肠道中的芽孢杆菌属通常被认为有益于宿主的健康[46], 而肉食杆菌属[47]和支原体属[48]具有致病作用。花鲈和日本黄姑鱼肠道、配合饲料和养殖网箱海水中均发现肉食杆菌属, 表明饲料和海水可能是肉食杆菌属感染网箱养殖花鲈和日本黄姑鱼的途径[48]。支原体属出现在姬虾虎鱼(Gillichthys mirabilis)肠道中[49], 有时可在野生和养殖的鲑肠道微生物中占优势[50]。春季网箱养殖的日本黄姑鱼肠道中出现支原体属, 其影响有待进一步分析。
4 结论在门的水平上, 花鲈肠道优势菌包括厚壁菌门、变形菌门、放线菌门和拟杆菌门, 日本黄姑鱼肠道优势菌包括厚壁菌门、变形菌门、软壁菌门和放线菌门。在属的水平上, 花鲈肠道优势菌包括Clostridium_sensu_stricto_1、乳球菌属、发光杆菌属、芽孢杆菌属和假单胞菌属, 日本黄姑鱼肠道优势菌包括芽孢杆菌属、乳球菌属、大洋芽孢杆菌属、支原体属和肉食杆菌属。花鲈和日本黄姑鱼肠道细菌多样性低于养殖网箱内海水中的细菌多样性。配合饲料对日本黄姑鱼肠道细菌的影响可能大于海水对肠道细菌的影响。
| [1] |
Zha Y H, Eiler A, Johansson F, et al. Effects of predation stress and food ration on perch gut microbiota[J]. Microbiome, 2018, 6: 28. DOI:10.1186/s40168-018-0400-0 |
| [2] |
Song W, Li L Z, Huang H L, et al. The gut microbial community of Antarctic fish detected by 16S rRNA gene sequence analysis[J]. BioMed Research International, 2016, 2016: 1-7. |
| [3] |
Ringø E, Sperstad S, Kraugerud O F, et al. Use of 16S rRNA gene sequencing analysis to characterize culturable intestinal bacteria in Atlantic salmon (Salmo salar) fed diets with cellulose or non-starch polysaccharides from soy[J]. Aquaculture Research, 2008, 39(10): 1087-1100. DOI:10.1111/j.1365-2109.2008.01972.x |
| [4] |
Ringø E, Sperstad S, Myklebust R, et al. Characterisation of the microbiota associated with intestine of Atlantic cod (Gadus morhua L.)[J]. Aquaculture, 2006, 261(3): 829-841. DOI:10.1016/j.aquaculture.2006.06.030 |
| [5] |
Sun Y Z, Yang H L, Ling Z C, et al. Microbial communities associated with early stages of intensively reared orangespotted grouper (Epinephelus coioides)[J]. Aquaculture Research, 2015, 46(1): 131-140. DOI:10.1111/are.12167 |
| [6] |
Sun Y Z, Yang H L, Ma R L, et al. Molecular analysis of autochthonous microbiota along the digestive tract of juvenile grouper Epinephelus coioides following probiotic Bacillus pumilus administration[J]. Journal of Applied Microbiology, 2011, 110(4): 1093-1103. DOI:10.1111/j.1365-2672.2011.04967.x |
| [7] |
Wu S G, Wang G T, Angert E R, et al. Composition, diversity, and origin of the bacterial community in grass carp intestine[J]. PLoS ONE, 2012, 7(2): e30440. DOI:10.1371/journal.pone.0030440 |
| [8] |
Wu S G, Tian J Y, Gatesoupe F J, et al. Intestinal microbiota of gibel carp (Carassius auratus gibelio) and its origin as revealed by 454 pyrosequencing[J]. World Journal of Microbiology and Biotechnology, 2013, 29(9): 1585-1595. DOI:10.1007/s11274-013-1322-4 |
| [9] |
Ingerslev H C, von Gersdorff Jørgensen L, Lenz Strube M, et al. The development of the gut microbiota in rainbow trout (Oncorhynchus mykiss) is affected by first feeding and diet type[J]. Aquaculture, 2014, 424-425: 24-34. DOI:10.1016/j.aquaculture.2013.12.032 |
| [10] |
Larsen A M, Mohammed H H, Arias C R. Characterization of the gut microbiota of three commercially valuable warmwater fish species[J]. Journal of Applied Microbiology, 2014, 116(6): 1396-1404. DOI:10.1111/jam.12475 |
| [11] |
Li J Q, Ni J, Li J, et al. Comparative study on gastrointestinal microbiota of eight fish species with different feeding habits[J]. Journal of Applied Microbiology, 2014, 117(6): 1750-1760. DOI:10.1111/jam.12663 |
| [12] |
Li X M, Zhu Y J, Yan Q Y, et al. Do the intestinal microbiotas differ between paddlefish (Polyodon spathala) and bighead carp (Aristichthys nobilis) reared in the same pond?[J]. Journal of Applied Microbiology, 2014, 117(5): 1245-1252. DOI:10.1111/jam.12626 |
| [13] |
Ni J J, Yan Q Y, Yu Y H, et al. Factors influencing the grass carp gut microbiome and its effect on metabolism[J]. FEMS Microbiology Ecology, 2014, 87(3): 704-714. DOI:10.1111/1574-6941.12256 |
| [14] |
Xia J H, Lin G, Fu G, et al. The intestinal microbiome of fish under starvation[J]. BMC Genomics, 2014, 15(1): 266. DOI:10.1186/1471-2164-15-266 |
| [15] |
Ghanbari M, Kneifel W, Domig K J. A new view of the fish gut microbiome: Advances from next-generation sequencing[J]. Aquaculture, 2015, 448: 464-475. DOI:10.1016/j.aquaculture.2015.06.033 |
| [16] |
Li H, Zhong Q P, Wirth S, et al. Diversity of autochthonous bacterial communities in the intestinal mucosa of grass carp (Ctenopharyngodon idellus) (Valenciennes) determined by culture-dependent and culture-independent techniques[J]. Aquaculture Research, 2015, 46(10): 2344-2359. DOI:10.1111/are.12391 |
| [17] |
Neuman C, Hatje E, Zarkasi K Z, et al. The effect of diet and environmental temperature on the faecal microbiota of farmed Tasmanian Atlantic Salmon (Salmo salar L)[J]. Aquaculture Research, 2016, 47(2): 660-672. DOI:10.1111/are.12522 |
| [18] |
Neuman C, Hatje E, Smullen R, et al. The effect of fishmeal inclusion and prebiotic supplementation on the hindgut faecal microbiota of farmed Tasmanian Atlantic salmon (Salmo salar L)[J]. Journal of Applied Microbiology, 2018, 125(4): 952-963. DOI:10.1111/jam.13930 |
| [19] |
Dehler C E, Secombes C J, Martin S A M. Environmental and physiological factors shape the gut microbiota of Atlantic salmon parr (Salmo salar L)[J]. Aquaculture, 2017, 467: 149-157. DOI:10.1016/j.aquaculture.2016.07.017 |
| [20] |
Li T T, Long M, Li H, et al. Multi-omics analysis reveals a correlation between the host phylogeny, gut microbiota and metabolite profiles in cyprinid fishes[J]. Frontiers in Microbiology, 2017, 8: 454. |
| [21] |
Etyemez Büyükdeveci M, Balcázar J L, Demirkale İ, et al. Effects of garlic-supplemented diet on growth performance and intestinal microbiota of rainbow trout (Oncorhynchus mykiss)[J]. Aquaculture, 2018, 486: 170-174. DOI:10.1016/j.aquaculture.2017.12.022 |
| [22] |
Soriano E L, Ramírez D T, Araujo D R, et al. Effect of temperature and dietary lipid proportion on gut microbiota in yellowtail kingfish Seriola lalandi juveniles[J]. Aquaculture, 2018, 497: 269-277. DOI:10.1016/j.aquaculture.2018.07.065 |
| [23] |
Reshma K J, Sumithra T G, Nair A V, et al. An insight into the gut microbiology of wild-caught Mangrove Red Snapper, Lutjanus argentimaculatus (Forsskal, 1775)[J]. Aquaculture, 2018, 497: 320-330. DOI:10.1016/j.aquaculture.2018.08.008 |
| [24] |
Giménez Papiol G, Roque A, Estévez A. Variations in bacterial community of rearing water and gut of common Dentex, Dentex dentex (Linnaeus 1758), larvae using three microalgae management approaches[J]. Journal of the World Aquaculture Society, 2018, 49(5): 953-963. DOI:10.1111/jwas.12437 |
| [25] |
Le D, Nguyen P, Nguyen D, et al. Gut microbiota of migrating wild rabbit fish (Siganus guttatus) larvae have low spatial and temporal variability[J]. Microbial Ecology, 2020, 79(3): 539-551. DOI:10.1007/s00248-019-01436-1 |
| [26] |
Fogarty C, Burgess C M, Cotter P D, et al. Diversity and composition of the gut microbiota of Atlantic salmon (Salmo salar) farmed in Irish waters[J]. Journal of Applied Microbiology, 2019, 127(3): 648-657. DOI:10.1111/jam.14291 |
| [27] |
Parata L, Nielsen S, Xing X, et al. Age, gut location and diet impact the gut microbiome of a tropical herbivorous surgeonfish[J]. FEMS Microbiology Ecology, 2020, 96(1): 1-10. |
| [28] |
Tehrani A B, Nezami B G, Gewirtz A T, et al. Obesity and its associated disease: A role for microbiota?[J]. Neurogastroenterology & Motility, 2012, 24(4): 305-311. |
| [29] |
Nicholson J K, Holmes E, Kinross J, et al. Host-gut microbiota metabolic interactions[J]. Science, 2012, 336(6086): 1262-1267. DOI:10.1126/science.1223813 |
| [30] |
Wang A R, Ran C, Ringø E, et al. Progress in fish gastrointestinal microbiota research[J]. Reviews in Aquaculture, 2018, 10(3): 626-640. |
| [31] |
Li X H, Zhou L, Yu Y H, et al. Composition of gut microbiota in the gibel carp (Carassius auratus gibelio) varies with host development[J]. Microbial Ecology, 2017, 74(1): 239-249. DOI:10.1007/s00248-016-0924-4 |
| [32] |
Yan Q Y, Li J J, Yu Y H, et al. Environmental filtering decreases with fish development for the assembly of gut microbiota[J]. Environmental Microbiology, 2016, 18(12): 4739-4754. DOI:10.1111/1462-2920.13365 |
| [33] |
Yang G, Jian S Q, Cao H Z, et al. Changes in microbiota along the intestine of grass carp (Ctenopharyngodon idella): Community, interspecific interactions, and functions[J]. Aquaculture, 2019, 498: 151-161. DOI:10.1016/j.aquaculture.2018.08.062 |
| [34] |
Ye L, Amberg J J, Chapman D C, et al. Fish gut microbiota analysis differentiates physiology and behavior of invasive Asian carp and indigenous American fish[J]. The ISME Journal, 2014, 8(3): 541-551. DOI:10.1038/ismej.2013.181 |
| [35] |
Wu Y B, Ren X, Chai X J, et al. Replacing fish meal with a blend of poultry by-product meal and feather meal in diets for giant croaker (Nibea japonica)[J]. Aquaculture Nutrition, 2018, 24(3): 1085-1091. DOI:10.1111/anu.12647 |
| [36] |
Wang J, Tao Q Y, Wang Z, et al. Effects of fish meal replacement by soybean meal with supplementation of functional compound additives on intestinal morphology and microbiome of Japanese seabass (Lateolabrax japonicus)[J]. Aquaculture Research, 2017, 48(5): 2186-2197. DOI:10.1111/are.13055 |
| [37] |
Pan Y Y, Gu X Y, Zhang D M, et al. Effects of fasting and refeeding on Lateolabrax japonicus intestinal microflora diversity[J]. Journal of Fisheries of China, 2016, 40(5): 776-784. [潘艳艳, 顾晓英, 张德民, 等. 饥饿及恢复喂食对花鲈肠道菌群多样性的影响[J]. 水产学报, 2016, 40(5): 776-784.] |
| [38] |
González-Félix M L, Gatlin D M Ⅲ, Urquidez-Bejarano P, et al. Effects of commercial dietary prebiotic and probiotic supplements on growth, innate immune responses, and intestinal microbiota and histology of Totoaba macdonaldi[J]. Aquaculture, 2018, 491: 239-251. DOI:10.1016/j.aquaculture.2018.03.031 |
| [39] |
Yamamoto F Y, Yin F, Rossi W Jr, et al. β-1, 3 glucan derived from Euglena gracilis and Algamune? enhances innate immune responses of red drum (Sciaenops ocellatus L[J]. Fish & Shellfish Immunology, 2018, 77: 273-279. |
| [40] |
Wei N, Wang C C, Xiao S J, et al. Intestinal microbiota in large yellow croaker, Larimichthys crocea, at different ages[J]. Journal of the World Aquaculture Society, 2018, 49(1): 256-267. DOI:10.1111/jwas.12463 |
| [41] |
Zhang C, Zheng X F, Ren X, et al. Bacterial diversity in gut of large yellow croaker Larimichthys crocea and black sea bream Sparus macrocephalus reared in an inshore net pen[J]. Fisheries Science, 2019, 85(6): 1027-1036. DOI:10.1007/s12562-019-01349-5 |
| [42] |
Guerreiro I, Serra C R, Oliva-Teles A, et al. Short communication: Gut microbiota of European sea bass (Dicentrarchus labrax) is modulated by short-chain fructooligosaccharides and xylooligosaccharides[J]. Aquaculture International, 2018, 26(1): 279-288. DOI:10.1007/s10499-017-0220-4 |
| [43] |
Xu N, Tan G C, Wang H Y, et al. Effect of biochar additions to soil on nitrogen leaching, microbial biomass and bacterial community structure[J]. European Journal of Soil Biology, 2016, 74: 1-8. DOI:10.1016/j.ejsobi.2016.02.004 |
| [44] |
Egerton S, Culloty S C, Whooley J, et al. The gut microbiota of marine fish[J]. Frontiers in Microbiology, 2018, 9: 873. DOI:10.3389/fmicb.2018.00873 |
| [45] |
Desai A R, Links M G, Collins S A, et al. Effects of plantbased diets on the distal gut microbiome of rainbow trout (Oncorhynchus mykiss)[J]. Aquaculture, 2012, 350-353: 134-142. DOI:10.1016/j.aquaculture.2012.04.005 |
| [46] |
Zheng X F, Tang J Y, Zhang C F, et al. Bacterial composition, abundance and diversity in fish polyculture and mussel-fish integrated cultured ponds in China[J]. Aquaculture Research, 2017, 48(7): 3950-3961. DOI:10.1111/are.13221 |
| [47] |
Leisner J J, Laursen B G, Prévost H, et al. Carnobacterium: positive and negative effects in the environment and in foods[J]. FEMS Microbiology Reviews, 2007, 31(5): 592-613. DOI:10.1111/j.1574-6976.2007.00080.x |
| [48] |
Ringø E, Zhou Z, Vecino J L G, et al. Effect of dietary components on the gut microbiota of aquatic animals[J]. Aquaculture Nutrition, 2016, 22(2): 219-282. |
| [49] |
Holben W E, Williams P, Saarinen M, et al. Phylogenetic analysis of intestinal microflora indicates a novel mycoplasma phylotype in farmed and wild salmon[J]. Microbial Ecology, 2002, 44(2): 175-185. DOI:10.1007/s00248-002-1011-6 |
| [50] |
Bano N, de Rae Smith A, Bennett W A, et al. Dominance of Mycoplasma in the guts of the Long-Jawed Mudsucker, Gillichthys mirabilis, from five California salt marshes[J]. Environmental Microbiology, 2007, 9(10): 2636-2641. DOI:10.1111/j.1462-2920.2007.01381.x |
 2020, Vol. 27
2020, Vol. 27

