真骨鱼类生活在复杂多变的水环境中, 为维持体内平衡, 并能更好地适应环境变化, 演化了多样有效的离子渗透压调节机制[1]。鱼类渗透调节过程主要是通过鳃、肾、肠道等器官的联合作用实现的。其中, 鳃是鱼类特有的重要的渗透调节器官, 不仅参与气体交换, 还参与渗透压调节、酸碱平衡调节以及含氮废物排放等多种功能[2]。有研究表明, 鱼类鳃丝表皮上含有大量的离子细胞(线粒体丰富细胞, mitochondrion-rich cells), 它们是鳃执行渗透压调节的重要功能单位, 离子细胞的调节功能是通过在细胞膜上大量的离子转运子和离子通道蛋白完成的[3-4]。扫描电镜观察发现, 鳃离子细胞有不同的亚型, 并装配有不同的离子转运系统[5-6]。如海水适应鱼类鳃离子细胞一般含有3种主要的离子转运子: Na+/K+-ATP酶(Na+/K+- ATPase, NKA)、Na+/K+/2Cl-协同转运子(Na+/K+/2Cl- cotransporter, NKCC)和氯离子通道蛋白(Cl- channel homologous to human cystic fibrosis transmembrane conductance regulator, CFTR)[7-8], 分别位于离子细胞的外、底、侧膜, 在海水适应过程中起协同作用[7, 9-11]。
目前, 鱼类盐度适应机制研究报道多集中在这些离子转运蛋白的活性、mRNA与蛋白表达[9, 12-13]。最近, 通过RNA测序技术, 探讨了鱼类在盐度适应过程中的能量分配与利用[14]。鉴于鱼类盐度适应机制是一个复杂的生理过程, 涉及大量蛋白质的联合作用, 因此, 蛋白质水平研究结果可能为鱼类渗透机理研究提供新的视角和全面认识。随着对模式动物蛋白质研究工作的深入, 蛋白质抗体芯片(protein chip)已成为蛋白质芯片中发展较为成熟的技术, 具有高通量、微型化、集成化、平行性检测等特点, 可用于检测某一特定的生理过程或者病理过程中相关蛋白质的表达谱, 该技术与质谱(MS)技术结合, 可作为重要生理活动差异表达蛋白质筛选与鉴定的高效工具[15-17]。为此, 在前期开展红罗非鱼在盐胁迫下生理生化、鳃离子细胞水平变化及几种离子转运蛋白mRNA表达变化的研究基础上[18], 本研究采用抗体芯片技术结合串联质谱(MS/MS)技术筛选盐胁迫下红罗非鱼鳃组织差异表达蛋白, 并对候选差异蛋白进行免疫组化和免疫印迹验证, 以期为广盐性鱼类盐度胁迫应答机制研究提供更多数据资料。
1 材料与方法 1.1 实验材料红罗非鱼(Oreochromis mossambicus♀ × O. niloticus♂)于2015年10月取自上海海洋大学罗非鱼种质资源试验站, 运回实验室后, 在室内控温循环水族箱中饲养, 待适应新环境后, 挑选体重(50.0±3.5) g规格均匀、健康活泼的个体进行盐度胁迫实验。
设置淡水组(0)、盐度组(10、20、30), 盐度组用水通过在淡水中加入相应量的海水晶配制。实验前一天停止喂食, 实验开始时将鱼从淡水中直接移至各盐度组中, 每组放鱼30尾, 各设3个重复。实验期间, 水温保持在(25.8±0.5)℃, 溶解氧(5.7±0.5) mg/L。胁迫后0 h、12 h、24 h、48 h、72 h、96 h分别采样, 每组随机选取3~5尾。
mAbPTM抗体芯片、抗体由艾比玛特生物医药(上海)有限公司提供, 纳升液电喷雾-四极杆-飞行时间串联质谱联用仪(Dionex ultimate3000- maxis impact UHR, Themofisher-Bruker; Nano-LC- qTOF-MS/MS)来自上海交通大学大型仪器共享平台质谱分析测试中心。防脱玻片、SABC三步法试剂盒购自武汉博士德公司, 蛋白提取、标准曲线及Western blot相关试剂购于Thermo, Western blot GAPDH、抗体稀释液购自上海威奥生物科技有限公司, 其他试剂则为国产分析纯。
1.2 实验方法 1.2.1 鳃组织蛋白提取无菌操作取淡水组、各盐度组鳃组织, 剪碎后于研钵中加液氮研磨, 按照0.1 g组织加200~400 μL RIPA buffer(50 mmol/L Na3PO4 pH8.0, 150 mmol/L NaCl, 0.1% SDS, 1.5% NP-40, 0.5% deoxycholate (脱氧胆酸), 2 mmol/L MgCl2)进行蛋白裂解。4℃下12000 r/min离心30 min, 收集上清, BCA法测定蛋白浓度, 稀释至10 mg/mL, –80℃保存。
1.2.2 芯片筛选取淡水组、30盐度组样品进行生物素biotin标记质控合格后, 用cy3-链霉亲和素(strepavidin-cy3)荧光染料进行标记, 分别与mAbPTM抗体芯片(每张芯片包含10368个不同的抗体点和432个对照点)进行杂交, 使用Genepix 4000荧光扫描仪检测, 根据平行杂交抗体芯片所得的荧光数据, 使用PDQuestTM 2-D Analysis Software 8.0软件分析淡水组与盐度组间的蛋白位点差异。
1.2.3 质谱与生物信息学分析蛋白样品与亲和素标记的磁珠在4℃孵育2 h, 去除与亲和素结合的蛋白, 取上清液进行免疫沉淀实验, 免疫沉淀方法参考贺莉等[19]的方法进行。取适量样品, 进行10%的SDS-聚丙烯酰胺凝胶电泳考马斯亮蓝染色。切取洗脱特异性的目的蛋白条带, 送至上海交通大学大型仪器共享平台质谱分析测试中心采用纳升液相色谱-四极杆飞行时间串联质谱法(Nano-LC-qTOF-MS/MS)进行二级串联质谱分析。利用data Analysis4.1软件和Mascot 4.0搜索引擎将所得数据与NCBI和SWISS数据库比对分析。
1.2.4 免疫组化分别取0、10、20、30盐度下胁迫96 h的罗非鱼第二鳃弓鳃片, 进行石蜡包埋, 切片厚度为5~8 μm。切片脱蜡后, 用3%去离子水室温孵育10 min以灭活内源性酶, 通过抗原修复液进行抗原修复, 滴加10%山羊血清封闭30 min。添加1 : 50稀释的一抗, 4℃孵育过夜, 添加二抗(兔抗鼠IgG)室温孵育30 min。添加辣根过氧化物酶标记的高灵敏度链霉亲和素(HSS- HRP)室温30 min, 水洗后二氨基联苯胺(DAB)显色5~ 10 min, 苏木精复染, 脱水、透明、封片。阴性对照组除不添加一抗外(以正常山羊血清代替), 其他步骤同上。
1.2.5 Western Blot取各盐度组蛋白样品, 将蛋白抽提液在SDS-PAGE(10%的浓缩胶和5%的分离胶)上分离后, 用半干转膜法(fast semi-dry blotter)将蛋白转至PVDF膜进行免疫杂交反应, 一抗稀释比例为1 : 500, 二抗为兔抗鼠IgG, 工作浓度为1 : 2000。用Supersignal West Pico发光试剂反应, Bio-Rad Chemi Doc XRS凝胶成像仪拍照, 并用Quantity One图像分析系统分析条带光密度。
1.3 数据处理与分析实验数据利用SPSS 22.0统计软件分析, 单因素方差分析进行显著性检验, Duncan多重比较检测各测量指标的差异, P < 0.05为差异显著。
2 结果与分析 2.1 蛋白抗体芯片筛选与免疫沉淀实验抗体芯片扫描结果, 与淡水对照组比较, 盐度组共检测到181个差异表达蛋白位点, 其中, 表达上调的142个, 表达下调39个, 差异最高倍数达到11.7倍(变化倍数≥1.5)。与碱度组芯片筛选结果(未发表数据)交叉比较, 得到55个只在盐度组特异表达的抗体点, 其中特异上调的40个, 特异下调的15个。
挑选5个特异表达的蛋白位点(抗体编号分别为241-B3、85-B7、92-B9、304-B8、263-B2)进行免疫沉淀实验, 经电泳分离染色, 其中, 获得了3个特异结合的蛋白条带(抗体编号为92-B9、304-B8、263-B2)。目的条带割胶送串联质谱分析(图 1)。
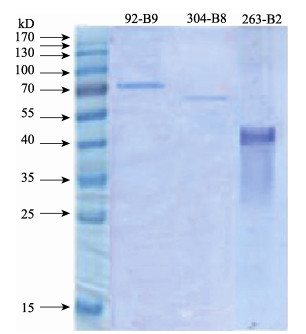
|
图 1 红罗非鱼鳃差异蛋白的免疫沉淀分析 Fig.1 Immunoprecipitation of differentially expressed proteins in gill of red tilapia (Oreochromis mossambicus♀ × O. niloticus♂) |
对3个显著差异表达蛋白点(抗体编号92-B9, 263-B2, 304-B8)进行质谱鉴定与生物信息学比对, 获得了3个蛋白(表 1):蛋白质二硫键异构酶A3(protein disulfide-isomerase A3, PDIA3)、中间丝蛋白(intermediate filament protein ON3-like, IFs)、转运蛋白63(translocation protein SEC63, SEC63), Mascot检索分值26~322分, 肽段匹配数13~39个, 序列覆盖率60%~84.6%。
|
|
表 1 红罗非鱼鳃差异蛋白质谱鉴定结果 Tab.1 Identification of differentially expressed proteins in red tilapia (Oreochromis mossambicus♀ × O. niloticus♂) gill by Nano-LC-qTOF-MS/MS |
在淡水和盐度组(10、20、30)红罗非鱼鳃组织中, PDIA3、IF免疫阳性细胞主要分布在鳃小片基部, 20盐度组较少, 30盐度组表达最多; SEC63未检测到阳性反应(图 2)。阴性对照组均未发现免疫阳性细胞。
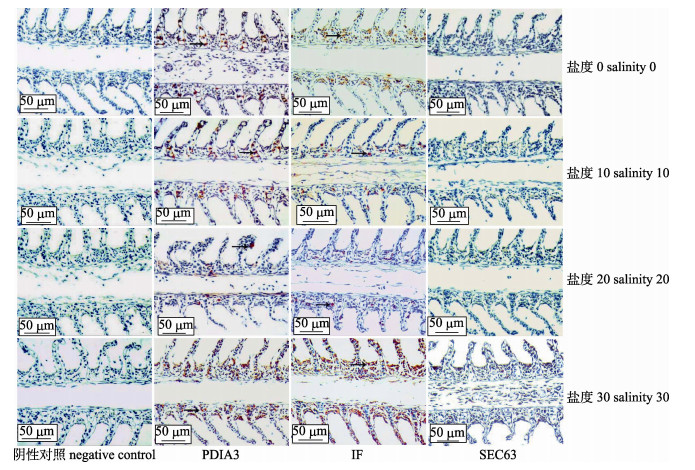
|
图 2 不同盐度胁迫下红罗非鱼鳃PDIA3、IF、SEC63蛋白免疫组化 箭头表示免疫反应阳性细胞. Fig.2 Immunohistochemistry of PDIA3, IF and SEC63 proteins in gill of red tilapia (Oreochromis mossambicus♀ × O. niloticus♂) under different salinities Arrows indicate positive cells. |
不同盐度对红罗非鱼鳃蛋白表达量影响如图 3所示。SEC63蛋白未检测到条带, IF、PDIA3蛋白均检测到清晰条带, 半定量分析表明, IF和PDIA3蛋白表达量随盐度增加均呈先降低、后升高的变化趋势, 其中, 30盐度组表达量最高, 20盐度组表达显著低于其他盐度组(P < 0.05)。
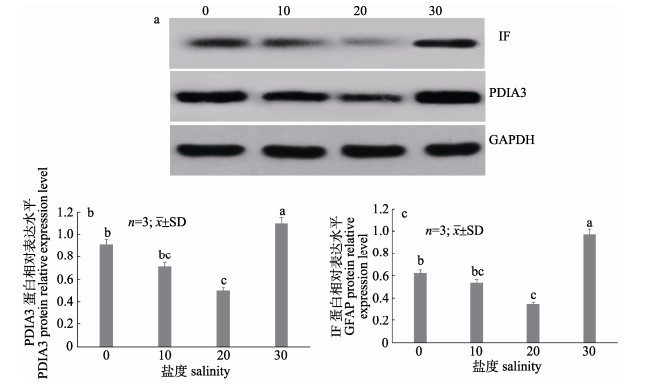
|
图 3 不同盐度下红罗非鱼鳃蛋白IF与PDIA3表达情况
a.不同盐度下鳃组织中两个蛋白的免疫印迹; b. IF蛋白表达情况; c. PDIA3蛋白表达情况. 图中字母相同表示差异不显著(P > 0.05), 不同字母表示差异显著(P < 0.05). Fig.3 The expression of IF and PDIA3 proteins in gill of red tilapia (Oreochromis mossambicus♀ × O. niloticus♂) under different salinities a. The western blot results of IF and PDIA3 in gill of red tilapia under different salinities; b. The expression of IF protein; c. The expression of PDIA3 protein. Bars with the same superscript letters are not significantly different (P > 0.05), while those with different superscript letters are significantly different (P < 0.05). |
在30盐度胁迫下不同时间点红罗非鳃IF、PDIA3蛋白表达量如图 4所示。随胁迫时间变化, IF表达量总体呈先降低、后升高的变化趋势, 在48 h达到最低值, 72 h开始有明显回升, 96 h趋于稳定; PDIA3表达量随时间变化显著升高, 48 h内变化不明显(P > 0.05), 在96 h升高且远高于0 h (P < 0.01)。
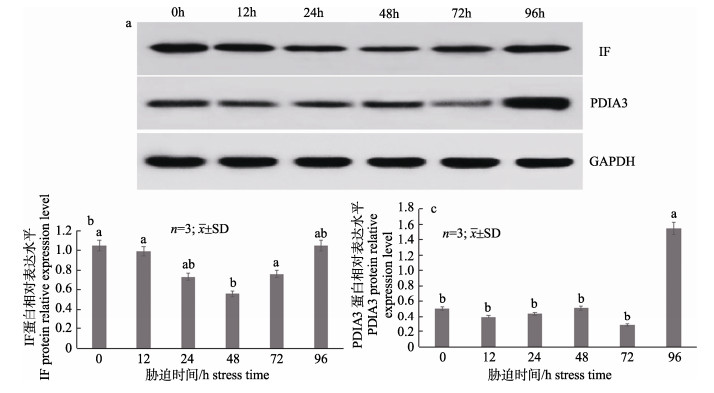
|
图 4 30盐度下不同时间点红罗非鱼鳃蛋白IF和PDIA3表达情况
a. 30盐度不同时间点鳃组织中两个蛋白的免疫印迹; b. IF蛋白表达情况; c. PDIA3蛋白表达情况. 图中字母相同表示差异不显著(P > 0.05), 不同字母表示差异显著(P < 0.05). Fig.4 Relative protein expression of IF and PDIA3 in gill of red tilapia (Oreochromis mossambicus♀ × O. niloticus♂) at different times under 30 salinity a. The western blot results of IF and PDIA3 in gill of red tilapia at different times under 30 salinity; b. The expression of IF protein; c. The expression of PDIA3 protein. Bars with the same superscript letters are not significantly different (P > 0.05), while those with different superscript letters are significantly different (P < 0.05). |
本研究采用蛋白抗体芯片结合质谱分析技术, 对盐胁迫下红罗非鱼鳃组织中差异表达蛋白进行筛选, 初步获得了3个候选差异蛋白:中间丝蛋白(IF)、转运蛋白(SEC63)和二硫化物异构酶A3(PDIA3), 有关这3个蛋白在鱼类盐度胁迫中的表现与功能研究, 之前尚未见有报道。通过免疫组化和免疫印迹技术对候选差异蛋白进行了初步验证, IF、PDIA3均呈现有明显的免疫组化和免疫印迹阳性反应, 与质谱结果相印证, IF、PDIA3蛋白表达水平在20盐度中最低, 淡水或30盐度下表达水平均有提高。这些经过筛选与盐胁迫相关的蛋白结果在国内外尚未见到报道, 猜测它们可能是与红罗非鱼鳃应对盐胁迫相关的应激蛋白。由于未检测到SEC63阳性免疫反应和明显目的条带, 该蛋白是否为盐度胁迫响应蛋白还需进一步研究。
细胞骨架(cytoskeleton)是真核细胞中由多种蛋白质聚合而成的三维纤维状网架体系。细胞骨架包括微丝、微管和中间丝。细胞骨架在细胞分裂、细胞生长、细胞物质运输、细胞壁合成等许多生命活动中都具有非常重要的作用。当生物处于不利环境中时, 外界的环境胁迫因子会通过对细胞结构产生损伤, 进一步影响到生物体组织器官的生理活动[20]。研究表明, 低温胁迫会引起大菱鲆(Scophthalmus maximus)表皮细胞骨架蛋白表达量下调, 破坏了表皮细胞的正常生理功能, 甚至引起表皮细胞凋亡[21]。李明云等[22]对低温胁迫下大黄鱼(Larimichthys crocea)肝脏蛋白质组进行了分析, 也发现细胞骨架蛋白——角蛋白18(CK- 18)表达水平呈现下调。本研究中, 红罗非鱼鳃中间丝蛋白(IF)随盐度胁迫强度增加呈现先降低、再升高的变化趋势, 20盐度时表达量最低。推测这可能与广盐性红罗非鱼的等渗点较高有关[23-25]。淡水、低盐度(10)下, 红罗非鱼处于低盐胁迫; 30盐度下, 红罗非鱼处于高盐胁迫, 两种非正常生理状态均会引起鳃表皮细胞的体积变化, 因此, 细胞骨架蛋白(中间丝蛋白IF)表达量增加可能与其维持细胞结构、发挥正常生理功能有关。在高盐度(30)胁迫过程中, 中间丝蛋白IF在胁迫48 h后表达量开始显著升高, 推测红罗非鱼耐受调节机制已启动, 以避免盐度胁迫对鳃表皮细胞造成损伤从而破坏红罗非鱼鳃正常的渗透调节生理功能。中间丝是最稳定的细胞骨架成分, 组成也较复杂, 可根据基因组结构、氨基酸序列的同源性和组织特异性表达模式分为5个不同家族: Ⅰ型(酸性)角蛋白, Ⅱ型(碱性)角蛋白, Ⅲ型结蛋白(desmin)、胶质纤维酸性蛋白(glial fibrillary acidic protein)、周边蛋白(peripherin)和波形蛋白(vimentin), Ⅳ型中间丝三联体(包括神经丝, 神经丝是广泛存在于动物成熟神经元中的一种中间丝蛋白), V型核纤层蛋白(A、B、C), 本研究中通过质谱得到的中间丝蛋白类型还有待进一步确定。
内质网是真核细胞重要的细胞器, 常与细胞核外膜连在一起, 按其表面有无核糖体分为光面内质网和糙面内质网, 是细胞内蛋白质合成的重要场所。当细胞受损时, 粗面内质网内的蛋白质合成会下降或消失; 当损伤恢复时, 其蛋白质合成也随之恢复。二硫化物异构酶A3(PDIA3)是内质网中一种重要的蛋白质折叠催化剂, 通过内质网驻留信号驻留在内质网中, 在新生肽的合成、加工和运输、成熟多肽的正确折叠, 以及逆境胁迫下受损蛋白的修复和重折叠等生理过程中起重要作用[26-27]。研究表明, 缺氧胁迫可能会引起中国明对虾(Fenneropenaeus chinensis)肝胰脏细胞内蛋白未折叠反应(UPR)被抑制或蛋白质合成抑制, 从而引起PDI表达量下降[28]。本研究中, 红罗非鱼鳃组织中PDIA3蛋白表达量随盐度升高呈现先降低、后升高的变化趋势, 在20盐度下表达量最低, PDIA3蛋白表达量随盐度的变化趋势与中间丝蛋白也基本一致。推测在低盐、高盐胁迫下, 细胞内离子浓度改变可能引起内质网中部分蛋白质在合成、加工与折叠过程中受损, PDIA3表达量增加有助于这些受损蛋白质的修复, 继而维持细胞的正常生理功能。在30盐度胁迫过程中, PDIA3在胁迫96 h表达量上升最为显著。
本研究表明, 抗体芯片-串联质谱法是盐胁迫下红罗非鱼鳃差异表达蛋白筛选和鉴定的一种有效方法, 初步获得了2个耐盐胁迫相关候选蛋白。在此基础上, 进一步开展其他差异表达蛋白的质谱鉴定工作, 可望从蛋白质水平为罗非鱼耐盐生理机制研究提供更多基础资料。
| [1] |
Hwang P P, Lee T H. New insights into fish ion regulation and mitochondrion-rich cells[J]. Comparative Biochemistry and Physiology Part A: Molecular & Integrative Physiology, 2007, 148(3): 479-497. |
| [2] |
Tang C H, Hwang L Y, Lee T H. Chloride channel ClC-3 in gills of the euryhaline teleost, Tetraodon nigroviridis: expression, localization and the possible role of chloride absorption[J]. Journal of Experimental Biology, 2010, 213(5): 683-693. DOI:10.1242/jeb.040212 |
| [3] |
Kültz D, Bastrop R, Jürss K, et al. Mitochondria-rich (MR) cells and the activities of the Na+/K+-ATPase and carbonic anhydrase in the gill and opercular epithelium of Oreochromis mossambicus adapted to various salinities[J]. Comparative Biochemistry and Physiology Part B: Comparative Biochemistry, 1992, 102(2): 293-301. |
| [4] |
Hiroi J, McCormick S D. New insights into gill ionocyte and ion transporter function in euryhaline and diadromous fish[J]. Respiratory Physiology & Neurobiology, 2012, 184(3): 257-268. |
| [5] |
Chang I C, Lee T H, Yang C H, et al. Morphology and function of gill mitochondria-rich cells in fish acclimated to different environments[J]. Physiological & Biochemical Zoology, 2001, 74(1): 111-119. |
| [6] |
Evans D H, Piermarini P M, Choe K P. The multifunctional fish gill: dominant site of gas exchange, osmoregulation, acid-base regulation, and excretion of nitrogenous waste[J]. Physiological Reviews, 2005, 85(1): 97-177. DOI:10.1152/physrev.00050.2003 |
| [7] |
Hiroi J, Mc Cormick S D, Ohtani-Kaneko R, et al. Functional classification of mitochondrion-rich cells in euryhaline Mozambique tilapia (Oreochromis mossambicus) embryos, by means of triple immunofluorescence staining for Na+/K+-ATPase, Na+/K+/2Cl- cotransporter and CFTR anion channel[J]. Journal of Experimental Biology, 2005, 208(Pt 11): 2023-2036. |
| [8] |
Marshall W S. Mechanosensitive signalling in fish gill and other ion transporting epithelia[J]. Acta Physiologica, 2011, 202(3): 487-499. DOI:10.1111/apha.2011.202.issue-3 |
| [9] |
Singer T D, Tucker S J, Marshall W S, et al. A divergent CFTR homologue: highly regulated salt transport in the euryhaline teleost F. heteroclitus[J]. American Journal of Physiology-Cell Physiology, 1998, 274(3 Pt 1): C715-723. |
| [10] |
Wu Y C, Lin L Y, Lee T H. Na+, K+, 2Cl- cotransporter: a novel marker for identifying freshwater- and seawater-type mitochondria-rich cells in gills of the euryhaline tilapia, Oreochromis mossambicus[J]. Zoological Studies, 2003, 42(1): 186-192. |
| [11] |
Evans T G, Somero G N. A microarray-based transcriptomic time-course of hyper- and hypo-osmotic stress signaling events in the euryhaline fish Gillichthys mirabilis: osmosensors to effectors[J]. Journal of Experimental Biology, 2008, 211(Pt 22): 3636-3649. |
| [12] |
Lin C H, Huang C L, Yang C H, et al. Time-course changes in the expression of Na, K-ATPase and the morphometry of mitochondrion-rich cells in gills of euryhaline tilapia (Oreochromis mossambicus) during freshwater acclimation[J]. Journal of Experimental Zoology Part A: Comparative Experimental Biology, 2004, 301(1): 85-96. |
| [13] |
Scott G R, Richards J G, Forbush B, et al. Changes in gene expression in gills of the euryhaline killifish Fundulus heteroclitus after abrupt salinity transfer[J]. American Journal of Physiology-Cell Physiology, 2004, 287(2): C300-C309. DOI:10.1152/ajpcell.00054.2004 |
| [14] |
Xu Z L, Gan L, Li T Y, et al. Transcriptome profiling and molecular pathway analysis of genes in association with salinity adaptation in Nile tilapia Oreochromis niloticus[J]. PLoS ONE, 2015, 10(8): e0136506. DOI:10.1371/journal.pone.0136506 |
| [15] |
Zhang C X, Liu H P, Tang Z M, et al. Cell detection based on protein array using modified glass slides[J]. Electrophoresis, 2003, 24(18): 3279-3283. DOI:10.1002/(ISSN)1522-2683 |
| [16] |
Zhu J, Zhu Y M, Han J X. Application of protein chip technology in the proteomics research[J]. International Journal of Laboratory Medicine, 2004, 25(3): 223-225. [胡洁, 朱有名, 韩金祥. 蛋白质芯片技术在蛋白质组研究领域的应用[J]. 国际检验医学杂志, 2004, 25(3): 223-225. DOI:10.3969/j.issn.1673-4130.2004.03.012] |
| [17] |
Zhu D, Zhang C H, Huang H X, et al. Effect of Jinhuang Fuzheng powder on cytokines of immunosuppressive mice with protein antibody micro-array[J]. China Journal of Chinese Materia, 2012, 37(23): 3624-3627. [朱丹, 张春花, 黄慧学, 等. 蛋白质抗体芯片技术检测金黄扶正散对免疫抑制小鼠细胞因子的影响[J]. 中国中药杂志, 2012, 37(23): 3624-3627.] |
| [18] |
Thammaratsuntorn J. Biochemical, cellular and molecular responses of red tilapia (Oreochromis niloticus ♀ × O. mossambicus♂) to salinity and alkalinity stresses[D]. Shanghai: Shanghai Ocean University, 2015: 20-54.
|
| [19] |
He L, Wu K C, Hui X L, et al. Screening of receptors of the peptide CGNSNPKSC that bind specifilcally to tumor vascular endothelium by immunoprecipitation and mass spectromethy[J]. Journal of Modern Oncology, 2008, 16(8): 1259-1263. [贺莉, 吴开春, 惠晓莉, 等. 应用免疫沉淀-质谱法筛选肿瘤血管靶向肽CGNSNPKSC的结合受体[J]. 现代肿瘤医学, 2008, 16(8): 1259-1263. DOI:10.3969/j.issn.1672-4992.2008.08.001] |
| [20] |
Zhai Z H. Cell Biology[M]. 4th Edition. Beijing: Higher Education Press, 2011. [翟中和. 细胞生物学[M]. 第四版.北京: 高等教育出版社, 2011.]
|
| [21] |
Xue B G. Research of hereditary feature and serum biochemical indexes of character related to cold-temperature tolerance of the turbot[D]. Shanghai: Shanghai Ocean University, 2011: 46-63. [薛宝贵.大菱鲆耐低温性状相关表型遗传特征及生理生化指标研究[D].上海: 上海海洋大学, 2011: 46-63.]
|
| [22] |
Li M Y, Ji D W, Wu H Q, et al. 2-DE Analysis in liver of Pseudosciaena crocea during low temperature stress[J]. Journal of Agricultural Biotechnology, 2010, 18(2): 323-328. [李明云, 冀德伟, 吴海庆, 等. 低温胁迫下大黄鱼肝脏蛋白质组双向电泳分析[J]. 农业生物技术学报, 2010, 18(2): 323-328. DOI:10.3969/j.issn.1674-7968.2010.02.020] |
| [23] |
Liao I C, Chang S L. Studies on the feasibility of red tilapia culture in saline water[C]//Proceedings, International Sympos ium on Tilapia in Aquaculture Nazareth, Israel: Tel Aviv University, 1983: 524-533.
|
| [24] |
Farmer G J, Beamish F W H. Oxygen consumption of Tilapia nilotica in relation to swimming speed and salinity[J]. Journal of the Fisheries Research Board of Canada, 1969, 26(11): 2807-2821. DOI:10.1139/f69-277 |
| [25] |
Martinez-Palacios C A, Ross L G, Rosado-Vallado M. The effects of salinity on the survival and growth of juvenile Cichlasoma urophthalmus[J]. Aquaculture, 1990, 91(1-2): 65-75. DOI:10.1016/0044-8486(90)90177-O |
| [26] |
Kaufmann R, Kirsch D, Spengler B. Sequenching of peptides in a time-of-flight mass spectrometer: evaluation of postsource decay following matrix-assisted laser desorption ionisation (MALDI)[J]. International Journal of Mass Spectrometry and Ion Processes, 1994, 131: 355-385. DOI:10.1016/0168-1176(93)03876-N |
| [27] |
Noiva R, Lennarz W J. Protein disulfide isomerase. A multifunctional protein resident in the lumen of the endoplasmic reticulum[J]. Journal of Biological Chemistry, 1992, 267(6): 3553-3556. |
| [28] |
Jiang H. Copmarative proteomic profiles of the hepatopancreas in Fenneropenaeus chinensis response to stresses[D]. Qingdao: The Institute of Oceanology, Chinese Academy of Sciences, 2009: 58-106. [蒋昊.中国明对虾在胁迫条件下肝胰脏的差异蛋白质组学研究[D].青岛: 中国科学院海洋研究所, 2009: 58-106.]
|
 2018, Vol. 25
2018, Vol. 25

