2. 海洋生物科学国际联合研究中心, 上海海洋大学, 上海 201306;
3. 水产科学国家级实验教学示范中心, 上海海洋大学, 上海 201306
2. International Research Center for Marine Biosciences Conferred by Ministry of Science and Technology, Shanghai Ocean University, Shanghai 201306, China;
3. National Demonstration Center for the Experimental Teaching of Fisheries Science, Shanghai Ocean University, Shanghai 201306, China
缺刻缘绿藻(Myrmecia incisa Reisigl)是一种单细胞微藻, 隶属绿藻门(Chlorophyta)、共球藻纲(Trebouxiophyceae)[1]。它的细胞呈球形或卵圆形, 常聚集在一起形成类似非定型群体的细胞团。该藻的显著特征是能积累花生四烯酸(arachidonic acid, ArA), 尤其在氮饥饿或磷饥饿等胁迫条件下, ArA最高含量可达到藻体干重的7%[2], 是目前已知ArA含量较高的藻株, 具有潜在的开发利用价值。在利用薄层层析(thin layer chromatography TLC)技术分离获得该藻的三酰甘油(triacylglycerol, TAG)后, 欧阳珑玲等[3]再利用气相色谱-质谱(gas chromatography-mass spectrometer, GC-MS)联用技术分析TAG的脂肪酸组成, 发现ArA占其总脂肪酸含量的68.0%;若在氮饥饿4 d后, 该含量可达75.6%。该数据非常接近桐油酸(D9, 11t, 13t-十八碳三烯酸)和蓖麻油酸(12-羟基-D9-十八碳一烯酸)等特殊脂肪酸分别在油桐(Vernicia fordii)、蓖麻(Ricinus communis)等种子TAG的百分含量(分别为85%和89%)[4-5]。
鉴于脂肪酸去饱和作用等酰基修饰过程主要发生在磷脂酰胆碱(也称卵磷脂, phosphatidylcholine, PC)、磷脂酰乙醇胺(也称脑磷脂, phosphatidylet ha nolamine, PE)等膜磷脂甘油骨架的sn-2碳位上[6-7], 而长链多不饱和脂肪酸(polyunsaturated fatty acid, PUFA)的延长主要发生在内质网的酰基-CoA池(acyl-CoA pool)中[8], 这样以PC和PE等甘油磷脂为中心, 就必然存在着去酰基/酰基化的反应过程, 即Lands循环[9]。在该循环过程中起着重要作用的酶如溶血磷脂酰乙醇胺酰基转移酶(lysophosp hatidylethanolamine acyltransferase, LPEAT)[10], 通过对溶血磷脂酰乙醇胺(lysophosphatidy leth an o lamine, LPE)进行酰基化作用, 使得酰基-CoA池中的长链多不饱和脂肪酰基有机会结合到LPE甘油骨架上的sn-2碳位上, 这样, PE就携带有长链PUFA[8]。这些含有特殊脂肪酸的PE, 再沿着不依赖酰基-CoA的TAG生物合成途径, 在磷脂:二酰甘油酰基转移酶(phospholipid:DAG acyltransferase, PDAT)的作用下, 将所携带的长链PUFA转到DAG上, 从而产生带有长链PUFA等特殊脂肪酸的TAG[11-14]。
自上述的分析可知, LPEAT在膜脂脂肪酸组成的不对称性和多样性以及TAG中长链PUFA等特殊脂肪酸的引入起着非常重要的作用。目前, 有关LPEAT的报道, 多来自对人类等哺乳动物及无脊椎动物的研究[15]。在植物中, 也只对拟南芥(Arabidopsis thaliana)的LPEAT进行了探讨[16-17]; 有关藻类LPEAT及其对藻类油脂合成代谢影响的研究至今未见报道。为此, 本研究在缺刻缘绿藻转录组[18]文库数据基础上, 通过NCBI(http://www.ncbi.nlm.nih.gov/)的BLASTx比对, 发现contig8647与胶球藻(Coccomyxa subel lipsoidea) C-169的溶血磷脂酸酰基转移酶(lysophosphatidic acid acyltransferase, LPAAT)序列(GenBank登录号: XP_005646810)有57%的相似度, 根据这段序列设计引物, 利用cDNA末端快速扩增技术(rapid amplification of cDNA ends, RACE)得到其cDNA全长序列。通过NCBI的BLASTp比对发现, 该基因编码的氨基酸序列与其他物种的LPEAT序列相似度高, 并与LPEAT聚类在一起, 故在此本研究将其命名为MiLPEAT。鉴于LPEAT主要是在膜磷脂LPE上进行酰基化作用, 在了解氮饥饿条件下, 缺刻缘绿藻的膜磷脂可以转变成贮藏的中性脂TAG的基础上[3], 本研究分析了该藻在氮饥饿4 d前后MiLPEAT的基因转录量以及LPE与PE相对含量的变化, 为了解缺刻缘绿藻脂类合成特性, 尤其为氮饥饿胁迫条件下该藻含丰富ArA的TAG积累机制提供理论依据。
1 材料与方法 1.1 藻种及培养缺刻缘绿藻H4301由暨南大学张成武教授馈赠, 来源于布拉格查理斯大学藻类收藏中心(culture collection of algae of Charles University of Prague)。将藻接种于BG-11液体培养基[19]中, 在25℃和115 µmol quanta/(m2·s)的光照培养箱中培养, 光周期为光照:黑暗=14 h : 10 h[2], 每天不定期地摇晃。待长至指数生长期, 4℃下以5500 r/min离心10 min, 收集藻细胞, 并用灭菌去离子水洗涤3次后再次同样离心收集藻细胞, 立即提取RNA或用液氮速冻, -80℃保存备用。
用于实时荧光定量PCR (quantitative real-time PCR, q-RT-PCR)实验的藻细胞先接种于内径为3.5 cm的圆柱体玻璃管(容量约500 mL)中, 利用Philips荧光灯单侧提供24 h连续光照并使用过滤空气充气培养。先用BG-11培养基在115 µmol quanta/(m2·s)条件下培养14 d, 5500 r/min离心收集藻体, 用灭菌去离子水洗涤3次并同样离心收集, 然后将其转入到新鲜的或缺氮的BG-11培养基中, 同样条件培养。在接种后的0 h、4 h、8 h、12 h、16 h、20 h、24 h、32 h、40 h、48 h、56 h、64 h、72 h、80 h、88 h、96 h分别取样, 同样离心、洗涤并收集藻细胞, 液氮速冻后-80℃保存备用。每组设置3个平行样品。
用于氮饥饿胁迫下脂类含量变化分析的藻细胞培养方式同q-RT-PCR实验, 为保证数据统计分析的可靠性, 每组设8个平行, 在接种后的0 d和4 d分别取样, 离心收集, 液氮速冻后用冷冻干燥机将藻细胞冻干。
1.2 缺刻缘绿藻总RNA的提取及LPEAT基因cDNA全长序列的克隆参照TRIzol试剂(Invitrogen公司)的说明书抽提缺刻缘绿藻的总RNA。按照SMART RACE cDNA扩增反应试剂盒(Clontech公司)的使用说明, 分别反转录合成5′-和3′-RACE技术所需的cDNA第一链, 作为基因克隆的PCR反应模板。
根据缺刻缘绿藻转录组数据库[18]中筛选得到的contig8647序列利用Primer3web (http://bioinfo.ut.ee/primer3/)网站在线设计3′-及5′-RACE基因特异性引物及巢式引物各两对: 5′RACE1(F/R)和3′RACE1(F/R)(表 1), 由上海生工生物工程有限公司合成。
|
|
表 1 缺刻缘绿藻LPEAT基因克隆及q-RT-PCR所用引物 Tab.1 Primers employed for gene cloning and q-RT-PCR of LPEAT from Myrmecia incisa |
利用设计的巢式引物通过RACE技术获得cDNA的5′-和3′-末端序列。25 µL LPEAT 5′- RACE的第一轮PCR反应体系: PCR级水16.75 mL, 10×Advantage 2 PCR缓冲液2.5 mL, dNTPs 2 mL, 5′RACE1 (R) (表 1)0.5 mL, 10×UPM 2.5 mL, 5′-RACE cDNA 1 mL, 聚合酶混合液0.25 mL。5′-RACE第二轮巢式PCR反应体系基本同第一轮, 只是引物为NUP和5′RACE1 (F)(表 1), 模板为第一轮PCR产物。反应程序为: 94℃变性5 min; 94℃变性30 s, 70℃退火30 s, 72℃延伸2 min, 25个循环; 72℃延伸10 min。
25 µL LPEAT 3′-RACE反应体系基本上同5′-RACE。在第一轮PCR反应体系中将引物及模板分别改成3′RACE1 (F)(表 1)及3′-RACE cDNA; 在第二轮巢式PCR反应体系中, 将引物及模板分别改成3′RACE1 (R)(表 1)及第一轮PCR产物。反应程序也基本上同5′-RACE, 但退火温度分别为65℃(第一轮)和67℃(第二轮巢式)。根据LPEAT基因RACE扩增结果及拼接后得到的cDNA全长, 分别设计上下游引物1cDNA (F)和1cDNA (R)(表 1)进行序列验证。
利用胶回收试剂盒(艾德来生物科技有限公司, 北京)回收纯化上述PCR所扩增的目的片断, 并将它们与pMD® T-19载体(TaKaRa公司, 大连)连接后转入大肠杆菌(Escherichia coli)DH5a感受态细胞(天根生化科技有限公司, 北京), 筛选阳性克隆并经过菌落PCR验证后送上海生工生物工程有限公司进行序列分析。
1.3 缺刻缘绿藻DNA的提取及LPEAT基因DNA全长序列的克隆DNA提取采用十六烷基三甲基溴化铵(cetyltrimethyl ammonium bromide, CTAB)法[20]。
基于所得MiLPEAT的cDNA全长序列分段设计了3对引物DNA1 (F/R)、DNA2 (F/R)、DNA3 (F/R)(表 1), 以缺刻缘绿藻基因组DNA为模板进行PCR扩增。25 μL反应体系含有PCR级水9.5 mL、上下游引物各1 mL、基因组DNA模板1 mL及PCR Master Mix 12.5 mL。反应程序: 94℃预变性3 min; 30个循环为94℃变性45 s, 在表 1所列相应引物的退火温度退火45 s, 72℃延伸2 min; 最后72℃延伸10 min。
按上述cDNA克隆方法进行目的片断回收、连接、转化大肠杆菌、阳性克隆筛选和测序。拼接后得到MiLPEAT的DNA全长序列, 并利用NCBI的SPIDEY程序(http://www.ncbi.nlm.nih.gov/spidey/spideyweb.cgi)比对其cDNA全长序列以了解内含子位置。
1.4 缺刻缘绿藻LPEAT基因的生物信息学分析MiLPEAT的cDNA全长序列经过NCBI的ORF Finder预测得到其开放阅读框(ORF), 并利用Primer Premier 5将其翻译成蛋白质序列, 然后再运用相应的生物学网络服务器(表 2)分别对MiLPEAT所编码蛋白的等电点、分子量、跨膜区、疏水性、信号肽、转运肽、结构域、功能位点、二级结构等进行预测。
|
|
表 2 序列分析所用网络服务器 Tab.2 Web services applied in the sequence analysis |
利用Clustal_X软件[21]将缺刻缘绿藻与其他植物的LPEAT氨基酸序列进行比对, 并利用BioEdit软件[22]进行编辑。从NCBI的GenBank中下载植物等相关物种的LPEAT, 并以3个甘油-3-磷酸酰基转移酶(glycerol-3-phosphate acyltransferase, GPAT)9氨基酸序列作为外群, 使用MEGA6软件[23]的MUSCLE程序先进行比对, 然后构建邻接(Neighbor-Joining, NJ)聚类图。
1.5 缺刻缘绿藻LPEAT基因在氮饥饿胁迫下转录量分析q-RT-PCR使用的引物1 (F)和1 (R)(表 1)按照SYBR® Premix Ex Taq™ (Tli RNaseH Plus)试剂盒说明书(TaKaRa公司, 大连)中的引物设计原理设计, 并使用BLAST检索确认引物的特异性。选择本实验室获得的缺刻缘绿藻β-actin基因(GenBank登录号: FJ548973)[24]作为内参基因, 设计的引物act (F)和act (R)见表 1。
将提取的缺刻缘绿藻总RNA使用PrimeScript® RT reagent Kit With gDNA Eraser (Perfect Real Time)(TaKaRa公司, 大连)进行反转录合成cDNA第一链。先进行基因组DNA去除反应, 该反应体系包含gDNA Eraser 1 µL、总RNA 1 µg及5×gDNA Eraser Buffer 2 µL, 加无RNase的dH2O至10 µL。在PCR仪中于42℃孵育2 min。然后依次加入4xRT Primer Mix 1 µL、PrimeScript® RT酶混合物Ⅰ 1 µL及5×PrimeScript®缓冲液2 4 µL并补加无RNase的dH2O至20 µL。在PCR仪中于37℃孵育15 min、85℃孵育5 s, 从而反转录得到cDNA第一链。
利用q-RT-PCR方法在7500 PCR仪(Bio-Rad公司, USA)中对MiLPEAT的基因转录水平进行检测。反应体系包括Dye Ⅱ 0.4 µL、正/反向引物各1 µL、cDNA第一链模板2 µL及2×SYBR Primix Ex TaqTM Ⅱ 10 µL, 并加无RNase的dH2O至20 µL。反应程序: 95℃预变性30 s; 然后40个循环包括95℃变性5 s, 60℃退火34 s; 最后95℃变性15 s, 60℃退火1 min, 95℃变性15 s。
对所有样品的检测均进行3次重复。收集数据并使用2−∆∆CT法进行分析[25], 结果为3个样品的平均值±标准差(x±SD), 运用t检验进行显著性分析。
1.6 缺刻缘绿藻LPE及PE在氮饥饿胁迫前后相对含量的变化分析缺刻缘绿藻总脂的提取参考Bligh等[26]的方法并稍作改良。50 mg冻干藻粉中加入3 mL经75℃预热的异丙醇处理后, 加入1 mL氯仿、0.6 mL蒸馏水和1 mL玻璃珠涡旋振荡, 并收集有机相; 残渣用5 mL含0.01%的2, 6-二叔丁基-4-甲基苯酚(butylated hydroxytoluene, BHT)的氯仿/甲醇/水(1 : 2 : 0.8, 体积比)溶液重悬, 涡旋振荡15 min后离心收集有机相, 重复1次; 合并收集的有机相并加入1/2体积灭菌超纯水充分混匀后, 离心收集下层有机相。所得总脂用CNWBOND Carbon-GCB固相萃取柱(CNW公司, 德国)以去除其中的叶绿素。
将总脂通过液相色谱-质谱(liquid chro mat o graphy-mass spectrometer, LC-MS)联用技术进行分析, LC-MS条件参照Liu等[14]。原始数据经Thermo SIEVE 2.1 Qualitative Analysis Software (Thermo Fisher Scientific, USA)处理后, 就获得了包括保留时间(retention time)、质荷比(mass to charge)、观察量(样本)和峰强值(归一化处理)的二维数据矩阵。基于上述信息, 利用Thermo自带软件Lipid Search得到脂类化合物结构信息。氮饥饿胁迫4 d下, 缺刻缘绿藻细胞中PE和LPE的变化以相对丰度(%)表示, 即氮饥饿胁迫下藻细胞PE或LPE峰强值与正常培养细胞中PE或LPE峰强值的百分比[27-29]。结果为8个样品的平均值±标准差(x±SD), 运用t检验进行显著性分析。
2 结果与分析 2.1 缺刻缘绿藻LPEAT基因的cDNA及DNA全长序列的克隆首先根据缺刻缘绿藻转录组数据库[18]中长252 bp的contig8647序列设计一对引物8647 (F)和8647 (R)(表 1)验证这个contig序列, 并根据这个contig的验证序列设计引物5′RACE1 (R/F)以及3′RACE1 (R/F)(表 1), 利用RACE技术扩增该基因的5′-及3′-末端序列, 第二轮巢式PCR的扩增结果如图 1所示。3′-末端的序列分析结果表明, 所得目的片段大小为705 bp, 并含有真核生物核编码基因特征性的poly A尾巴; 5′-末端序列的测序结果表明目的片段的大小为511 bp。
将所得的5′-及3′-末端序列与验证的contig8647序列进行拼接, 然后根据拼接的序列重新设计一对引物1cDNA (R/F)(表 1)并进行PCR扩增反应以验证该序列。测序结果显示, 该基因的全长cDNA序列长为1 303 bp; 经ORF finder预测可知, 它的ORF序列长为981 bp, 编码326个氨基酸残基; 终止密码子为TGA; 5′-非翻译区(UTR)长为129 bp, 3′-UTR序列长为193 bp。通过NCBI的BLASTp比对(结果未展示)发现该基因所编码蛋白属于溶血磷脂酰基转移酶(lysophospholipid acyltransferase, LPLAT)超级家族成员, 且与众多物种的LPEAT具有40%以上的同源性。如与小米(Setaria italica)的LPEAT(XP_004962349.1)有43%的同源性, 与拟南芥中已进行功能鉴定的LPEAT1(NP_ 001321299.1)有39%的同源性。值得一提的是, BLASTp结果虽然显示其与胶球藻C-169的GPAT (XP_005646810.1)具有49%的同源性, 但后者的功能并未经过证实, 且通过聚类分析(具体见下文)指明我们得到的序列并不与GPAT聚类在一起, 所以本研究将自缺刻缘绿藻克隆的该基因命名为MiLPEAT, GenBank中的登录号为MG558460。
根据MiLPEAT的cDNA序列设计3对引物DNA1 (F/R)、DNA2 (F/R)、DNA3 (F/R)(表 1), 以缺刻缘绿藻基因组DNA为模板进行PCR扩增, 得到长度为222 bp、1533 bp以及200 bp的目的片段(图 1)。将得到的结果再进行拼接并验证, 最终获得MiLPEAT的全长DNA序列, 它长1871 bp。利用SPIDEY软件将MiLPEAT的cDNA及DNA序列进行比对, 发现MiLPEAT含有6个内含子, 均位于编码区, 长度自5′-末端起依次为80 bp、84 bp、84 bp、82 bp、138 bp和106 bp; 这些序列均符合“GT-AG”剪接规则, 即每个内含子的5′-端开始的两个核苷酸都是GT, 3′-端末尾的两个核苷酸都是AG; 它们将ORF分割成7个外显子(图 1)。
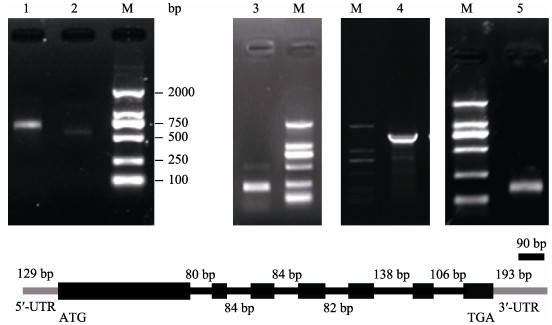
|
图 1 缺刻缘绿藻LPEAT基因cDNA及DNA全长扩增产物琼脂糖凝胶电泳图(上)及基因结构图(下) M: DL2000 DNA分子量标准品; 泳道1: 3′-RACE第二轮巢式引物PCR扩增产物; 泳道2: 5′-RACE第二轮巢式引物PCR扩增产物; 泳道3: DNA1(F/R)引物PCR扩增产物; 泳道4: DNA2(F/R)引物PCR扩增产物; 泳道5: DNA3(F/R)引物PCR扩增产物.基因结构图(下图)中两端灰色直线分别代表 5′-和3′-UTR; 黑色矩形框代表外显子; 黑色直线代表内含子. Fig.1 Agarose gel electrophoretogram of PCR products for full-length cDNA and DNA cloning of MiLPEAT (upper panel) and its gene structure M: DL2000 DNA standard marker; Lane 1: Products of the second 3′-RACE including two rounds of PCR reaction; Lane 2: Products of the second 5′-RACE including two rounds of PCR reaction; Lane 3: PCR products of DNA1 (F/R) primers; Lane 4: PCR products of DNA2 (F/R) primers; Lane 5: PCR products of DNA3 (F/R) primers; the grey lines represent 5′-/3′-UTR; the black boxes represent exons; and the black lines represent introns. |
经ComputepI/MW网络服务器(表 2)预测可知, MiLPEAT所编码蛋白的分子量为36.2 kD, 等电点为9.3。通过SMART搜索工具并利用InterProscan Sequence Search程序(表 2)分析表明, MiLPEAT含有一个磷酸酰基转移酶结构域PlsC, 位于其氨基酸序列的I119~G234之间; 还有一个含LPLAT_ LPCAT1-like结构域蛋白, 位于A86~R308之间, 以及酰基转移酶结构域, 位于P112~K232之间。通过序列比对(图 2)发现, 相对比较保守的氨基酸也多位于这个区域。在该区域中存在4个模体(motif), 即NH(x)4D、GCxYVxR、FPEGT及PVxPVx, 这与Lewin等[30]对LPAAT、LPEAT等4种酰基转移酶氨基酸模体的总结结果一致。其中“NH(x)4D”模体Ⅰ(图 2), 正是所有磷脂酰基转移酶的功能结构域, 包含活性位点部分, 表明MiLPEAT应是一种磷脂酰基转移酶。

|
图 2 植物LPEAT多重同源序列比对 在LPEAT1和LPEAT2中完全保守的氨基酸用黑色阴影标出; 灰色阴影指示分别在LPEAT1或LPEAT2中完全保守的氨基酸; 氨基酸上的短横线标示酰基转移酶的4个模体; 星号指示的残基在酶的催化过程中起作用; 三角形指示的残基与底物结合有关; 羧基端的双赖氨酸模体用方框标明. Fig.2 Amino acid sequence alignment of LPEATs from different species of plants High similarity amino acid residues and KK motif are shaded black and grey, respectively. Four conserved acyltransferase motifs are indicted above the alignment (motif Ⅰ: NH(x)4D; motif Ⅱ: GCxYVxR; motif Ⅲ: FPEGT; motif Ⅳ: PVxPVx). Asterisks denote the residues that play a role in LPEAT catalysis. Triangles denote the residues that are important in binding the substrate LPE. The carboxyl-terminal dilysine motif is indicated in a box. |
MiLPEAT二级结构预测结果表明, 该蛋白质可能含有9个α-螺旋区。在MiLPEAT二级结构中, 最长的两个α-螺旋区域分别由35个和21个氨基酸残基组成, 相应地位于V31~L65及Q73~I93之间。由于它们均长于20个氨基酸, 因此理论上是能够跨膜的。
Phobius和TMHMM Server v. 2.0的跨膜区预测结果也表明, 在L37~C63的区域形成一次跨膜; TMpred则预测在I93~Q109区域又进行一次跨膜。而结合疏水性分析结果, 本研究发现L37~C63区域的疏水性很高; 经Predict Protein预测, 可知MiLPEAT中疏水性氨基酸占总氨基酸含量的47.87%, 有助于保证蛋白空间结构的稳定性, 使其内部折叠形成二级结构和三级结构。综上所述, MiLPEAT至少在L37~C63区域存在一个跨膜区, 从而说明该蛋白为膜结合蛋白。
通过TargetP、Predotar、ChloroP、SignalP、Phobius和iPSORT等网络服务器(表 2)的预测, 发现MiLPEAT不带有信号肽; BaCelLo的预测结果显示, MiLPEAT定位于细胞核; 而利用Wolfpsort则预测它可能定位在内质网上; Protein Prowler的预测结果表明, MiLPEAT在分泌途径中。这些有关MiPLEAT亚细胞定位的不确定预测也出现在拟南芥中: AtLPEAT1和AtLPEAT2都没有明确的亚细胞定位, 但通过Aramemnon拓扑学一致性搜索发现它们都被预测处在分泌途径[16]; 而在拟南芥细胞器蛋白质组学的研究报道中, AtLPEAT1则被预测定位于内质网上[31]。显然, 有关LPEAT的亚细胞定位需要更多直接的细胞学证据。
在酵母LPEAT所编码蛋白ALE1p的羧基端中存在一个“KKxx”双赖氨酸模体[32], 其作用可能使这个膜蛋白自高尔基体返回并驻留内质网上[33]。有趣的是, MiLPEAT的羧基端也存在这样的模体(图 2), 这在一定程度上支持了MiLPEAT内质网定位并参与分泌途径的Wolfpsort及Protein Prowler预测结果。
由于GPAT9与LPEAT都具有相同的酰基供体并且酰基化相似的酰基受体, 因而在构建系统发育树时可以作为外群[34]。相对于高度保守的GPAT9来说, LPEAT1和LPEAT2序列差异较大, 甚至保守模体都不完全相同(图 2), 这可能解释了为什么1型和2型的LPEAT功能不完全一致[16]。从我们构建的NJ系统发育树上(图 3), 可以了解到1型和2型的LPEAT在进化关系上明显(靴带值分别达到99%和100%)呈两个独立的分支; 本研究所克隆得到的MiLPEAT显然(靴带值为99%)与1型LPEAT聚类在一起(图 3), 预示着MiLPEAT具有1型LPEAT的结构和功能, 为后续功能鉴定等研究奠定了基础。
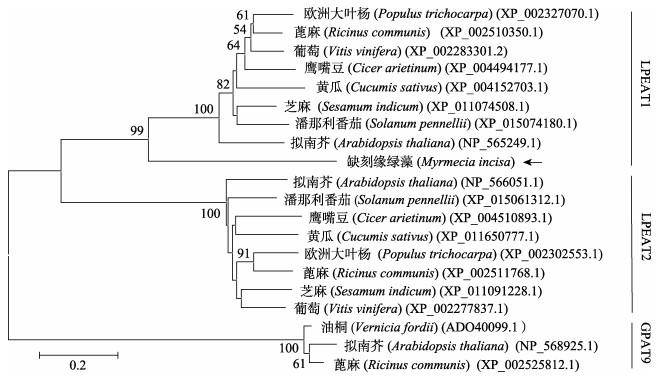
|
图 3 基于LPEAT蛋白氨基酸序列所构建的邻接聚类图 节点上的数据为靴带值, 小于50%的数值不显示; 拉丁学名后括号内的数据指示基因登录号; 箭号指示本研究的物种. Fig.3 Phylogenetic tree inferred from the deduced amino acid sequences of LPEAT genes Branch lengths are proportional to the number of substitutions per site (see the scale bar). The numbers at the nodes indicate the neighbour-joining bootstrap proportion (BP) values (only values≥50% are shown). All of the accession numbers are presented in the parentheses after the Latin names of each species. The position of MiLPEAT is marked by an arrow. |
以β-actin基因作为内参基因, 利用q-RT-PCR方法, 分析了缺刻缘绿藻的MiLPEAT在氮饥饿过程中的基因相对转录量变化。结果表明(图 4), 在氮饥饿培养过程中, MiLPEAT的基因相对转录量在前8 h内快速增加并达到峰值, 氮饥饿8 h时的基因转录量约是刚开始时(0 h)的3.4倍, 两者之间的差异显著(P < 0.05);随后4 h内, 转录量迅速下降, 然后始终在与氮饥饿刚开始时的水平上下波动, 且无明显差异。这些结果说明氮饥饿在刚开始时对缺刻缘绿藻MiLPEAT的基因转录具有明显的上调作用。
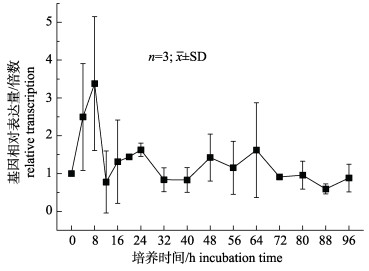
|
图 4 MiLPEAT在氮饥饿过程中的基因相对转录量变化 Fig.4 Transcriptional level of MiLPEAT during the course of nitrogen starvation |
利用LC-MS联用分析技术, 对氮饥饿处理0 d和4 d的缺刻缘绿藻脂质样品进行了定性与定量的分析。结果(图 5)显示, 与处理0 d的脂质样品相比, 经过4 d氮饥饿处理后, 缺刻缘绿藻LPE的相对丰度下降了约49%(P < 0.01), 而PE的相对丰度只增加了10% (P=0.264 > 0.05)。说明氮饥饿过程中, 缺刻缘绿藻中的LPE因被酰基化为PE, 而其量减少。结合MiLPEAT在该过程中的转录量变化(图 4)来分析, LPE的减少及PE的增加与该基因在氮饥饿条件下转录量的上调有一定的关系。
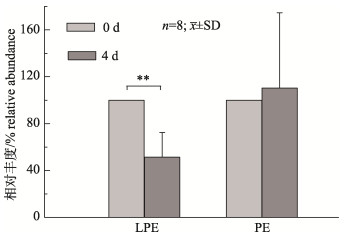
|
图 5 缺刻缘绿藻在氮饥饿胁迫前后溶血磷脂酰乙醇胺(LPE)和磷脂酰乙醇胺(PE)的相对含量变化 “**”表示差异极显著(P < 0.01). Fig.5 Comparison of the lysophosphatidylethanolamine (LPE) and phosphatidylethanolamine (PE) levels before and after the stress of nitrogen starvation "**" means significant difference (P < 0.01). |
本研究是有关真核藻类LPEAT基因研究的首次报道。其实, 自缺刻缘绿藻的转录组数据库[18]中, 本研究筛选到的含有酰基转移酶结构域PlsC的基因不只本研究的MiLPEAT, 还有一个定位于叶绿体中的GPAT基因[35]、一个预测在内质网中的GPAT基因以及一个在分泌途径中的LPAAT基因。通过这些基因序列的克隆与聚类分析(数据未展示)了解到, 本研究所克隆的基因与其他物种的LPEAT聚成一支, 明显不同于GPAT以及LPAAT。在此基础上, 本研究扩大了相应物种的LPEAT数据, 再进行聚类。结果(图 3)显示, MiLPEAT与其他高等植物的LPEAT1关系最近, 但不同于LPEAT2, 支持了Shockey等[34]认为LPEAT的1型与2型有较大差异的结论; MiLPEAT虽与高等植物的LPEAT1相近, 但仍不同于它们(从序列比对图 2中也可以看出), 因而就从LPEAT1这一分支的根部明显独立出一亚支(subclade)。由于缺少藻类LPEAT的确切数据, 这一亚支有待完善; 这个聚类结果是否体现出藻类的低等性, 也有待于进一步探讨。
LPEAT是一类将LPE酰基化为PE的酰基转移酶, 它主要在Lands循环[9]中起作用, 即利用酰基池中的酰基-CoA, 在LPE碳骨架的sn-2上接上酰基, 从而产生PE。通过这样的循环, 使得PE等膜脂上的脂肪酸有机会与酰基池中的脂肪酸发生交换, 这样就能导致膜脂的不对称及多样性[8]。通过序列比对(图 2), 可知本研究自缺刻缘绿藻中克隆的基因所编码的MiPLEAT具有1-酰基甘油- 3-磷酸酰基转移酶(1-acylglycerol-3-phosphate O-acyltransferase, AGPAT)这个家族特征性的保守模体: NH(x)4D、GCxYVxR、FPEGT及PVxPVx[36]。据Lewin等[30]及Yamashita等[36]的报道:在这些模体中, NH(x)4D中的残基Asp(D)和His(H)是重要的酰基转移酶活性中心, 虽在2型中有的Asp突变成了Glu(E), 但在1型中却高度保守, 这也预示着1型和2型的LPEAT在酶活性方面可能存在着差异; FPEGT模体在所有比对的序列中高度保守, 显示了它的重要性, 该模体中的Gly(G)是酰基转移酶的重要活性中心, 而Glu则是底物中磷的结合位点; 模体PVxPVx中的Pro(P)也是酰基转移酶催化活性中心; GCxYVxR模体中的Arg(R)是甘油-3-磷酸底物结合的重要位点。所有这些重要位点在MiLPEAT中都高度保守(图 2), 从而从一级结构上确保了它的酰基转移酶作用。
二级结构的预测及疏水性氨基酸计算结果表明, MiLPEAT至少含有一个跨膜区, 位于L37~C63区域。虽然缺乏明确的亚细胞定位分析结果, 但结合上述LPEAT在膜脂形成中的作用分析以及它与酵母LPEAT[33]羧基端相同的双赖氨酸模体“KKxx”(图 2), 推测这个跨膜蛋白在内质网上定位的可能性较大。
1994年, Jackowski等[37]在大肠杆菌中发现了一个双功能酰基载体蛋白合成酶, 它具有PlsC酰基转移酶保守结构域, 并且能够利用及酰化1-酰基-甘油磷脂乙醇胺(1-acyl-glycerophospho eth anolamine, 1-acylGPE), 这是在动物之外的物种中最早发现的LPEAT。2009年, St lberg等[16]在高等植物的拟南芥中也发现了两个能够酰化LPE的酰基转移酶, 即AtLPEAT1和AtLPEAT2。Jasieniecka-Gazarkiewicz等[17]在拟南芥的LPEAT插入突变株中发现, LPEAT的突变不仅使拟南芥的生长紊乱、叶子变小、根变短、挂果少、种子油脂含量低等特性, 也导致叶中LPE含量大量增加。说明拟南芥LPEAT对LPE的酰基化作用一旦受阻, 就会影响到植株生长等性能。
当缺刻缘绿藻处于氮饥饿条件下, 本研究发现LPE的相对丰度显著性地减少(图 5), 这可能是由于MiLPEAT的基因转录量(图 4)或表达量有所增加, 以致更多的LPE被酰基化为PE。但在图 5中也发现, 在氮饥饿4 d后, 藻细胞中PE的相对丰度并没有达到预期值(在MiLPEAT的作用下, 降低50%的LPE, 理论上就应该多产生相应的PE)。这可能是由于生成的PE大部分被PDAT作用以合成TAG, 从而造成细胞膜脂含量的降低而TAG含量的增加[3, 38]。因为已知TAG既可以沿着Kennedy途径[38-39]从头合成, 也可能由PDAT作用[8, 14], 将PE等膜脂甘油骨架sn-2位上的脂肪酸转至DAG的sn-2位上而形成。当然, 这个推测还需要实验进一步证实。
| [1] |
Reisigl H. Zur systematik und ökologie alpiner Bodenalgen[J]. Österreichische Botanische Zeitschrift, 1964, 111(4): 402-499. DOI:10.1007/BF01372910 |
| [2] |
Tong M, Yu S Y, Ouyang L L, et al. Comparison of increased arachidonic acid content in Myrmecia incisa cultured during the course of nitrogen or phosphorus starvation[J]. Journal of Fisheries of China, 2011, 35(5): 763-773. [童牧, 于水燕, 欧阳珑玲, 等. 氮饥饿与磷饥饿促使缺刻缘绿藻花生四烯酸含量增加的比较[J]. 水产学报, 2011, 35(5): 763-773.] |
| [3] |
Ouyang L L, Li X L, Zhou Z G. Alternations of photosynthetic membrane lipids and triacylglycerol and their fatty acids in Myrmecia incisa grown in a shift from nitrogen starvation to replenishment[J]. Marine Fisheries, 2016, 38(6): 653-662. [欧阳珑玲, 李晓蕾, 周志刚. 缺刻缘绿藻的光合膜脂与中性脂及其脂肪酸组成在氮饥饿/氮恢复培养过程中的变化[J]. 海洋渔业, 2016, 38(6): 653-662. DOI:10.3969/j.issn.1004-2490.2016.06.011] |
| [4] |
Dyer J M, Stymne S, Green A G, et al. High-value oils from plants[J]. Plant Journal, 2008, 54(4): 640-655. DOI:10.1111/j.1365-313X.2008.03430.x |
| [5] |
Park J Y, Kim D K, Wang Z M, et al. Production and characterization of biodiesel from tung oil[J]. Applied Biochemistry and Biotechnology, 2008, 148(1-3): 109-117. DOI:10.1007/s12010-007-8082-2 |
| [6] |
Stymne S, Appelqvist L . The biosynthesis of linoleate from oleoyl-CoA via oleoyl-phosphatidylcholine in microsomes of developing safflower seeds[J]. European Journal of Biochemistry, 1978, 90(2): 223-229. DOI:10.1111/ejb.1978.90.issue-2 |
| [7] |
Sperling P, Linscheid M, St cker S, et al. In vivo desaturation of cis-Δ-9-monounsaturated to cis-Δ9, 12-diunsaturated alkenylether glycerolipids[J]. Journal of Biological Chemistry, 1993, 268(36): 26935-26940. |
| [8] |
Bates P D. Understanding the control of acyl flux through the lipid metabolic network of plant oil biosynthesis[J]. Biochimica et Biophysica Acta (BBA)-Molecuar and Cell Biology of Lipids, 2016, 1861(9 Pt B): 1214-1225. |
| [9] |
Lands W E M. Metabolism of glycerolipides:A comparison of lecithin and triglyceride synthesis[J]. Journal of Biological Chemistry, 1958, 231(2): 883-888. |
| [10] |
Shindou H, Hishikawa D, Harayama T, et al. Generation of membrane diversity by lysophospholipid acyltransferases[J]. Journal of Biochemistry, 2013, 154(1): 21-28. DOI:10.1093/jb/mvt048 |
| [11] |
Kim H U, Lee K R, Go Y S, et al. Endoplasmic reticulum-located PDAT1-2 from castor bean enhances hydroxy fatty acid accumulation in transgenic plants[J]. Plant and Cell Physiology, 2011, 52(6): 983-993. DOI:10.1093/pcp/pcr051 |
| [12] |
Yoon K, Han D, Li Y, et al. Phospholipid:diacylglycerol acyltransferase is a multifunctional enzyme involved in membrane lipid turnover and degradation while synthesizing triacylglycerol in the unicellular green microalga Chlamydomonas reinhardtii[J]. Plant Cell, 2012, 24(9): 3708-3724. DOI:10.1105/tpc.112.100701 |
| [13] |
Fan J, Yan C, Zhang X, et al. Dual role for phospholipid:diacylglycerol acyltransferase:enhancing fatty acid synthesis and diverting fatty acids from membrane lipids to triacylglycerol in Arabidopsis leaves[J]. Plant Cell, 2013, 25(9): 3506-3518. DOI:10.1105/tpc.113.117358 |
| [14] |
Liu X Y, Ouyang L L, Zhou Z G. Phospholipid:diacylglycerol acyltransferase contributes to the conversion of membrane lipids into triacylglycerol in Myrmecia incisa during the nitrogen starvation stress[J]. Scientific Reports, 2016, 6: 26610. DOI:10.1038/srep26610 |
| [15] |
Shindou H, Shimizu T. Acyl-CoA:lysophospholipid acyltransferases[J]. Journal of Biological Chemistry, 2009, 284(1): 1-5. |
| [16] |
Stålberg K, Ståhl U, Stymne S, et al. Characterization of two Arabidopsis thaliana acyltransferases with preference for lysophosphatidylethanolamine[J]. BMC Plant Biology, 2009, 9: 60. DOI:10.1186/1471-2229-9-60 |
| [17] |
Jasieniecka-Gazarkiewicz K, Lager I, Carlsson A S, et al. Acyl-CoA:lysophosphatidylethanolamine acyltransferase activity regulates growth of Arabidopsis[J]. Plant Physiology, 2017, 174(2): 986-998. DOI:10.1104/pp.17.00391 |
| [18] |
Ouyang L L, Chen S H, Li Y, et al. Transcriptome analysis reveals unique C4-like photosynthesis and oil body formation in an arachidonic acid-rich microalga Myrmecia incisa Reisigl H4301[J]. BMC Genomics, 2013, 14: 396. DOI:10.1186/1471-2164-14-396 |
| [19] |
Stanier R Y, Kunisawa R, Mandel M, et al. Purification and properties of unicellular blue-green algae (Order Chlorococcales)[J]. Bacteriology Reviews, 1971, 35(2): 171-205. |
| [20] |
Richards E, Reichardt M, Rogers S. Preparation of genomic DNA from plant tissue[M]. //Ausubel F M, Brent R, Kingston R, et al. Current Protocols in Molecular Biology. New York: John Wiley & Sons, 2003.
|
| [21] |
Thompson J D, Gibson T J, Plewniak F, et al. The CLUSTAL_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research, 1997, 25(24): 4876-4882. DOI:10.1093/nar/25.24.4876 |
| [22] |
Hall T A. BioEdit:a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nucleic Acids Symposium Series, 1999, 41: 95-98. |
| [23] |
Tamura K, Stecher G, Peterson D, et al. MEGA6:Molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [24] |
Yu S Y, Li H, Tong M, et al. Identification of a Δ6 fatty acid elongase gene for arachidonic acid biosynthesis localized to the endoplasmic reticulum in the green microalga Myrmecia incisa Reisigl[J]. Gene, 2012, 493(2): 219-227. DOI:10.1016/j.gene.2011.11.053 |
| [25] |
Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [26] |
Bligh E G, Dyer W J. A rapid method of total lipid extraction and purification[J]. Canadian Journal of Biochemistry and Physiology, 1959, 37(8): 911-917. DOI:10.1139/o59-099 |
| [27] |
Okazaki Y, Kamide Y, Hirai M Y, et al. Plant lipidomics based on hydrophilic interaction chromatography coupled to ion trap time-of-flight mass spectrometry[J]. Metabolomics, 2013, 9(Suppl 1): 121-131. |
| [28] |
Su X, Xu J, Yan X, et al. Lipidomic changes during different growth stages of Nitzschia closterium f. minutissima[J]. Metabolomics, 2013, 9(2): 300-310. DOI:10.1007/s11306-012-0445-1 |
| [29] |
Li Z G, Zeng H Z, Ao P X, et al. Lipid response to short-term chilling shock and long-term chill hardening in Jatropha curcas L. seedlings[J]. Acta Physiologiae Plantarum, 2014, 36(10): 2803-2814. DOI:10.1007/s11738-014-1653-2 |
| [30] |
Lewin T M, Wang P, Coleman R A. Analysis of amino acid motifs diagnostic for the sn-glycerol-3-phosphate acyltransferase reaction[J]. Biochemistry, 1999, 38(18): 5764-5771. DOI:10.1021/bi982805d |
| [31] |
Dunkley T P, Hester S, Shadforth I P, et al. Mapping the Arabidopsis organelle proteome[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(17): 6518-6523. DOI:10.1073/pnas.0506958103 |
| [32] |
Jain S, Stanford N, Bhagwat N, et al. Identification of a novel lysophospholipid acyltransferase in Saccharomyces cerevisiae[J]. Journal of Biological Chemistry, 2007, 282(42): 30562-30569. DOI:10.1074/jbc.M706326200 |
| [33] |
Stornaiuolo M, Lotti L V, Borgese N, et al. KDEL and KKXX retrieval signals appended to the same reporter protein determine different trafficking between endoplasmic reticulum, intermediate compartment, and Golgi complex[J]. Molecular Biology of the Cell, 2003, 14(3): 889-902. DOI:10.1091/mbc.e02-08-0468 |
| [34] |
Shockey J, Regmi A, Cotton K, et al. Identification of Arabidopsis GPAT9(At5g60620) as an essential gene involved in triacylglycerol biosynthesis[J]. Plant Physiology, 2016, 170(1): 163-179. DOI:10.1104/pp.15.01563 |
| [35] |
Ouyang L L, Li H, Yan X J, et al. Site-directed mutagenesis from Arg195 to His of a microalgal putatively chloroplastidial glycerol-3-phosphate acyltransferase causes an increase in phospholipid levels in yeast[J]. Frontiers in Plant Science, 2016, 7: 286. |
| [36] |
Yamashita A, Nakanishi H, Suzuki H, et al. Topology of acyltransferase motifs and substrate specificity and accessibility in 1-acyl-sn-glycero-3-phosphate acyltransferase 1[J]. Biochimica et Biophysica Acta (BBA)-Molecular and Cell Biology of Lipids, 2007, 1771(9): 1202-1215. |
| [37] |
Jackowski S, Jackson P D, Rock C O. Sequence and function of the aas gene in Escherichia coli[J]. Journal of Biological Chemistry, 1994, 269(4): 2921-2928. |
| [38] |
Chen C X, Sun Z, Cao H S, et al. Identification and characterization of three genes encoding acyl-CoA:diacylglycerol acyltransferase (DGAT) from the microalga Myrmecia incisa Reisigl[J]. Algal Research, 2015, 12: 280-288. DOI:10.1016/j.algal.2015.09.007 |
| [39] |
Kennedy E P, Weiss S B. The function of cytidine coenzymes in the biosynthesis of phospholipids[J]. Journal of Biological Chemistry, 1956, 222(1): 193-214. |
 2018, Vol. 25
2018, Vol. 25
