2. 上海海洋大学 农业部淡水水产种质资源重点实验室, 上海 201306
2. Key Laboratory of Freshwater Fishery Germplasm Resources, Ministry of Agriculture; Shanghai Ocean University, Shanghai 201306, China
TGF-β/BMP/activin超家族细胞因子结合细胞质膜上的受体, 经Smad蛋白质家族的介导, 将细胞外信号传递到细胞核内, 调控一系列基因的表达, 最终决定细胞的命运和行为[1]。在从线虫到人类广泛的物种范围内, 该信号通路对细胞应答、分化和发育等生物学过程具有重要的调节作用[2-4]。Smad家族中受体调节型Smad (R-Smad)在细胞因子配体结合细胞膜上的受体后被磷酸化, R-Smad单体转变为活性的三聚体, 进一步与通用辅助因子Smad4交换一个亚基变为更为稳定的异源三聚体[5-6]。异源复合体进入细胞核结合到靶基因的启动子上, 与转录共调节因子相互作用调节靶基因的转录水平的表达[7]。已有的研究表明Smad2/Smad3(属于R-Smad)与人的肌肉萎缩有关, Smad2/Smad3在myostatin、activin A及其他转化生长因子家族成员下游介导肌肉组织中蛋白的丢失和萎缩[8-9]。
对于虾蟹等水产甲壳动物, 目前尚未见到有关于Smad基因克隆及功能的研究报道。中华绒螯蟹(Eriocheir sinensis)是中国重要的经济养殖动物, 其肌肉生长和蜕壳周期密切相关, 尤其是肥大的螯足肌肉, 蜕壳前发生可逆性萎缩以顺利蜕出狭小的关节, 蜕壳后再恢复重建[10]。为了探讨中华绒螯蟹Smad是否参与了蜕壳过程中肌肉萎缩和生长的生理调节, 本研究克隆了编码中华绒螯蟹全长Smad3(命名为EsSmad3)的cDNA序列, 并研究了其在性成熟个体的不同组织及幼体不同蜕壳时期不同部位肌肉组织的转录表达情况。本工作为进一步研究Smad3可能参与中华绒螯蟹蜕壳过程中肌肉萎缩和生长调节的分子机制打下基础。
1 材料与方法 1.1 实验材料中华绒螯蟹取自上海海洋大学崇明养殖基地, 体重(126±7.41) g, 取其肝胰腺、表皮、眼柄、心肌、精巢、卵巢、螯足、鳃及三角膜等组织, 置于–80℃冰箱保存备用。另取体重(8.24±2.17) g的一年生幼蟹, 按照文献[11-12]的方法鉴定蜕皮周期, 鉴定出处于蜕皮间期C期、蜕皮前晚期D3–4期及蜕皮后A~B期个体, 分别取其螯足、第三步行足和腹部第Ⅴ腹节肌肉, 置于–80℃冰箱保存备用。
1.2 实验方法 1.2.1 中华绒螯蟹EsSmad3基因全长克隆利用Trizol法提取中华绒螯蟹肝胰腺组织总RNA, 同时用寡聚T引物和六聚体随机引物将其反转录成cDNA第一链作为PCR的模板, 根据NCBI公共数据库的转录组序列信息设计引物(表 1), 进行PCR反应, 获得EsSmad3 cDNA核心片段。PCR反应加样体系: cDNA模板1 μL, 上下游引物(10 µmol/L)各2 μL, 2×PCR Mix 25 μL, 加无菌去离子水至总体积50 μL。设置的PCR反应条件: 94℃预变性4 min; 94℃变性30 s, 56.5℃退火30 s, 72℃延伸1 min, 35个循环; 72℃延伸10 min。
|
|
表 1 中华绒螯蟹Smad3基因序列扩增引物信息 Tab.1 The information of the primers for Smad3 amplification in Eriocheir sinensis |
根据得到的EsSmad3 cDNA的核心序列, 采用Primer Premier 6.0软件进行3′(5′) RACE引物设计, 由生工生物工程(上海)股份有限公司进行合成(表 1)。3′(5′) Race模板采用GeneRacerTM Kit (Invitrogen, 货号: L1500-01)进行合成。以RACE cDNA第一链为模板, 用EsSmad3-3′(5′)Race-F1和3′(5′) Gene Racer outer primer(试剂盒提供)进行第一轮PCR, 反应条件: 3′RACE, 94℃预变性2 min, 94℃ 30 s、72℃ 1min、5循环, 94℃ 30 s、70℃ 1 min、5循环, 94℃ 30 s、66℃ 1 min、25循环; 5′Race, 94℃预变性2 min, 94℃ 30 s、72℃ 2 min、5循环, 94℃ 30 s、70℃ 2 min、5循环, 94℃ 30 s、66℃ 2 min、25循环。然后, 以第一轮PCR产物稀释液为模板, 用EsSmad3-3′(5′)Race-F2和3′(5′) Gene Racer inner primer(试剂盒提供)行第二轮PCR。反应程序为: 3′RACE 94℃预变性2 min, 94℃ 30 s、66℃ 1 min、30个循环; 5′ RACE 94℃预变性2 min, 94℃ 30 s、66℃ 1 min, 30个循环。PCR产物经1.2%的琼脂糖电泳检测, 切胶回收, 克隆至pUCm-T载体, 蓝白斑筛选后送上海生工生物有限公司测序。
1.2.2 中华绒螯蟹Smad3的生物信息学分析Race技术获得的两端cDNA序列拼接使用DNAStar软件; 序列的相似性分析使用BLAST (http://www.ncbi.nlm.nih.gov/blast)和Bioedit (http://www.mbio.ncsu.edu/bioedit/bioedit.html)上的Clustal[13]; 开放阅读框预测及蛋白质序列的转换使用了NCBI网站的ORF finder[14](https://www.ncbi.nlm.nih.gov/orffinder/)和Expasy的翻译工具(http://web.expasy.org/translate/), cDNA序列和氨基酸序列编辑使用Bioedit; 结构域预测使用SMART (http://smart.embl-heidelberg.de)和NCBI网站的BLAST搜索工具及CDD数据库[15]; 利用同源建模工具SWISS-MODEL (https://www.swissmodel.expasy.org/interactive)和Pymol[16]软件对蛋白质结构进行建模和分析。多序列比对使用了Bioedit上的Clustal多序列比对工具; 系统发育树构建使用MEGA6.0[17]上的Clustal序列比对工具和最大似然(ML)法发育树构建工具, 进行1000次自展检验(bootstrap)评估进化树分支可信度。
1.2.3 实时荧光定量PCR检测EsSmad3基因表达以不同组织及不同蜕壳时期不同部位肌肉组织的cDNA作为荧光定量PCR (qRT-PCR)模板, 荧光定量PCR引物和内参根据EsSmad3 cDNA序列和中华绒螯蟹18S RNA基因序列设计(表 1)。利用BIO-RAD CFX Connect™荧光定量仪的2–ΔΔCt法检测EsSmad3 mRNA的各组织相对表达情况, 反应程序设置为95℃ 3 min; 95℃ 10 s, 55℃20 s, 72℃ 20 s, 75℃5 s, 40个循环; 熔解曲线从65℃上升到95℃, 0.5℃/s。数据分析利用SPSS 18.0软件中的单因子变异数, 差异显著性以0.05为标准, 数据结果用平均值±标准差(x±SD)表示。
2 结果与分析 2.1 中华绒螯蟹Smad3序列分析本研究克隆到的编码EsSmad3的cDNA全长为2021 bp(图 1, GenBank登录号为KY858966), 包括部分5′非翻译区(5′-UTR, 36 bp)和3′非翻译区(3′-UTR, 656 bp); 37~1365位核苷酸是一个编码442个氨基酸的最大开放阅读框(ORF), BLASTP同源搜索结果表明这些氨基酸形成的蛋白与NCBI蛋白非冗余数据库中其他物种的Smad3具有最高的相似性。

|
图 1 中华绒螯蟹Smad3基因核苷酸序列及推测的氨基酸序列 方框内为起始密码子和终止密码子. Fig.1 Nucleotide and deduced amino acid sequences of EsSmad3 Frames indicate the start codon and stop codon respectively. |
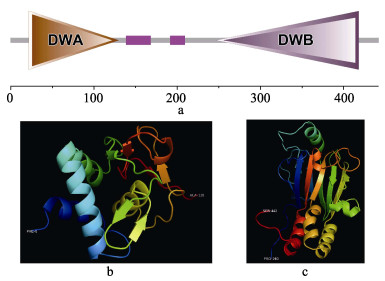
利用SMART (Simple Modular Architecture Research Tool)进行结构域分析显示, EsSmad3具有两个特征性结构域DWA(由22~133位氨基酸构成)和DWB(由247~418位氨基酸构成)(图 2); 用BLAST搜索NCBI的CDD(conserved domain database)数据库显示EsSmad3含有MH1 (7-128位氨基酸)和MH2 (241~431位氨基酸)两个结构域。利用swiss-model同源建模工具预测分别得到的由6~126位氨基酸组成的MH1结构域和240~442位氨基酸组成的MH2结构域三维结构模型(图 2), MH1结构域由4个α螺旋、6个β折叠及5个loop形成紧凑的球状结构, MH2域的结构特点是含有一个β三明治结构, 由多个β折叠组成夹芯, 两端由保守的3个α螺旋和loop-α螺旋结构所夹裹。
|
图 2 EsSmad3功能结构域和三维结构 a.结构域组成; b. DWA(MH1)结构域三维结构; c. DWB(MH2)结构域三维结构. Fig.2 The functional domains and 3-D structure of the EsSmad3 a. Domain architecture; b. 3-D structure of DWA (MH1) domain; c. 3-D structure of DWB (MH2) domain. |
将EsSmad3与人(Homo sapiens)、斑马鱼(Danio rerio)、果蝇(Drosophila melanogaster)中的同源蛋白序列进行比较(序列号分别为NP_005893.1、NP_778258.1、NP_511079.1), 序列一致性分别为0.679、0.691、0.619。序列较保守的区域位于MH1结构域和MH2结构域(图 3下划线部分), EsSmad3的MH1与上述序列一致性分别为0.720、0.736、0.626, MH2的序列一致性分别为0.927、0.922、0.943。形成三聚体化界面的氨基酸(图 3“#”标注部分)位于MH2结构域, 除3个氨基酸位点不同外(

|
图 3 Smad/Smad3蛋白序列在4种动物中的比较
Es、Dm、Dr、Hs分别表示中华绒螯蟹、黑腹果蝇、斑马鱼和人的Smad蛋白序列. MH1结构域用粗下划线表示, MH2结构域用双下划线表示, *表示的氨基酸为DNA结合位点, 方框内的氨基酸为锌原子结合位点, #标识的氨基酸为蛋白质三聚化相互作用界面, 其中 |
|
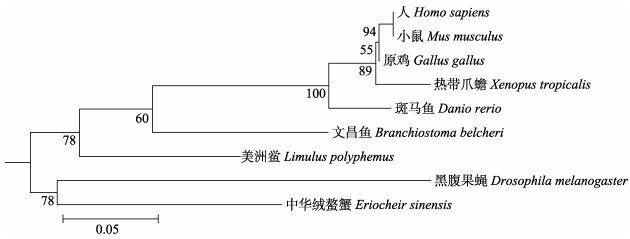
图 4 基于不同物种Smad/Smad3氨基酸序列构建的ML系统发育树 物种及其Smad/Smad3的GenBank序列号分别为: Branchiostoma belcheri, 文昌鱼, XP_019622748.1; Drosophila melanogaster, 黑腹果蝇, NP_511079.1; Danio rerio, 斑马鱼, NP_778258.1; Eriocheir sinensis, 中华绒螯蟹, KY858966; Gallus gallus, 原鸡, NP_989806.1; Homo sapiens, 人, NP_005893.1; Limulus polyphemus, 美洲鲎, XP_013776260.1; Mus musculus, 小鼠, NP_058049.3; Xenopus tropicalis, 热带爪蟾, NP_001008436.1. Fig.4 Phylogenetic tree derived from multiple alignments of Smad/Smad3 amino acid sequences from various species The Accession No. of each Smad/Smad3 are as follows: Branchiostoma belcheri, XP_019622748.1; Drosophila melanogaster, NP_511079.1; Danio rerio, NP_778258.1; Eriocheir sinensis, KY858966; Gallus gallus, NP_989806.1; Homo sapiens, NP_005893.1; Limulus polyphemus, XP_013776260.1; Mus musculus, NP_058049.3; Xenopus tropicalis, NP_001008436.1. |
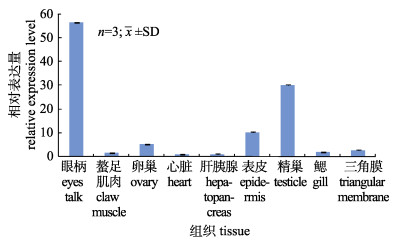
以基因特异性引物EsSmad3-RTF/RTR为引物, 18S RNA为内参基因, 利用荧光定量PCR方法检测EsSmad3 mRNA在中华绒螯蟹性成熟期眼柄、螯足肌肉、卵巢、心脏、肝胰腺、表皮、精巢、鳃和三角膜等9种组织的表达情况, 如图 5所示, EsSmad3在上述所有检测的9种组织中均有表达, 在眼柄中表达量最高, 其次为精巢; 在心脏和肝胰腺中表达量最低。
|
图 5 中华绒螯蟹Smad3基因在不同组织中的相对表达 标准差上方不同字母表示差异显著(P < 0.05). Fig.5 The relative expression level of EsSmad3 in different tissues Different letters above the error bars indicate significant differences (P < 0.05). |
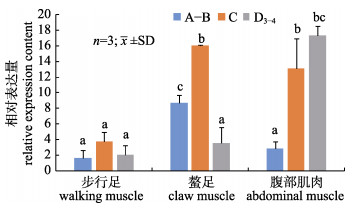
取不同蜕壳时期中华绒螯蟹幼蟹的螯足、步行足和腹部肌肉组织, 以EsSmad3-RTF/RTR为引物, 18S RNA为内参基因, 利用荧光定量PCR方法检测各组织EsSmad3 mRNA的表达情况(图 6), 结果显示, 在一个完整的蜕壳周期中, 步行足肌肉组织中EsSmad3 mRNA表达在蜕壳间期高于蜕壳前D3-4期和蜕壳后A~B期, 但无显著的统计学差异(P > 0.05)。螯足肌肉在蜕壳前晚期D3–4期急剧下调(P < 0.05), 蜕壳后A~B期开始表达量显著升高(P < 0.05), 直至蜕皮间期C期。腹部肌肉组织中EsSmad3 mRNA水平在蜕皮间期C期显著高于蜕壳后A~B期, 这种上调一直持续到蜕壳前晚前期D3-4。
|
图 6 中华绒螯蟹Smad3基因在蜕壳过程中不同部位肌肉组织中的相对表达 标准差上方不同字母表示差异显著(P < 0.05). A~B:蜕壳后期; C:蜕壳间期; D3-4:蜕壳前晚期. Fig.6 The relative expression content of EsSmad3 mRNA in different muscle tissues during the molt cycle Different letters above the error bars indicate significant differences (P < 0.05). A-B: post-molt stage; C: inter-molt stage; D3-4: later post-molt stage. |
在脊椎动物中, 包括Smad3和Smad4在内的R-Smad以及Smad4具有相同的结构域组成:包括一个N端的DNA结合结构域(MH1结构域)和一个C端的效应结构域(MH2结构域), 二者由不保守的氨基酸组成的连接子(linker)连接。MH2结构域是一个功能多样的蛋白质-蛋白质相互作用模块[7]。利用Race技术我们首次克隆得到编码中华绒螯蟹Smad3的cDNA序列, 最大的开放阅读框编码一个442个氨基酸长度的蛋白质, 用BLAST搜索NCBI的CDD数据库显示其两端分别含有MH1和MH2两个结构域, 以及在不同结构域中的DNA结合位点以及蛋白质相互作用界面; SMART结构域预测得出DWA (Domain A in dwarfin family proteins)和DWB (Domain B in dwarfin family proteins)两个结构域, 据SMART结构域数据库信息这两个结构域在Smad蛋白中分别代表MH1和MH2;三维结构的同源建模得到MH1和MH2结构域的立体模型, 其立体结构与人的MH1和MH2的晶体结构解析结果相近[1, 18]。BLAST搜索结果显示EsSmad3与脊椎动物的Smad3更为相似。多序列比对发现, 4种动物(人、小鼠、黑腹果蝇、中华绒螯蟹)Smad3具有非常保守的MH1和MH2结构域以及锌原子结合位点、三聚体化位点等活性位点; DNA结合功能所涉及的氨基酸在人、小鼠、中华绒螯蟹Smad3中是完全相同; 人和小鼠Smad3的C末端是SSVS, 中华绒螯蟹和果蝇的C末端是SSMS, 这种SSXS氨基酸基序的最后两个丝氨酸是R-Smad发生磷酸化的位点[19-20]。总之, 从一级序列的比较到结构域预测、三维结构同源模拟的结果看, 本实验获得的cDNA序列编码的蛋白质是中华绒螯蟹Smad3 (EsSmad3)。
最近的研究表明, 人(或模式动物小鼠)Smad家族多种蛋白参与肌肉生长的控制[21-22]。转化生长因子β (TGFβ)家族配体如Myostatin、TGFβ结合到肌细胞活化素Ⅱ型受体ActRⅡA、ActRⅡB及TGFβRⅡ上, 进一步募集酪氨酸激酶活化素Ⅰ型受体(ALK4, ALK5, ALK7), 导致Smad2和Smad3 (Smad2/Smad3)磷酸化。Smad2/Smad3和Smad4形成转录调节复合体进入细胞核, 通过影响染色质的结构降低一系列肌肉生长相关基因的表达[21]。另一方面, 骨形态发生蛋白(Bone Morphogenetic Protein, BMP)结合到其细胞膜受体BMPRⅡ或ActRⅡA/ActRⅡB上, 募集和激活另一些活化素Ⅰ型受体(ALK2, ALK3, ALK6), 导致Smad家族中的Smad1/5/8磷酸化及活化[23]。活化后的Smad1/5/8复合体和活化的Smad2/3对肌肉生长的影响相反, 它以与Smad2/3类似的方式与Smad4结合成Smad1/5/8-Smad4转录调控复合体, 提高细胞生长分化相关的基因包括肌肉生长相关基因的表达[22, 24]。用转基因小鼠模型研究表明, 肌肉萎缩和肌肉过度生长之间的平衡依赖于Smad4被Myostatin信号通路还是被BMP信号通路所募集之间的平衡[25]。
利用荧光实时定量RT-PCR技术检测性成熟期动物体不同组织中EsSmad3转录水平, 结果显示其在眼柄、螯足肌肉、卵巢、心脏、肝胰腺、表皮、精巢、鳃和三角膜都有表达, 而在眼柄和精巢中表达显著。这或许因为性成熟期动物组织器官中细胞增生分化和激素分泌活动较为旺盛, 有待于进一步深入研究。
同时, 荧光实时定量RT-PCR技术检测结果显示, 在中华绒螯蟹幼蟹一个完整的蜕壳周期中, 螯足、步行足和腹部肌肉中EsSmad3的表达量都表现为蜕皮间期C高于蜕壳后A–B期, 这和甲壳动物从A–B期到C期蛋白质合成速度的增加及肌肉总量增加相一致, 而此时正是动物肌肉生长的时期[26-27]。在蜕壳前晚期D3–4期, 螯足肌肉中EsSmad3的mRNA水平急剧下降, 此时肌肉在形态上表现为肌纤维横截面积减小, 肌丝降解, 肌肉重量降低以便蜕壳时顺利蜕出狭小的关节[28]。蜕壳前并不发生明显萎缩的步行足肌肉中, EsSmad3的mRNA水平相对于蜕壳间期略有下降, 但无统计学差异。腹部肌肉中EsSmad3基因的表达在蜕壳前晚期D3-4期则急剧上调。已有研究证实, 此时甲壳动物步行足和腹部肌肉蛋白质合成速率相对于蜕壳间期是增加的, 这有助于蜕皮后肌肉的生长, 如肌纤维长度增加(肌节数目增加或者原有肌节拉长)、肌原纤维(粗肌丝和细肌丝)数目增加等[29-31]。当然, 肌肉的生长发育受到多种基因和信号通路的调控, 如叉头转录因子FOXO、核因子NF-kB及PI3K-Akt-mTOR信号通路等, 肌肉最终的生长发育状况是由各种调节因素共同作用的结果[31]。本研究表明, 在中华绒螯蟹蜕壳过程中EsSmad3基因在不同部位的肌肉组织中具有不同的表达模式, 与蜕壳周期密切相关, 也与肌肉组织的形态结构变化紧密相连, 说明EsSmad3参与了中华绒螯蟹蜕壳诱导的肌肉萎缩、生长及重建过程。
在脊椎动物中的一些经济物种如牛、羊、猪或者用以进行医学研究的模式物种如小鼠, 肌肉的生长发育及其分子机制已经取得了长足进展; 但是在甲壳动物, 这一领域的研究还很薄弱, 本研究通过RACE克隆技术获得编码EsSmad3全长的cDNA序列, 分析了其表达特征, 为进一步研究甲壳动物肌肉生长发育机制打下了基础。
| [1] |
Shi Y G, Wang Y F, Jayaraman L, et al. Crystal structure of a Smad MH1 domain bound to DNA:insights on DNA binding in TGF-β signaling[J]. Cell, 1998, 94(5): 585-594. DOI:10.1016/S0092-8674(00)81600-1 |
| [2] |
Heldin C H, Miyazono K, Ten D P. TGF-β signalling from cell membrane to nucleus through SMAD proteins[J]. Nature, 1997, 390(6659): 465-471. DOI:10.1038/37284 |
| [3] |
Derynck R, Zhang Y, Feng X H. Smads:transcriptional activators of TGF-beta responses[J]. Cell, 1998, 95(6): 737-740. DOI:10.1016/S0092-8674(00)81696-7 |
| [4] |
Attisano L, Wrana J L. Smads as transcriptional co-modulators[J]. Current Opinion in Cell Biology, 2000, 12(2): 235-243. DOI:10.1016/S0955-0674(99)00081-2 |
| [5] |
Wrana J L. Phosphoserine-dependent regulation of protein-protein interactions in the Smad pathway[J]. Structure, 2002, 10(1): 5-7. |
| [6] |
Wu G, Chen Y G, Ozdamar B, et al. Structural basis of Smad2 recognition by the Smad anchor for receptor activation[J]. Science, 2000, 287(5450): 92-97. DOI:10.1126/science.287.5450.92 |
| [7] |
Qin B Y, Lam S S, Correia J J, et al. Smad3 allostery links TGF-β receptor kinase activation to transcriptional control[J]. Genes & Development, 2002, 16(15): 1950-1963. |
| [8] |
Sartori R, Milan G, Patron M, et al. Smad2 and 3 transcription factors control muscle mass in adulthood[J]. American Journal of Physiology-Cell Physiology, 2009, 296(6): C1248-C1257. DOI:10.1152/ajpcell.00104.2009 |
| [9] |
Cohen S, Nathan J A, Goldberg A L. Muscle wasting in disease:molecular mechanisms and promising therapies[J]. Nature Reviews Drug Discovery, 2015, 14(1): 58-74. DOI:10.1038/nrd4467 |
| [10] |
Tian Z H, Jiao C Z. Muscle atrophy and growth induced by molting in Crustacean:a review[J]. Fisheries Science, 2016, 35(5): 603-606. [田志环, 焦传珍. 甲壳动物蜕皮诱导的肌肉萎缩与生长[J]. 水产科学, 2016, 35(5): 603-606.] |
| [11] |
Tian Z H, Kang X J, Mu S M. The molt stages and the hepatopancreas contents of lipids, glycogen and some selected inorganic elements during the molt cycle of the Chinese mitten crab Eriocheir sinensis[J]. Fisheries Science, 2012, 78(1): 67-74. DOI:10.1007/s12562-011-0426-8 |
| [12] |
Kang X J, Tian Z H, Wu J L, et al. Molt stages and changes in digestive enzyme activity in hepatopancreas during molt cycle of Chinese mitten crab, Eriocheir sinensis[J]. Journal of Fishery Sciences of China, 2012, 19(5): 806-812. [康现江, 田志环, 吴江立, 等. 中华绒螯蟹蜕皮周期及蜕皮过程中肝胰腺消化酶活性的变化[J]. 中国水产科学, 2012, 19(5): 806-812.] |
| [13] |
Larkin M A, Blackshields G, Brown N P, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23(21): 2947-2948. DOI:10.1093/bioinformatics/btm404 |
| [14] |
Rombel I T, Sykes K F, Rayner S, et al. ORF-FINDER:a vector for high-throughput gene identification[J]. Gene, 2002, 282(1-2): 33-41. DOI:10.1016/S0378-1119(01)00819-8 |
| [15] |
Marchler-Bauer A, Derbyshire M K, Gonzales N R, et al. CDD:NCBI's conserved domain database[J]. Nucleic Acids Research, 2015, 43(D1): D222-D226. DOI:10.1093/nar/gku1221 |
| [16] |
Rigsby R E, Parker A B. Using the PyMOL application to reinforce visual understanding of protein structure[J]. Biochemistry and Molecular Biology Education, 2016, 44(5): 433-437. DOI:10.1002/bmb.v44.5 |
| [17] |
Tamura K, Stecher G, Peterson D, et al. MEGA6:Molecular Evolutionary Genetics Analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12): 2925-2729. |
| [18] |
Wu J W, Hu M, Chai J J, et al. Crystal structure of a phosphorylated Smad2[J]. Molecular Cell, 2001, 8(6): 1277-1289. DOI:10.1016/S1097-2765(01)00421-X |
| [19] |
Abdollah S, Macı́A S Silva M, Tsukazaki T, et al. TβRI phosphorylation of Smad2 on Ser465 and Ser467 is required for Smad2-Smad4 complex formation and signaling[J]. Journal of Biology Chemistry, 1997, 272(44): 27678-27685. DOI:10.1074/jbc.272.44.27678 |
| [20] |
Souchelnytskyi S, Tamaki K, Engstr m U, et al. Phosphorylation of Ser465 and Ser467 in the C terminus of Smad2 mediates interaction with Smad4 and is required for transforming growth factor-b signaling[J]. Journal of Biology Chemistry, 1997, 272(44): 28107-28115. DOI:10.1074/jbc.272.44.28107 |
| [21] |
Gaarenstroom T, Hill C S. TGF-β signaling to chromatin:How Smads regulate transcription during self-renewal and differentiation[J]. Seminars in Cell & Developmental Biology, 2014, 32: 107-118. |
| [22] |
Rindom E, Vissing K. Mechanosensitive molecular networks involved in transducing resistance exercise-signals into muscle protein accretion[J]. Frontiers in Physiology, 2016, 7: 547. |
| [23] |
Walsh D W, Godson C, Brazil D P, et al. Extracellular BMP-antagonist regulation in development and disease:tied up in knots[J]. Trends in Cell Biology, 2010, 20(5): 244-256. DOI:10.1016/j.tcb.2010.01.008 |
| [24] |
Miyazono K, Miyazawa K. Id:a target of BMP signaling[J]. Science's STKE, 2002, 2002(151): pe40. |
| [25] |
Amthor H, Sartori R, Schirwis E, et al. O.20 BMP signalling controls muscle mass[J]. Neuromuscular Disorders, 2013, 23(9-10): 850-851. |
| [26] |
de Oliveira Cesar J R, Zhao B P, Malecha S, et al. Morphological and biochemical changes in the muscle of the marine shrimp Litopenaeus vannamei during the molt cycle[J]. Aquaculture, 2006, 261(2): 688-694. DOI:10.1016/j.aquaculture.2006.08.003 |
| [27] |
Covi J A, Bader B D, Chang E S, et al. Molt cycle regulation of protein synthesis in skeletal muscle of the blackback land crab, Gecarcinus lateralis, and the differential expression of a myostatin-like factor during atrophy induced by molting or unweighting[J]. Journal of Experimental Biology, 2010, 213(1): 172-183. DOI:10.1242/jeb.034389 |
| [28] |
Ismail S Z M, Mykles D L. Differential molt-induced atrophy in the dimorphic claws of male fiddler crabs, Uca pugnax[J]. Journal of Experimental Zoology Part A:Ecological and Integrathe Plysiclogy, 1992, 263(1): 18-31. |
| [29] |
El Haj A J, Govind C K, Houlihan D F. Growth of lobster leg muscle fibers over intermolt and molt[J]. Journal of Crustacean Biology, 1984, 4(4): 536-545. DOI:10.2307/1548067 |
| [30] |
Haj A, Clarke S, Harrison P, et al. In vivo muscle protein synthesis rates in the American lobster Homarus americanus during the moult cycle and in response to 20-hydroxyecdysone[J]. Journal of Experimental Biology, 1996, 199: 579-585. |
| [31] |
Tian Z H, Jiao C Z, Wu X G, et al. Changes in histology and main proteins content in claw closer muscle of Chinese mitten crab Eriocheir sinensis during the molt cycle[J]. Journal of Fishery Sciences of China, 2017, 24(5): 1072-1078. [田志环, 焦传珍, 吴旭干, 等. 中华绒螯蟹蜕壳过程中螯足肌肉组织学及主要蛋白质含量变化[J]. 中国水产科学, 2017, 24(5): 1072-1078.] |
| [32] |
Abuhagr A M, MacLea K S, Chang E S, et al. Mechanistic target of rapamycin (mTOR) signaling genes in decapod crustaceans:cloning and tissue expression of mTOR, Akt, Rheb, and p70 S6 kinase in the green crab, Carcinus maenas, and blackback land crab, Gecarcinus lateralis[J]. Comparative Biochemistry and Physiology Part A:Molecular & Integrative Physiology, 2014, 168: 25-39. |
 2018, Vol. 25
2018, Vol. 25

