2. 浙江澳凌水产种业科技有限公司, 浙江 长兴 313106;
3. 上海海洋大学 上海市教委水产动物遗传育种协同创新中心, 上海 201306
2. Zhejiang Aoling Aquatic Seed Industry Technology Co., Changxing 313106, China;
3. Collaborative Innovation Center of Aquatic Animal Breeding Center Certified by the Shanghai Municipal Education Commission; Shanghai Ocean University, Shanghai 201306, China
中华绒螯蟹(Eriocheir sinensis)又称河蟹(以下简称河蟹)、大闸蟹、毛蟹, 营养价值高, 是中国久负盛名的美食, 也是重要的淡水养殖种类, 主要养殖于长江、黄河、辽河水系, 2015年全国中华绒螯蟹养殖总产量约82.4万t, 占淡水养殖甲壳类产量的30.60%, 具有极其重要的地位[1]。由于多年来中华绒螯蟹养殖群体的近亲繁殖、采用小规格亲本人工育苗等原因, 导致中华绒螯蟹群体种质严重退化, 养殖性能下降[2], 因此迫切的需要对不同水系的中华绒螯蟹进行种质资源的保护和良种选育, 而良种选育很重要的一个环节是进行家系选育。通过构建大规模家系, 能够准确地了解系谱信息, 对亲本进行有效的筛选[3], 这既能避免近亲繁殖, 又能不断地进行优良性状的遗传改良, 缩短育种时间, 提高中华绒螯蟹产量。
为了保证精确的家系信息, 以往的家系主要靠分池饲养, 但是大规模的家系分池饲养使得基础设施投入加大, 管理强度增加, 造成资源的大量消耗。如果进行家系同池混养不但可以解决这个问题, 而且可以避免其他环境因素干扰, 减少因环境差异而引起的遗传参数的估计偏差[4], 但是最终需要对同池混养的家系进行亲缘关系鉴定。传统上, 水产动物同池混养家系的鉴定方法是进行电子标记区分, 但是中华绒螯蟹具有褪壳的生物学特性, 需要成熟的植入技术以及被标记的中华绒螯蟹个体规格足够大, 否则电子标记容易脱落且死亡率高, 所以电子标记技术用于中华绒螯蟹家系的应用有待开发。而DNA分子标记技术具有多态性高、共显性遗传、分布广、检测手段简单快速、重复性好等优点[5], 因此常被应用于水生动物的品种检测、基因定位、资源评价、物种亲缘关系分析等方面, 具有广阔的应用前景, 其中微卫星(microsatellites)又称简单序列重复(simple sequence repeats, SSR), 是重要的分子标记之一。微卫星标记广泛分布于真核生物基因组中, 因为其种类多、分布广, 具有高度的变异性、多态性、共显性、遵循孟德尔遗传定律等优点, 在水产动物的分子标记辅助育种和家系系谱分析等方面得到广泛的应用[6-8]。
微卫星多重PCR(multiplex PCR)是指在同一个反应中同时扩增两个或多个位点的聚合酶链式反应[9]。相比于微卫星单独的PCR扩增, 多重PCR具有省时高效、经济节约的优点, 无论是在人类医学还是在动植物研究中均有成熟的应用[10-12], 特别是对于水产动物基于大样本的亲子鉴定和遗传多样性评估时, 能减少人工操作失误, 减少模板的使用, 提供快捷、高效、廉价的方法[13], 在三疣梭子蟹(Portunus trituberculatus) [14-15]、美洲牡蛎(Crassostrea virginica Gmelin)[16]、斑节对虾(Penaeus monodon)[17]和中国对虾(Fenneropenaeus chinense)[18]等水产动物中, 均有微卫星多重PCR的研究报道。但是, 目前对于中华绒螯蟹的微卫星研究, 主要集中于微卫星标记开发[19-20]、群体遗传多样性评估[21-22]和QTL图谱定位[23]等方面, 还未见有关中华绒螯蟹微卫星标记多重PCR体系建立和家系亲子鉴定的相关报道, 这不利于大规模中华绒螯蟹育种的进程。鉴于此, 本研究首次建立了中华绒螯蟹的微卫星多重PCR体系, 并成功将其应用于中华绒螯蟹家系的亲子鉴定中, 大大提高了分析效率, 为中华绒螯蟹的种群体选育和家系管理提供一种便捷、高效的技术方法, 促进了中华绒螯蟹产业的健康发展。
1 材料方法 1.1 样本采集实验所用中华绒螯蟹来源于上海海洋大学基地构建的系谱图已知的2014年家系, 选取其中3个养殖家系(辽河家系、长江家系、黄河家系)共95个子代体, 以及3个家系所对应的亲本。此外, 选取20个2015年中华绒螯蟹养殖群体个体, 用于微卫星标记多态性的筛选以及多重PCR体系的构建。所有个体均取其胸肢肌肉组织, 用95%乙醇固定24 h后−20℃冻存。
1.2 DNA提取将上述肌肉固定样品参照刘皓等[24]优化的酚-氯仿法抽提基因组DNA, 采用1%的琼脂糖凝胶电泳检测提取DNA的完整性, 核酸蛋白检测仪检测DNA浓度和质量, 将合格的DNA用ddH2O稀释至50 ng/µL, −40℃保存备用。
1.3 微卫星多重PCR引物的筛选以及体系优化从本实验室自主开发的微卫星标记中选取多态性好的位点, 合成引物时用荧光基团FAM或者HEX修饰上游引物的5′端(表 1)。将每对引物的片段大小、退火温度、引物的荧光标记等信息据输入Multiplex Manager1.0软件[25]进行模拟, 检测组合反应体系中的引物是否会产生错配、引物二聚体和发卡结构等, 模拟出几组最优的多重PCR引物组合。
|
|
表 1 中华绒螯蟹4组微卫星多重PCR的特性 Tab.1 Characteristics of the four set multiplex PCR panels of microsatellites in Eriocheir sinensis |
将模拟出的几组多重PCR引物分别进行反应体系构建及优化, 使用的DNA模板为2015年群体养殖中华绒螯蟹的20个个体的基因组DNA。初始的PCR反应体系为: PCR反应总体积为12.5 µL, 其中包括DNA模板1 µL(约50 ng), Premix 2×Taq(生工生物工程股份有限公司, 上海) 6.25 µL, 正反向引物各0.35 µL(引物浓度为10 pmol/µL)(引物由上海迈浦生物科技有限公司合成), ddH2O补充体积至12.5 µL, 避光冰浴操作。PCR扩增采用反应程序: 94℃预变性3 min; 94℃变性1 min, 55℃退火1 min, 72℃延伸1 min, 循环35次; 再72℃延伸10 min, 最终16℃保存。每次反应后, 将产物送上海迈浦生物科技有限公司进行STR测序分析(毛细管电泳检测ABI3730XL全自动DNA测序仪)。根据测序结果的峰值图进行每个引物反应量的调整, 每次调整0.05 µL量, 直至多重PCR反应体系各引物的扩增效率基本一致。最终将优化好的微卫星多重PCR组合用于3个中华绒螯蟹家系的亲子鉴定。
1.4 数据统计分析采用Gene Mapper4.0软件读取等位基因数据; 用POPGEN 3.2软件分析等位基因数(Na)、有效等位基因数(Ne)、期望杂合度(He)、观测杂合度(Ho)、香农信息指数(I); 采用PIC_CALC 0.6软件分析多态信息含量(PIC); 利用Cervus 3.0软件对基因型数据进行亲权分析。
2 结果与分析 2.1 微卫星多重PCR构建以2015年中华绒螯蟹养殖群体的20个个体的DNA为模板, 从150个微卫星位点中筛选40个多态性最好的位点, 将这40个多态性位点用Multiplex Manager 1.0软件模拟组合, 成功模拟组合出10组微卫星多重PCR体系。再将这10组多重PCR体系分别进行实验, 根据扩增效率分别调整引物浓度, 最终确立4组四重PCR体系, 如表 1所示。部分中华绒螯蟹个体的微卫星多重PCR等位基因峰值图如图 1所示。从图 1可看出, 4组多重PCR体系的位点均有较好的扩增效率, 并且能清晰地辨别出各位点的等位基因峰值。
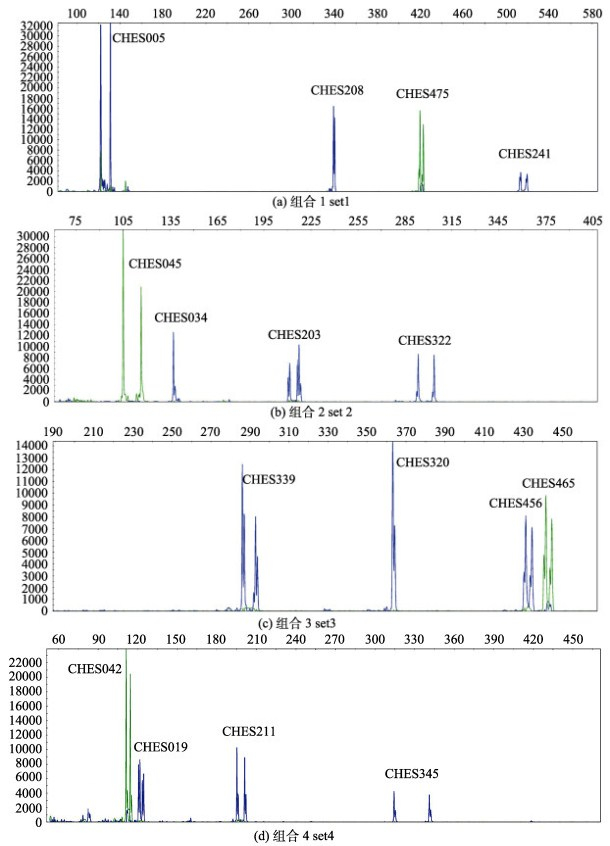
|
图 1 部分中华绒螯蟹个体四组多重PCR的基因扫描图 Fig.1 Part of gene scan results of four sets multiplex PCR in Eriocheir sinensis |
16个微卫星位点在3个家系中的遗传参数如表 2所示。等位基因数(Na)从5至14, 平均等位基因数为9.875;有效等位基因数(Ne)从4.0886到8.3095, 平均有效等位基因数为5.6542;观测杂合度(Ho)和期望杂合度(He)范围分别为0.297~ 0.9697和0.7592~0.8841, 平均观测杂合度为0.8206, 平均期望杂合度为0.8164;香农信息指数(I)为1.5217~2.2378, 平均香农信息指数为1.8650;多态信息含量(PIC)范围为0.7198~0.8686, 平均多态信息含量为0.7927, 各位点均为高度多态性(PIC > 0.5)。
|
|
表 2 微卫星位点在中华绒螯蟹3个家系中的遗传参数 Tab.2 Genetic parameters of the microsatellite loci in three families of Eriocheir sinensis |
中华绒螯蟹3个家系的遗传多样性参数见表 3, 长江家系的平均等位基因数(Na)最高, 但平均期望杂合度(He)和观测杂合度(Ho)稍低于其他两个家系; 香农信息指数(I)体现的群体遗传多样性为辽河家系 > 黄河家系 > 长江家系, 但是差异均不显著(P > 0.05)。
|
|
表 3 中华绒螯蟹3个家系的遗传多样性 Tab.3 Genetic diversity statistics of Eriocheir sinensis for the three families |
通过Cervus 3.0软件进行中华绒螯蟹3个家系亲子鉴定, 置信区间均设置为95%。四组微卫星多重PCR体系分别用于家系亲子鉴定, 结果如表 4所示。仅已知母本基因型时, 四组体系的亲子鉴定正确率分别从73.68%至96.84%;仅已知父本基因型时, 四组体系的亲子鉴定正确率从74.74%至86.32%;当双亲基因型均已知时, 四组体系的鉴定正确率从69.47%至85.26%。如图 2所示, 若双亲基因型均已知, 模拟的亲子鉴定正确率均高于实际的正确鉴定率, 并且可以看出四组微卫星多重PCR体系中组合1和组合4的鉴定效果最好。同时从表 4也可以看出, 将四组多重PCR体系累积进行家系亲子鉴定, 使用两组多重PCR体系时, 亲子鉴定正确率超过94.74%;使用三组微卫星多重PCR体系时, 亲子鉴定正确率超过98.95%, 同时使用四组微卫星多重PCR鉴定时, 正确鉴定率达100%。图 3所示, 根据单组多重PCR的鉴定成功率由高到低组合进行累积鉴定时, 可以看出使用3组多重PCR累积鉴定率可以达到100%。
|
|
表 4 中华绒螯蟹不同组合位点实际的正确鉴定率 Tab.4 The real success assignment rate of different multiplex PCR in Eriocheir sinensis |
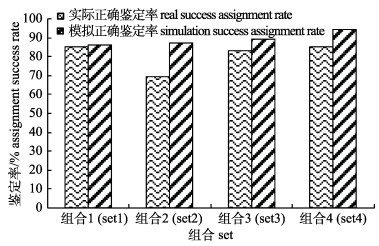
|
图 2 中华绒螯蟹四组微卫星多重PCR体系实际和模拟正确鉴定率 Fig.2 The real success assignment rate and simulation success assignment rate of four set multiplex PCR |
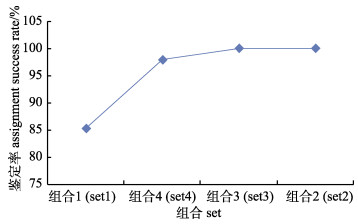
|
图 3 已知中华绒螯蟹双亲基因型时的实际累积鉴定正确率 根据单组微卫星多重PCR的成功鉴定率由高到低添加多重组合. Fig.3 The real cumulative success assignment rate of parents genotypes of known Eriocheir sinensis According to the success assignment rate of a single set of microsatellite multiple PCR, adding multiple combination from high to low. |
在大量样本分析时, 构建微卫星多重PCR体系能够极大地减少基因分型的费用和实验操作的工作量, 虽然前期工作量比较大, 但多重体系技术一旦建立, 后期的工作效率可以提高数倍。但是微卫星多重PCR体系的构建并非简单的PCR组合累加, 而是需要保证在同一反应体系中不同的引物均能高效地特异性扩增, 需要根据目标产物, 进行全面的分析和反复实验, 因此关键技术在于反应引物的组合和条件优化, 技术难度较大[26]。研究表明, 多重PCR体系能否成功构建与引物的退火温度、片段大小、引物的相对浓度等相关[27]。本研究中, 所用微卫星位点均为实验室自主开发的中华绒螯蟹微卫星位点, 并且挑选其中退火温度接近55 ℃的位点, 减少退火温度对扩增的影响, 最终将扩增效率好、多态性高的位点用于多重组合。微卫星多重PCR体系要求同种荧光标记引物之间的片段大小不能重叠, 以免相互干扰, 造成等位基因位点读取误差。本研究中同一体系的相近引物片段间隔均大于50 bp, 并且采用两种不同的荧光标记(FAM和HEX基团)加以区分, 结合毛细管电泳检分型, 相比传统的非变性聚丙烯酰胺凝胶电泳, 能够获得更加丰富和准确的等位基因数据[28]。对于实验中引物相对浓度的控制, 为了避免形成引物二聚体, 并且使各引物均有较好的扩增效率, 我们先用Multiplex Manager 1.0软件进行模拟, 挑选干扰较小的引物组合, 反复进行PCR扩增, 再根据PCR产物分型的结果分别进行引物浓度的调整, 最终获得4组四重PCR, 类似的方法在先前三角帆蚌(Hyriopsis cumingii)[29]和凡纳滨对虾(Litopenaeus vannamei)[30]的微卫星多重PCR研究中均有报道, 具有可行性。
3.2 微卫星多重PCR在虾蟹遗传育种中的应用微卫星标记常被用于中华绒螯蟹遗传多样性评估, 如对不同的地理种群、野生和养殖群体间、奇偶年中华绒螯蟹的评估等[21, 31-32]。从表 2可以看出, 本研究选用的16个微卫星标记在3个养殖家系中的平均观测杂合度(Ho)为0.8206, 平均期望杂合度(He)为0.8164, 高于许志强等[33]对4个天然群体中华绒螯蟹遗传特征分析的平均期望杂合度(He) 0.7411, 同样也高于唐刘秀等[34]对3个中华绒螯蟹育种基础群体遗传特征微卫星分析报道中的平均期望杂合度和平均观测杂合度。同时本研究结果显示, 平均多态信息含量为0.7927, 各位点的PIC值均大于0.5, 因此选用的微卫星标记具有高度多态性、丰富性, 显示出了较高的遗传多样性水平, 符合中华绒螯蟹的亲子鉴定要求。研究表明经过连续多代选育的家系容易出现杂合度降低, 遗传多样性下降的现象[35]。从表 3可以看出, 经过多代的繁殖后, 3个家系的期望杂合度和观测杂合度略低于本课题组先前对中华绒螯蟹2015年养殖群体的60个个体遗传多样性评估的平均观测杂合度0.7500, 平均期望杂合度0.8925[36], 虽然3个家系的遗传多样性有所下降, 但依然保持一个较高值, 这可能与这些家系没有经过高强度的人工选育有关, 也说明课题组选育的中华绒螯蟹群体遗传多样性处于合理水平。
亲权关系分析在品种选育过程中的亲本选留、系谱信息追踪、家系遗传参数评估以及避免近交衰退等方面扮演着重要的角色[37]。本研究采用遗传多样性丰富的4组共16个微卫星位点对3个家系进行亲子鉴定。由图 2可知, 4组微卫星多重PCR组合模拟的正确鉴定率均高于实际的鉴定正确率, 这可能与无效等位基因的存在、基因型统计误差等原因有关[38], 这种现象也出现于黄喉拟水龟(Mauremys mutica)[39]、太平洋牡蛎(Crassostrea gigas)[40]等水产动物的亲子鉴定研究中。由表 3可以看出, 当使用两组的多重PCR组合时(8个微卫星位点), 正确鉴定率超过94.74%;使用4组组合时(16个微卫星位点), 能达到100%的正确鉴定率。在其他虾蟹研究中, 李东宇等[30]通过构建4组凡纳滨对虾微卫星多重PCR组合, 对11个家系进行亲子鉴定, 模拟和亲权鉴定准确率达到100%。孔杰等[41]利用3对微卫星引物构建了中国对虾的三重PCR体系, 并结合荧光标记引物的基因扫描技术(genescan)对中国对虾7个半同胞家系和16个全同胞家系进行识别, 结果能准确定位到各个家系。于洋[42]开发了凡纳滨对虾SSR标记, 并构建了2组微卫星多重PCR体系用于8个家系亲子鉴定, 结果显示194个子代中的189个能够准确鉴定出对应的亲本, 平均鉴定准确率达到98%。研究表明, 微卫星位点的多态性对于亲子鉴定亲本排除概率的效率有很大的影响, 当达到同样的鉴定效果时, 所需多态性高的微卫星位点数量远少于多态性低位点的数量[43], 所以微卫星位点的筛选至关重要。本研究所选用的微卫星位点, 多态信息含量(PIC)范围从0.7198至0.8686, 平均多态信息含量为0.7927, 均为高度多态性的位点, 能达到亲子鉴定的要求。由图 3和表 3可以看出, 使用组合1(CHES005、CHES208、CHES475、CHES241)和组合2(CHES045、CHES034、CHES203、CHES322), 或者组合2和组合3(CHES339、CHES320、CHES456、CHES465)时, 正确鉴定率达到98.95%;选用组合1、2和组合3时, 或者选用组合1、3和组合4时, 累积正确鉴定率达到了100%, 因此表明在一定程度上, 增加微卫星标记的数量可以提高家系鉴定的准确率, 但当微卫星标记达到一定数量后, 鉴定率处于平稳趋势, 即使再增加微卫星标记的数量也不一定能提高鉴定率, 反而增加成本和工作量。Lafarga-de la Cruz等[44]在对杂交鲍的微卫星亲子鉴定研究中也发现, 最少使用5~6个高多态性微卫星位点时依然可以达到9个微卫星标记的亲子鉴定效果。所以, 在中华绒螯蟹亲子鉴定中, 可以选用多重组合1、2和组合3, 或者选用组合1、3和组合4累积进行鉴定, 不但可以获得准确的系谱信息, 还能减少工作量, 降低成本。综上所述, 本研究的4组微卫星多重PCR对于中华绒螯蟹家系的亲子鉴定具有很高的可靠性、识别性。
4 结论本研究构建的4组微卫星多重PCR在中华绒螯蟹家系的亲子鉴定上有较高识别力, 能大大提高分析效率, 为中华绒螯蟹的遗传多样性评估和选择育种, 尤其是家系选育提供便捷、高效的方法。
| [1] |
Department of Agriculture Fisheries Fishery Administration. Chinese Fishery Statistical Yearbook: 2016[M]. Beijing: China Agriculture Press, 2016: 29-30. [农业部渔业渔政管理局. 2016中国渔业统计年鉴[M]. 北京: 中国农业出版社, 2016: 29-30.]
|
| [2] |
He J, Wu X, Li J, et al. Comparison of the culture performance and profitability of wild-caught and captive pond-reared Chinese mitten crab (Eriocheir sinensis) juveniles reared in grow-out ponds: Implications for seed selection and genetic selection programs[J]. Aquaculture, 2014, 434: 48-56. DOI:10.1016/j.aquaculture.2014.07.022 |
| [3] |
Liu Y X, Sun Z H, Wang Y F, et al. Parentage determination in Japanese flounder (paralichthys olivaceus) families by microsatellite DNA marker analysis[J]. Journal of Fisheries of China, 2015, 39(11): 1593-1603. [刘永新, 孙朝徽, 王玉芬, 等. 牙鲆家系亲权鉴定的微卫星DNA标记分析[J]. 水产学报, 2015, 39(11): 1593-1603.] |
| [4] |
Wang H X, Wu C G, Zhang L S, et al. The application of microsatellite markers for parentage determination in selective breeding of Pacific white shrimp (Litopenaeus vannamei)[J]. Hereditas, 2006, 28(2): 179-183. [王鸿霞, 吴长功, 张留所, 等. 微卫星标记应用于凡纳滨对虾家系鉴别的研究[J]. 遗传, 2006, 28(2): 179-183.] |
| [5] |
Waaijenborg S, Verselewel de Wiff Hamer P C, Zwinderman A H. Quantifying the association between gene expressions and DNA-markers by penalized canonical correlation analysis[J]. Statistical Applications in Genetics and Molecular Biology, 2008, 7(1): Article 3. |
| [6] |
Lu X, Wang H, Liu B, et al. Three EST-SSR markers associated with QTL for the growth of the clam Meretrix meretrix, revealed by selective genotyping[J]. Marine Biotechnology, 2013, 15(1): 16-25. DOI:10.1007/s10126-012-9453-4 |
| [7] |
Castro J, Pino A, Hermida M, et al. A microsatellite marker tool for parentage assessment in gilthead seabream (Sparus aurata)[J]. Aquaculture, 2007, 272: S210-S216. DOI:10.1016/j.aquaculture.2007.08.020 |
| [8] |
Jerry D R, Evans B S, Kenway M, et al. Development of a microsatellite DNA parentage marker suite for black tiger shrimp Penaeus monodon[J]. Aquaculture, 2006, 255(1-4): 542-547. DOI:10.1016/j.aquaculture.2005.11.026 |
| [9] |
Neff B D, Fu P, Gross M R. Microsatellite multiplexing in fish[J]. Transactions of the American Fisheries Society, 2000, 129(2): 584-593. DOI:10.1577/1548-8659(2000)129<0584:MMIF>2.0.CO;2 |
| [10] |
Yang Y Q. Rapid diagnosis of systematic fungal infections based on multiplex PCR and microsatellite PCR[D]. Changchun: Jilin University, 2007: 39-70. [杨艳秋.多重PCR及微卫星标记技术用于深部病原真菌感染的快速诊断[D].长春: 吉林大学, 2007: 39-70.] http://cdmd.cnki.com.cn/article/cdmd-10183-2007095751.htm
|
| [11] |
Huang L G, Wang X L, Ou J T, et al. Genetic analysis of 32 microsatellite loci in 13 families of Wuzhishan pig by multiplex PCR and gene scanning technique[J]. Hereditas, 2005, 27(1): 70-74. [黄礼光, 王希龙, 欧江涛, 等. 应用多重PCR和基因扫描技术对五指山猪13个家系32个微卫星基因座的遗传分析[J]. 遗传, 2005, 27(1): 70-74.] |
| [12] |
Leonarduzzi C, Spanu I, Labriola M, et al. Development and characterization of three highly informative EST-SSR multiplexes for Pinus halepensis mill. and their transferability to other Mediterranean pines[J]. Plant Molecular Biology Reporter, 2016, 34(5): 993-1002. DOI:10.1007/s11105-016-0980-4 |
| [13] |
Porta J, Porta J M, Martbínez-Rodríguez G, et al. Development of a microsatellite multiplex PCR for Senegalese sole (Solea senegalensis) and its application to broodstock management[J]. Aquaculture, 2006, 256(1-4): 159-166. DOI:10.1016/j.aquaculture.2006.02.022 |
| [14] |
Ren X Y, Liu P, Li J, et al. Establishment and optimization of a microsatellite-labeling multiplex PCR system in Portunus trituberculatus[J]. Progress in Fishery Sciences, 2011, 32(3): 76-83. [任宪云, 刘萍, 李健, 等. 三疣梭子蟹微卫星多重PCR技术建立及条件的优化[J]. 渔业科学进展, 2011, 32(3): 76-83. DOI:10.3969/j.issn.1000-7075.2011.03.012] |
| [15] |
Lee H J, Lee D H, Yoon S J, et al. Characterization of 20 microsatellite loci by multiplex PCR in swimming crab, Portunus trituberculatus[J]. Genes & Genomics, 2013, 35(1): 77-85. |
| [16] |
Wang Y, Wang X Y, Wang A M, et al. A 16-microsatellite multiplex assay for parentage assignment in the eastern oyster (Crassostrea virginica Gmelin)[J]. Aquaculture, 2010, 308(S1): S28-S33. |
| [17] |
Li Y T, Wongprasert K, Shekhar M, et al. Development of two microsatellite multiplex systems for black tiger shrimp Penaeus monodon and its application in genetic diversity study for two populations[J]. Aquaculture, 2007, 266(1-4): 279-288. DOI:10.1016/j.aquaculture.2007.01.038 |
| [18] |
Li W Y, Wang W J, Kong J, et al. Establishment of the microsatellite quadruple PCR technology for Fenneropenaeus chinense and its application in the evaluation on the effect of releasing simulation[J]. Acta Oceanologica Sinica, 2012, 34(5): 213-220. [李伟亚, 王伟继, 孔杰, 等. 中国对虾微卫星四重PCR技术的建立及其在模拟放流效果评估方面的应用[J]. 海洋学报(中文版), 2012, 34(5): 213-220.] |
| [19] |
Xiong L W, Wang Q, Qiu G F. Large-scale isolation of microsatellites from Chinese mitten crab Eriocheir sinensis via a solexa genomic survey[J]. International Journal of Molecular Sciences, 2012, 13(12): 16333-16345. DOI:10.3390/ijms131216333 |
| [20] |
H nfling B, Weetman D. Characterization of microsatellite loci for the Chinese mitten crab, Eriocheir sinensis[J]. Molecular Ecology Notes, 2003, 3(1): 15-17. DOI:10.1046/j.1471-8286.2003.00336.x |
| [21] |
Liu Q, Liu H, Wu X G, et al. Genetic variation of wild and cultured populations of Chinese mitten crab Eriocheir sinensis from the Yangtze, Huanghe, and Liaohe River basins using microsatellite marker[J]. Oceanologia et Limnologia Sinica, 2015, 46(4): 958-968. [刘青, 刘皓, 吴旭干, 等. 长江、黄河和辽河水系中华绒螯蟹野生和养殖群体遗传变异的微卫星分析[J]. 海洋与湖沼, 2015, 46(4): 958-968.] |
| [22] |
Xiong L W, Li Z, Ma K Y, et al. Analysis of genetic diversity in cultured populations of the Chinese mitten crab (Eriocheir sinensis) by microsatellite markers[J]. Journal of Agricultural Biotechnology, 2012, 20(12): 1441-1448. [熊良伟, 李真, 马克异, 等. 利用微卫星DNA分子标记分析中华绒螯蟹养殖群体遗传分化[J]. 农业生物技术学报, 2012, 20(12): 1441-1448. DOI:10.3969/j.issn.1674-7968.2012.12.011] |
| [23] |
Qiu G F, Xiong L W, Liu Z Q, et al. A first generation microsatellite-based linkage map of the Chinese mitten crab Eriocheir sinensis, and its application in quantitative trait loci (QTL) detection[J]. Aquaculture, 2016, 451: 223-231. DOI:10.1016/j.aquaculture.2015.09.018 |
| [24] |
Liu H, Liu Q, Wu X G, et al. Comparative study on extraction of genome DNA from different tissues of Chinese mitten crab (Eriocheir sinensis) by dissimilar methods[J]. Guangdong Agricultural Sciences, 2014, 41(24): 147-150. [刘皓, 刘青, 吴旭干, 等. 不同提取方法和不同组织对中华绒螯蟹DNA提取效果的比较研究[J]. 广东农业科学, 2014, 41(24): 147-150. DOI:10.3969/j.issn.1004-874X.2014.24.034] |
| [25] |
Holleley C E, Geerts P G. Multiplex Manager 1.0: a cross- platform computer program that plans and optimizes multiplex PCR[J]. BioTechniques, 2009, 46(7): 511-517. DOI:10.2144/000113156 |
| [26] |
Chen M J, Fang T, Ke T, et al. Multiplex PCR—A molecular biotechnique of high efficiency and speed[J]. Journal of Wuhan University of Technology, 2005, 27(10): 33-36. [陈明洁, 方倜, 柯涛, 等. 多重PCR——一种高效快速的分子生物学技术[J]. 武汉理工大学学报, 2005, 27(10): 33-36. DOI:10.3321/j.issn:1671-4431.2005.10.010] |
| [27] |
Rithidech K N, Dunn J J, Gordon C R. Combining multiplex and touchdown PCR to screen murine microsatellite polymorphisms[J]. BioTechniques, 1997, 23(1): 40-44. |
| [28] |
Liu H, Liu Q, Wu X G, et al. Comparison of two detection systems of microsatellite markers in the research of Chinese mitten crabs (Eriocheir sinensis)[J]. Journal of Biology, 2015, 32(1): 90-94. [刘皓, 刘青, 吴旭干, 等. 中华绒螯蟹两种微卫星DNA分型方法的应用比较[J]. 生物学杂志, 2015, 32(1): 90-94. DOI:10.3969/j.issn.2095-1736.2015.01.090] |
| [29] |
Yin H, Bai Z Y, Han X K, et al. The development and application of multiplex PCR panels of microsatellites in Hyriopsis cumingii[J]. Biotechnology Bulletin, 2016, 32(1): 156-162. [殷浩, 白志毅, 韩学凯, 等. 三角帆蚌微卫星多重PCR体系的建立及其应用[J]. 生物技术通报, 2016, 32(1): 156-162.] |
| [30] |
Li D Y, Kong J, Meng X H, et al. Development of multiplex PCR systems of microsatellite markers for Pacific white shrimp (Litopenaeus vannamei) and its application for parentage identification[J]. Progress in Fishery Sciences, 2016, 37(3): 58-67. [李东宇, 孔杰, 孟宪红, 等. 凡纳滨对虾(Litopenaeus vannamei)微卫星多重PCR体系的建立及其在家系亲权鉴定中的应用[J]. 渔业科学进展, 2016, 37(3): 58-67.] |
| [31] |
Pan J L, Mu D K, Hao S, et al. Microsatellite DNA polymorphism analysis of Chinese mitten crab Eriocheir sinensis from two geographic populations[J]. Journal of Nanjing University: Natural Science, 2006, 42(5): 457-462. [潘建林, 牟大凯, 郝莎, 等. 中华绒螯蟹Eriocheir sinensis两个地理种群的微卫星DNA多态性分析[J]. 南京大学学报:自然科学], 2006, 42(5): 457-462.] |
| [32] |
Wang Z Q, Huang S, Mao H C, et al. Genetic differentiation analysis of the even and odd year populations of Chinese mitten crab[J]. Journal of Shanghai Ocean University, 2013, 22(5): 657-664. [王中清, 黄姝, 茅海成, 等. 中华绒螯蟹奇、偶年天然群体的遗传差异分析[J]. 上海海洋大学学报, 2013, 22(5): 657-664.] |
| [33] |
Xu Z Q, Ge J C, Li Y H, et al. The population genetic structure analysis on four stocks of Eriocheir sinensis from different water systems using microsatellite markers[J]. Journal of Nanjing University: Natural Science, 2011, 47(1): 82-90. [许志强, 葛家春, 李跃华, 等. 四水系中华绒螯蟹天然群体遗传特征的微卫星标记分析[J]. 南京大学学报:自然科学, 2011, 47(1): 82-90.] |
| [34] |
Tang L X, Xu Z Q, Ge J C, et al. Microsatellite analysis on the genetic structure of three breeding stocks of Eriocheir Sinensis[J]. Journal of Nanjing Normal University: Natural Science Edition, 2013, 36(2): 84-90. [唐刘秀, 许志强, 葛家春, 等. 中华绒螯蟹3个育种基础群体遗传特征的微卫星分析[J]. 南京师大学报:自然科学版, 2013, 36(2): 84-90.] |
| [35] |
Lind C E, Evans B S, Knaure J, et al. Decreased genetic diversity and a reduced effective population size in cultured silver-lipped pearl oysters (Pinctada maxima)[J]. Aquaculture, 2009, 286(1-2): 12-19. DOI:10.1016/j.aquaculture.2008.09.009 |
| [36] |
Xiao Q Z, Liu Q, Wu X G, et al. Genetic diversity analysis of wild and culture megalopa population of Eriocheir sinensis from Yangtze River[J]. Genomics and Applied Biology, 2017, 36(5): 1935-1945. [肖起珍, 刘青, 吴旭干, 等. 长江水系中华绒螯蟹野生和人工繁殖大眼幼体的遗传多样性分析[J]. 基因组学与应用生物学, 2017, 36(5): 1935-1945.] |
| [37] |
Zhang B, Song W P. Recent progress on microsatellite multiplex PCRs for parentage analysis of aquatic animals[J]. Marine Fisheries, 2012, 34(3): 350-356. [张博, 宋文平. 微卫星多重PCR在水生动物亲权分析中的研究进展[J]. 海洋渔业, 2012, 34(3): 350-356. DOI:10.3969/j.issn.1004-2490.2012.03.016] |
| [38] |
Dakin E E, Avise J C. Microsatellite null alleles in parentage analysis[J]. Heredity, 2004, 93(5): 504-509. DOI:10.1038/sj.hdy.6800545 |
| [39] |
Wen P, Zhao J, Li W, et al. The parentage assignment of Mauremys mutica using multiplex PCR of microsatellites[J]. Acta Hydrobiologica Sinica, 2015, 39(6): 1134-1141. [文萍, 赵建, 李伟, 等. 基于微卫星多重PCR技术的黄喉拟水龟亲子鉴定[J]. 水生生物学报, 2015, 39(6): 1134-1141.] |
| [40] |
Li R H, Li Q, Cornette F, et al. Development of four EST-SSR multiplex PCRs in the Pacific oyster (Crassostrea gigas) and their validation in parentage assignment[J]. Aquaculture, 2010, 310(1-2): 234-239. DOI:10.1016/j.aquaculture.2010.09.037 |
| [41] |
Kong J, Gao H, Yu F, et al. Parentage identification of Chinese shrimp (Fenneropenaeus chinensis) using microsatellite-based triplex PCR genescan technology[J]. Journal of Fishery Sciences of China, 2007, 14(1): 59-66. [孔杰, 高焕, 于飞, 等. 微卫星三重PCR基因扫描技术在中国明对虾家系标识中的应用[J]. 中国水产科学, 2007, 14(1): 59-66. DOI:10.3321/j.issn:1005-8737.2007.01.009] |
| [42] |
Yu Y. Development of molecular markers and their applications in selective breeding of the Pacific white shrimp, Litopeneaus vannamei[D]. Qingdao: University of Chinese Academy of Sciences (Institute of Oceanology), 2014: 45-56. [于洋.凡纳滨对虾分子标记的开发及其在遗传育种中的应用[D].青岛: 中国科学院研究生院(海洋研究所), 2014: 45-56.] http://cdmd.cnki.com.cn/Article/CDMD-80068-1014308951.htm
|
| [43] |
Zhou L, Chu Q, Liu L, et al. Simulation study on paternity identification in dairy cattle with microsatellite and SNP markers[J]. Acta Veterinaria et Zootechnica Sinica, 2011, 42(2): 169-176. [周磊, 初芹, 刘林, 等. 利用微卫星和SNP标记信息进行奶牛亲子鉴定的模拟研究[J]. 畜牧兽医学报, 2011, 42(2): 169-176.] |
| [44] |
Lafarge-de la Cruz F, Aguilar-Espinoza A, Gallardo-Escárate C. Parentage assignment in hybrid abalones (Haliotis rufescens×Haliotis discus hannai) based on microsatellite DNA markers[J]. Aquaculture Research, 2015, 46(1): 216-225. |
 2018, Vol. 25
2018, Vol. 25

