2. 上海海洋大学 水产与生命学院, 上海 201306
2. College of Fisheries and Life, Shanghai Ocean University, Shanghai 201306, China
鲇形目鱼类(Siluriformes), 俗称鲇类(catfishes), 隶属硬骨鱼纲(Osteichthyes)、骨鳔总目(Ostariophysi)的鱼类。鲇形目鱼类是世界经济鱼类的重要组成部分, 其在数量上占了整个淡水鱼类的三分之一左右。中国鲇形目鱼类有11科, 29属, 113种[1], 其中分布最广的是鲇科(Siluridae), 鲿科(Bagridae)次之。在我国鲿科鱼类分布广泛, 种类和数量均较为丰富, 学者将中国鲿科鱼类分为4属30种, 分别为:拟鲿属(Pseudobagrus Bleeker)14种, 黄颡鱼属(Pelteobagrus Bleeker)5种, 鳠属(Mystus Scopoli)4种, 
目前, 在鲇形目中科及其属间的系统发育关系, 仍然是鱼类分类学中关注和研究的热点问题之一[3]。鲇形目中科以及属间的分类依然比较混乱, 各种分类观点相互交织, 争议也相对较多[3-5]。在国内, 杨光等[6]基于核糖体DNA的18S和ITS序列分析8种鲇形目鱼类的系统发育关系; 彭作刚等[7]利用鲇形目鱼类的线粒体Cyt b基因、核DNA的RAG2基因和线粒体DNA全序列, 系统研究了欧亚部分鲇形目鱼类的发育关系, 并对其进行了分化时间推算; 张耀光等[8]及戴凤田等[9]对部分鲿科鱼类的骨骼特征和同工酶方面深入研究, 并以此探讨了鲿科鱼类的系统演化; 有些学者从分子水平对部分鲿科鱼类的系统发育做了研究和分析[4, 10-11]。国外学者从细胞遗传学的角度研究部分鲇形目鱼类, 发现染色体上DNA序列的重复能够突显其染色体的进化[12-14], 这一发现将为鲇形目鱼类的遗传进化及其分类的研究开辟一条新途径。Arce等[15]基于棘鲶科鱼类线粒体的COΙ基因和16S核糖体RNA进行了分子系统发育研究。Sullivan等[16]利用rag1和rag2核基因, 对鲇形目鱼类系统发育关系作了较为详尽的研究。这些新兴的分子分类研究必将为鲇形目鱼类的准确分类提供有效佐证。
线粒体DNA具有较为稳定的母系遗传特点, 其包含的一些基因片段已被广泛应用于物种鉴定和系统发育研究。COΙ (cytochrome oxidase subunit Ⅰ)基因作为线粒体DNA中相对比较保守的基因片段, 被越来越多的国内外学者所关注和研究; 因其具有片段长度适宜(650 bp左右), 易于扩增, 具有丰富的系统发育信号和进化速率适中等优点[17], 而被国内外学者普遍应用到动植物物种分类与鉴定及系统发育研究中。正因COΙ基因片段符合DNA条形码(指生物体内可以代表该物种且具有足够变异的、保守的、易扩增且相对较短的DNA片段)的基本要求, 而被科学家们用来鉴定物种及分析物种间亲缘关系, 验证并整修现有的分类学观点。
本文以线粒体COΙ基因作为分子标记, 对部分鲇形目鱼类进行物种鉴定及系统发育研究。通过构建分子系统发育树, 以探讨鲇形目内部科、属和种分类阶元的演化关系, 并为鲇形目鱼类在分子分类领域填补部分理论依据。
1 材料与方法 1.1 实验材料本研究采集鲇形目鱼类13种, 共134个体样本, 采集标本的鳍条或肌肉组织保存在无水乙醇中, 标本收藏于中国水产科学研究院珠江水产研究所。物种的形态学特征鉴定主要依据《中国淡水鱼类检索》[18]、《中国动物志》[1]和《珠江鱼类志》[19], 并从GenBank下载15种鲇形目鱼类的COI基因序列51条。研究样本信息如表 1所示。
|
|
表 1 样本名录及相关信息 Tab.1 Sampling information and GenBank accession numbers of COΙ genes |
本研究用Omega的组织DNA提取试剂盒(E.Z.N.A.TM Tissue DNA Kit)提取DNA, 提取好的DNA放于-25℃备用。
鱼类COI基因的PCR扩增使用的引物分别为FISH F1/FISH R1和FISH F2/ FISH R2, 是鱼类通用的[20]。
FISH F1: 5‘-TCAACCAACCACAAAGACA TTGGCAC-3‘
FISH R1: 5‘-TAGACTTCTGGGTGGCCAAA GAATCA-3‘
FISH F2: 5‘-TCGACTAATCATAAAGATAT CGGCAC-3‘
FISH R2: 5‘-ACTTCAGGGTGACCGAAGA ATCAGAA-3‘
PCR反应体系为20 μL: dNTP mix 1 μL, 10×Taq buffer 1.5 μL, Taq聚合酶0.5 μL, MgCl2 2 μL, 模板DNA 1 μL, ddH2O 12 μL, 上下游引物各为1.0 μL (浓度为10 μmol/L), 基因组DNA模板总量约为100 ng。PCR扩增条件: 94℃预变性4 min; 然后94℃变性30 s, 55℃退火40 s, 72℃延伸1 min, 此过程运行35个循环; 最后再72℃延伸8 min。用1%琼脂糖电泳检测PCR扩增产物含量。将PCR扩增产物用E.Z.N.A.TM Gel Extraction Kit (Omega Bio-Tek, Guangzhou, China)纯化回收, 并委托广州艾基生物技术有限公司进行测序。
1.3 数据分析先对本研究获得的134条鲇形目鱼类COI基因序列进行拼接, 并与NCBI数据库( https://www.ncbi.nlm.nih.gov/)参考序列进行比对, 用于结果分析的序列对比匹配度必须大于97%, 从而保证形态鉴定的可靠性和下载序列的准确性。并通过MEGA6.0的Kimura’s 2-parameter[21]模型对种间和种内的遗传距离进行计算。用MEGA6.0软件的邻接法(Neighbour-Joining, NJ)和最大释然法(Maximum Likelihood Tree, ML)分别构建分子系统发育树。用DNA sp 5.10统计单倍型[22]。
2 结果分析 2.1 COI基因特征分析本研究测得的134条COΙ基因序列连同从GenBank下载的51条COΙ序列进行比对, 修剪后长度均为536 bp (包含插入缺失位点), 并对其进行分析。结果显示, 排除5个仅有一条COΙ序列的物种, 在研究的180个体中存在61个单倍型, 单倍型平均变异指数为0.441, 核苷酸多样性平均指数为0.00555 (0.00000~0.04198); 23个物种中有3个物种共计5个位点存在碱基插入和缺失现象, 仅占比13.0%。多态性位点数总计为183个, 均值为7.956, 越南隐鳍鲇(Pterocryptis cochinchinensis)多态位点数最多为63个, 六须鲇(Silurus glanis)、新月半鲿(Hemibagrus menoda)、小眼半鲿(Hemibagrus microphthalmus)、狼真


|
|
表 2 23种鲇形目鱼类COΙ基因序列特征 Tab.2 Characters of COΙ sequences from 21 species of Siluriformes |
185个鲇形目鱼类线粒体COΙ基因片段, 长度为536 bp, 平均碱基组成为: A, 25.8%; T, 29.7%; C, 27.3%; G, 17.2%; A+T (55.5%)明显高于C+G (44.5%), 表明鲇形目鱼类COΙ基因碱基组成比例存在较为明显的偏倚性。3个密码子的四种碱基的含量差异较大, 第3个密码子的G+C平均含量高达51.6%, 而第2密码子的仅为38.0%, 第1密码子的为44.0%, 居中。在536 bp排列位点中, 有451个不变位点, 84个变异位点, 其中有51个转换位点和33个颠换位点。第2密码子位点上的转换和颠换位点最多, 转换为40个, 颠换为25个(表 3)。
|
|
表 3 185个鲇形目鱼类COΙ基因序列碱基分布特征 Tab.3 Characters of COΙ sequences from 185 samples of Siluriformes |
通过MEGA6.0的Kimura’s 2-parameter模型对种间和种内的遗传距离进行计算(表 4)。21个物种的种内平均遗传距离为0.006, 除越南隐鳍鲇和加鲁鲱鲇(Clupisoma garua)的种内平均遗传距离分别为0.033和0.045外, 其余种内遗传距离均小于0.020, 有7个物种的种内遗传距离为0。28个物种的种间平均遗传距离为0.195, 是种内遗传距离的32.5倍。其中隶属于鲿科的丝鳍半鲿(Hemibagrus filamentus)与鲇科的越南隐鳍鲇之间的遗传距离最大, 为0.250;斑鳠(Hemibagrus guttatus)和大鳍鳠(Hemibagrus macropterus)之间的种间遗传距离最小, 为0.039。
|
|
表 4 基于K2P模型获得鲇形目28个物种的种间遗传距离及23个物种的种内遗传距离 Tab.4 Genetic distance intraspecific species 23 and intersoecific 28 Siluriformes species based on Kimura's 2-parameter distance |
基于neighbour-joining (NJ)法和maximum likelihood (ML)法分别构建了鲇形目系统发育树(图 1, 图 2)。从这两个图中可以看出, 两种模型获得的发育树大致相似, 但对于部分分类阶元, 分子系统发育关系与传统分类学存在一定的差异。在种水平上, NJ树的分类可信度均达到99%, 而ML树除大鳍鳠的为86%以外, 其余均达到93%以上, 多数达到99%的可信度; 在属级分类水平上与形态分类稍有差异, 可信度分别为99%(NJ)和86.2%(ML), 其中, 本属于半鲿属的丝鳍半鲿并没有与其他半鲿属鱼类聚为一支, 本属于海鲇属的铲吻海鲇并没有与海鲇科的其他物种聚为一支, 而是与鲿科鱼类聚为一支; 科级分类水平与传统分类地位一致性的可信度分别为82.9%(NJ)和36.9%(ML); 目级水平与传统分类一致性的可信度分别为71%(NJ)和63%(ML)。
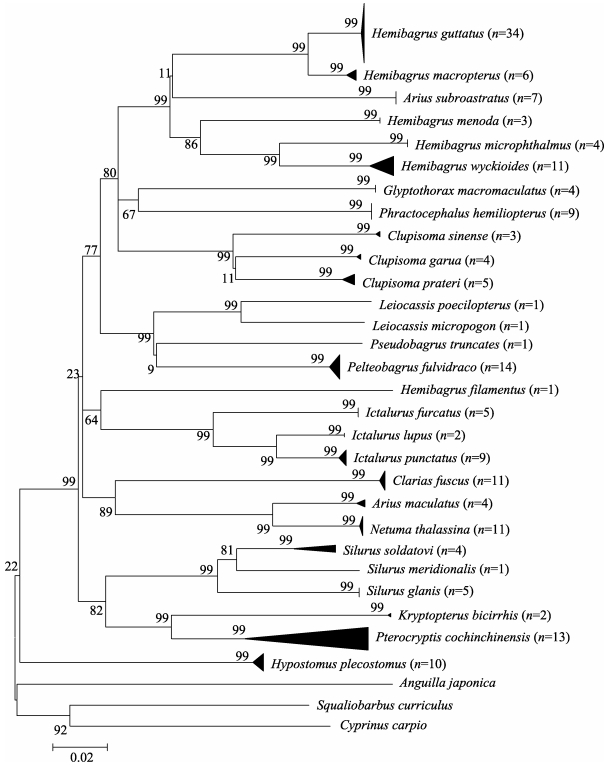
|
图 1 基于COΙ基因序列构建的NJ树 节点上数字显示1000次重复的bootstrap验证分析支持率, 标尺代表遗传距离单位为0.02/百万年. Fig.1 Neighbour-joining tree based on the COΙ gene sequences Bootstrap values in 1000 replications are shown on branches. The scale represents the genetic distance of 0.02/million years. |

|
图 2 基于COΙ基因序列构建的ML树 节点上数字显示1000次重复的bootstrap验证分析支持率, 标尺代表遗传距离单位为0.05/百万年. Fig.2 Maximum likelihood tree based on the COΙ gene sequences Bootstrap values in 1000 replications are shown on branches. The scale represents the genetic distance of 0.05/million years. |
研究结果表明, 部分鲇形目鱼类COΙ基因中存在碱基插入缺失现象, 但插入缺失位点较少; COΙ基因碱基组成存在偏倚现象, 这与曾庆等[5]关于鲇形目鲿科鱼类线粒体全序列研究结果一致; 鲇形目鱼类COΙ基因的平均G+C含量(44.5%)高于Ward等[23]研究得出的软骨鱼类的平均G+C含量, 与其研究结果一致。依托COΙ基因对部分鲇形目鱼类进行系统进化树的构建, 在NJ和ML两种模型下, 所得结果与传统分类一致性差异明显。结果表明, 在NJ模型下, 与传统分类的一致性较高; 种属水平鉴定与传统形态分类一致性较高(99%), 科级水平的一致性也有82.9%, 而在目级水平一致性相对较低, 仅为71%。由此可见, 依托COΙ基因构建NJ树, 对鲇形目鱼类科及其以下分类阶元的系统发育关系研究较为可靠。
3.2 部分鲇形目鱼类系统发育关系分析在鲇形目中, 鲿科和油鲇科的分类一直比较混乱, 本研究中分子系统发育树(NJ)显示, 鲿科鱼类的黄颡鱼属(Pelteobagrus)、拟鲿属(Pseudobagrus)和



油鲇科的红尾护头鲿先与






本研究的结果表明, COΙ基因不仅可以作为鲇形目鱼类有效种鉴定的分子辅助工具, 而且能够应用于其分子系统发育关系的研究。本研究成果是对鲇形目鱼类物种分类信息的有效扩充, 并对传统分类学提供必要的佐证。
| [1] |
Chu X L, Zheng B S, Dai D Y. Faunica Sinica, Osteichthyes, Siluriformes[M]. Beijing: Science Press, 1999. [褚新洛, 郑葆珊, 戴定远. 中国动物志, 硬骨鱼纲, 鲇形目[M]. 北京: 科学出版社, 1999.]
|
| [2] |
Mo T. Anatomy, relationships and systematics of the Bagridae (Teleostei:Siluroidei) with a hypothesis of Siluroid phylogeny[J]. Copeia, 1992, 1992(4): 1132-1134. DOI:10.2307/1446657 |
| [3] |
Grande L. Redescription of hypsidoris farsonensis (Teleostei:Siluriformes), with a reassessment of its phylogenetic relationships[J]. Journal of Vertebrate Paleontology, 1987, 7(1): 24-54. |
| [4] |
Peng Z G, He S P, Zhang Y G. Cytochrome b gene sequence variation and East Asia Bagridae phylogeny[J]. Progress in Natural Science, 2002, 12(6): 596-600. [彭作刚, 何舜平, 张耀光. 细胞色素b基因序列变异与东亚鲿科鱼类系统发育[J]. 自然科学进展, 2002, 12(6): 596-600. DOI:10.3321/j.issn:1002-008X.2002.06.007] |
| [5] |
Zeng Q. Molecular phylogeny and divergence time estimation of the bagrid catfishes (Actinopterygii: Siluriformes)[D]. Chongqing: Southwest University, 2013. [曾庆.鲇形目鲿科鱼类系统发育关系研究及其分化时间估算[D].重庆: 西南大学, 2013.]
|
| [6] |
Yang G, Yu J H, Xu P, et al. Phylogenetic relationship among 8 common species of Catfish based on ribosome 18S and ITS sequences[J]. Chinese Journal of Zoology, 2010, 45(4): 110-117. [杨光, 俞菊华, 徐跑, 等. 利用18S和ITS序列揭示8种鲇形目鱼类的系统发育[J]. 动物学杂志, 2010, 45(4): 110-117.] |
| [7] |
Peng Z G. Phylogenetic relationships of Eurasian catfishes (Otocephala: Siluriformes) and divergence time estimates for major otocephalan clades[D]. Wuhan: Chinese Academy of Sciences (Institute of Hydrobiology), 2005. [彭作刚.欧亚大陆鲇形目鱼类系统发育与骨鳔下区鱼类分化时间估计及其起源与演化过程研究[D].武汉: 中国科学院研究生院(水生生物研究所), 2005.]
|
| [8] |
Zhang Y G, Wang D S. Studies on the osteology of the bagrid catfishes from the Jialing River (Ⅳ):an approach to the phylogenetic relationship[J]. Journal of Southwest China Normal University (Natural Science), 1995, 20(4): 432-439. |
| [9] |
Dai F T, Su J X. Studies on Isozymes, Skeleton characters of eight bagrid catfishes and comments on phylogenetic relationship (Siluriformes:Bagridae)[J]. Acta Zootaxonomica Sinica, 1998, 23(4): 432-439. [戴凤田, 苏锦祥. 鲿科八种鱼类同工酶和骨骼特征分析及系统演化的探讨(鲇形目:鲿科)[J]. 动物分类学报, 1998, 23(4): 432-439. DOI:10.3969/j.issn.1000-0739.1998.04.015] |
| [10] |
Zhang Y, Zhang E, He S P. Studies on the structure of the control region of the Bagridae in China and its phylogentic significance[J]. Acta Hydrobiologica Sinica, 2003, 27(5): 463-467. [张燕, 张鹗, 何舜平. 中国鲿科鱼类线粒体DNA控制区结构及其系统发育分析[J]. 水生生物学报, 2003, 27(5): 463-467. DOI:10.3321/j.issn:1000-3207.2003.05.004] |
| [11] |
Lv F Y, Zuo Y L, Fan Y M, et al. Comparison study of the four species of Bagridae based on mtDNA control region[J]. Journal of South China Normal University (Natural Science Edition), 2008(2): 105-110. [吕凤义, 左艳玲, 范月明, 等. 4种鲿科鱼类基于线粒体DNA控制区序列的比较研究[J]. 华南师范大学学报自然科学版, 2008(2): 105-110.] |
| [12] |
Maneechot N, Yano C F, Bertollo L A, et al. Genomic organization of repetitive DNAs highlights chromosomal evolution in the genus Clarias (Clariidae, Siluriformes)[J]. Molecular Cytogenetics, 2016, 9(1): 4-13. DOI:10.1186/s13039-016-0215-2 |
| [13] |
Supiwong W, Liehr T, Cioffi M B, et al. Chromosomal evolution in naked catfishes (Bagridae, Siluriformes):A compareative chromosome mapping study[J]. Zoologischer Anzeiger-A Journal of Comparative Zoology, 2014, 253(4): 316-320. DOI:10.1016/j.jcz.2014.02.004 |
| [14] |
Kantek D L, Moreira Peres W A, Moreira-Filho O. Cytogenetic study of heptapterids (Teleostei, Siluriformes) with particular respect to the Nemuroglanis subclade[J]. Comparative Cytogenetics, 2015, 9(1): 17-29. DOI:10.3897/CompCytogen.v9i1.8488 |
| [15] |
Arce H M, Reis R E, Geneva A J, et al. Molecular phylogeny of thorny catfishes (Siluriformes:Doradidae)[J]. Molecular Phylogenetics and Evolution, 2013, 67(3): 560-577. DOI:10.1016/j.ympev.2013.02.021 |
| [16] |
Sullivan J P, Lundberg J G, Hardman M. A phylogenetic analysis of the major groups of catfishes (Teleostei:Siluriformes) using rag1 and rag2 nuclear gene sequences[J]. Molecular Phylogenetics and Evolution, 2006, 41(3): 636-662. DOI:10.1016/j.ympev.2006.05.044 |
| [17] |
Hebert P D N, Ratnasingham S, deWaard J R. Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proceedings:Biological Sciences, 2003, 270: S96-S99. |
| [18] |
Zhu S Q. Synopsis of Freshwater Fishes of China[M]. Nanjing: Jiangsu Science and Technology Publishing House, 1995. [朱松泉. 中国淡水鱼类检索[M]. 南京: 江苏科学技术出版社, 1995.]
|
| [19] |
Zheng C Y. Fishes of the Zhujiang River[M]. Beijing: Science Press, 1989. [郑慈英. 珠江鱼类志[M]. 北京: 科学出版社, 1989.]
|
| [20] |
Giusto D, Lera A, Morabito G, et al. The Internet of Things[M]. New York: Springer, 2010.
|
| [21] |
Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. Journal of Molecular Evolution, 1980, 16(2): 111-120. DOI:10.1007/BF01731581 |
| [22] |
Rozas J, Sánchez-Delbarrio J C, Messeguer X, et al. DnaSP, DNA polymorphism analyses by the coalescent and other methods[J]. Bioinformatics, 2003, 19(18): 2496-2497. DOI:10.1093/bioinformatics/btg359 |
| [23] |
Ward R D, Zemlak T S, Innes B H, et al. DNA barcoding Australia's fish species[J]. Philosophical Transactions of the Royal Society of London, 2005, 360(1462): 1847-1857. DOI:10.1098/rstb.2005.1716 |
| [24] |
Liang H W. The complete mitochondrial genomes of six catfishs (Siluriformes) and the phylogenetic relationships analysis[D]. Yangling: Northwest A&F University, 2012. [梁宏伟.六种鲇形目鱼类线粒体基因组克隆及其系统发育研究[D].杨凌: 西北农林科技大学, 2012.]
|
 2018, Vol. 25
2018, Vol. 25


