2. 中国水产科学研究院长江水产研究所, 湖北 武汉 430223;
3. 中国水产科学研究院黑龙江水产研究所, 中国水产科学研究院特殊生境鱼类种质特性与抗逆育种重点实验室, 黑龙江 哈尔滨 150001;
4. 上海海洋大学 水产与生命学院, 上海 201306
2. Yangtze River Fisheries Research Institute, Chinese Academy of Fishery Sciences, Wuhan 430223, China;
3. Heilongjiang River Fisheries Research Institute, Chinese Academy of Fishery Sciences, Harbin 150001, China;
4. College of Fisheries and Life, Shanghai Ocean University, Shanghai 201306, China
鳢科(Channidae)属鲈形目(Perciformes)鱼类, 因其头部形状与蛇头相似而被称为蛇头鱼(snake heads), 主要分布在亚洲和非洲。鳢科鱼类主要有两个属组成:鳢属(Channa)和副鳢属(parachanna)。其中, 副鳢属主要分布在西非中部, 共3种; 鳢属主要分布在亚洲, 共有26种, 西起中东的伊朗, 经南亚、东南亚、中国, 到朝鲜半岛、俄罗斯远东地区都有分布[1]。
我国鳢科鱼类主要有乌鳢(Channa argus)、斑鳢(Channa maculata)、月鳢(Channa asiatica)和南鳢(Channa gachua)[2]。在中国、南亚和东南亚国家, 鳢是重要经济鱼类。其中, 乌鳢、斑鳢及其杂交种是我国重要养殖对象, 年产量达50多万吨[3]。乌鳢在我国分布广泛, 除高原外, 从长江流域往北一直到黑龙江都有分布; 斑鳢由于低温适应能力较差, 主要分布在珠江流域及以南地区。月鳢主要分布在长江流域及以南地区, 包括台湾、海南岛等[4]。Zhang等[4]报道我国广西一鳢科鱼类新物种黑月鳢(Channa nox), 认为其与月鳢相近但存在显著形态差异, 应为独立物种。南鳢分布非常广泛, 在南亚、东南亚和中国都有分布, 世界自然保护联盟(IUCN)认为其为分类地位并未完全厘清, 可能是复合物种, 并将其列入红色保护名录, 其保护级别为LC (Least Concern) (http://www. iucnredlist.org/details/166123/0)。
除我国的土著品种外, 原产南亚和东南亚的多种鳢科鱼类由于体色艳丽, 主要以观赏为目的引进我国, 被称为雷龙鱼。主要有巴卡雷龙(巴卡鳢Channa barca)、黄金眼镜蛇雷龙(橙斑鳢Cha nna aurantimaculata)、蓝宝石雷龙(斯氏鳢Channa stewartii)、七彩雷龙(布氏鳢Channa bleheri)、大铅笔(小盾鳢Channa micropeltes)等。南亚和东南亚鳢科鱼类种类众多, 有些种实际为复合种, 并不断有新种被发现和确认[5-6]。由此可见对鳢科鱼类的分类学研究需要更加深入。
对鳢科鱼类的系统进化研究, 中外学者都有开展相关研究。Zhu等[7]、朱树人等[8]先后对COI和16S rRNA序列对乌鳢、斑鳢和月鳢进行了研究。陈晨等[9]利用COI序列对乌鳢及其他18种鳢进行了进化分析, 认为乌鳢和斑鳢进化关系更近。薛丹等[10]利用线粒体控制区序列分析了云南澜沧江和海南岛水系宽额鳢(南鳢)的遗传变异, 认为云南组群和海南组群间高度分化, 可能受云贵高原隆起和海南岛陆海分离影响。Li等[1]利用NADH脱氢酶亚基2 (ND2)基因进行了20种鳢科鱼类系统进化研究。Lakra等[11]利用COI序列重点对喜马拉雅山东部的鳢科鱼类进行了分析; Conte-Grand等[12]又利用较大数量序列, 进行了更大范围分析, 解决了一些物种误判对系统分类带来的矛盾。
线粒体DNA是独立于核基因之外的母系遗传的遗传物质, 具有进化速率快的特点, 是进行系统进化分析的常用工具之一。其中的细胞色素氧化酶亚基I基因(cytochrome oxidase subunit I gene, COI)相对比较保守, 易于扩增, 在物种鉴定中被广泛使用[13]。被越来越多的国内外学者用于构建DNA条形码数据库[14], 用于物种鉴定和系统发育分析。本研究利用COI基因对我国鳢科鱼类土著种乌鳢、斑鳢、月鳢不同种群样品及其他鳢科鱼类进行分析, 检测其不同群体的多样性, 探讨其用于物种鉴定的可行性。
1 材料与方法 1.1 实验材料本研究采集分布于我国的鳢科鱼类乌鳢、斑鳢、月鳢、南鳢4种及引进观赏鱼小盾鳢样本, 共149个体样本。其中乌鳢样本包括黑龙江(抚远段)27个、湖北长江流域样本13个、湖南湘江样本8个; 斑鳢样本包括珠江流域桂江段、贺江、北江段、三角洲(花都)、茂名鉴江、福建九龙江各10个, 以及海南昌化江和万泉河各1个; 月鳢样本包括长江流域17个、茂名鉴江3个、阳江漠阳河7个; 南鳢为海南昌化江2个(CHJNL1, CHJNL2)、万泉河1个(WQHNL); 小盾鳢样本9个购于广州花地湾观赏鱼市场。从GenBank数据库中下载其他鳢科鱼类COI基因序列122条, 本研究共分析鳢科25种271个样本。另外采集同属攀鲈亚目的攀鲈10个、叉尾斗鱼5个, 鲤形目的鲤和鳙各8个样本作为鳢科鱼类系统进化分析的外群样本。标本的鳍条组织保存于无水乙醇中, 收藏于中国水产科学研究院珠江水产研究所。物种的形态学特征鉴定主要依据《中国淡水鱼类检索》[15]、《广东淡水鱼类志》[2]和《海南岛淡水及河口鱼类志》[16]。研究样本信息如表 1所示。
|
|
表 1 样本名录及相关信息 Tab.1 Sampling information and GenBank accession numbers of COI genes |
首先按照Omega组织DNA提取试剂盒(E.Z.N.A.TM Tissue DNA Kit)操作程序提取样本DNA, 琼脂糖凝胶电泳检测质量后于-20℃冰箱保存。
COI基因扩增使用鱼类通用引物FISH F1/ FISH R1和FISH F2/ FISH R2[17], 引物序列如下:
FISH F1: 5′-TCAACCAACCACAAAGACAT TGGCAC-3′
FISH R1: 5′-TAGACTTCTGGGTGGCCAAA GAATCA-3′
FISH F2: 5′-TCGACTAATCATAAAGATAT CGGCAC-3′
FISH R2: 5′-ACTTCAGGGTGACCGAAGA ATCAGAA-3′
PCR扩增反应体系为20 μL, 包括: dNTP mix (0.25 mmol/L) 1 μL, 10×Taq buffer 2 μL, MgCl2 2 μL, 模板DNA 20 ng, 上下游引物(10 mmol/L)各1.0 μL, 加水补足20 μL。PCR扩增条件: 94℃预变性10 min; 然后94℃变性30 s, 55℃退火30 s, 72℃延伸30 s, 运行35循环; 最后72℃延伸8 min。PCR扩增后用1%琼脂糖电泳检测目标产物, 并用E.Z.N.A.TM Gel Extraction Kit (Omega Bio-Tek, Guangzhou, China)纯化回收, 委托广州艾基生物技术有限公司进行测序。
1.3 数据分析首先对获得的149个鳢和31个外群物种COI基因序列进行拼接比对, 并进一步与NCBI数据库(https://www.ncbi.nlm.nih.gov/)中进行Blast比对, 确保扩增序列的准确性。利用MEGA 6.0软件统计基因序列和相应氨基酸序列的变异, 利用Kimura’s 2-parameter[18]模型对种间和种内的遗传距离进行计算, 用邻接法(Neighbour-Joining, NJ)和最大似然法(Maximum Likelihood Tree, ML)分别构建分子系统进化树。
2 结果分析 2.1 鳢DNA条码COI基因序列多态性本研究获得的149条COI基因序列与从GenBank下载的122条序列进行比对。为使所有序列具有可比性, 将较长序列进行裁剪, 最终长度为576 bp。所有物种中均未出现碱基插入和缺失现象。鳢科鱼类COI序列的碱基A、T、C、G分别为23.8%、27.7%、31.2%和17.4%, C+G含量为48.6%; 271条序列中, 多态性位点数总计272个, 多态位点比例为47.2%, 其中parsim-infor mative位点数为261个, 占45.3%。序列共编码192个氨基酸, 其中多态位点为59个, 多态比例为30.7%。
2.2 鳢种内、种间遗传距离利用MEGA6.0软件的Kimura’s 2-parameter模型对鳢属及外群的种内和种间的遗传距离进行计算(表 2)。25种鳢的种内平均遗传距离为0.028, 其中橙斑鳢、侧眼鳢和拟眼鳢种内遗传距离为0, 巴卡鳢种内遗传距离最大为0.137, 超过了某些种间遗传距离。25种鳢的种间遗传距离为0.030~0.302, 平均为0.217。其中最大距离0.302为饰鳍鳢和黑体鳢之间, 甚至超过了与外群之间的距离。
|
|
表 2 基于K2P模型的鳢属25个物种及4个外群种间(对角线下)及种内遗传距离(对角线内) Tab.2 Genetic distance within-species 23 and between 28 Siluriformes species based on Kimura's 2-parameter distance |
以鲤形目的鲤、鳙和与鳢同属攀鲈亚目的攀鲈和叉尾斗鱼为外群, 基于邻接法(Neighbour-Joining, NJ)和最大似然法(Maximum likelihood, ML)构建的鳢属种间系统进化树(图 1, 图 2)。两种方法构建的进化树中, 多数物种种内个体均以较高支持率聚为一支, 然而巴卡鳢的7个样本分成3个差异极大的类群, 分别与橙斑鳢、斯氏鳢、布氏鳢组成不同的进化支系, 这4种鳢与缅甸鳢组成了一个巴卡鳢复合支系(图 3A); 斯氏鳢的7个样本中4个在巴卡鳢复合支系中, 而另外3个样本与南鳢、红尾鳢和茵列鳢组成南鳢复合支系(图 3B)。基于两种方法构建的进化树中, 作为外群的攀鲈都被归入了鳢属内, 而且与乌鳢、斑鳢、月鳢较近。鳢科鱼类种内个体间聚类支持率均较高, 包括巴卡鳢和南鳢复合支系内, 而种间或姊妹群间相互关系支持率均较低。
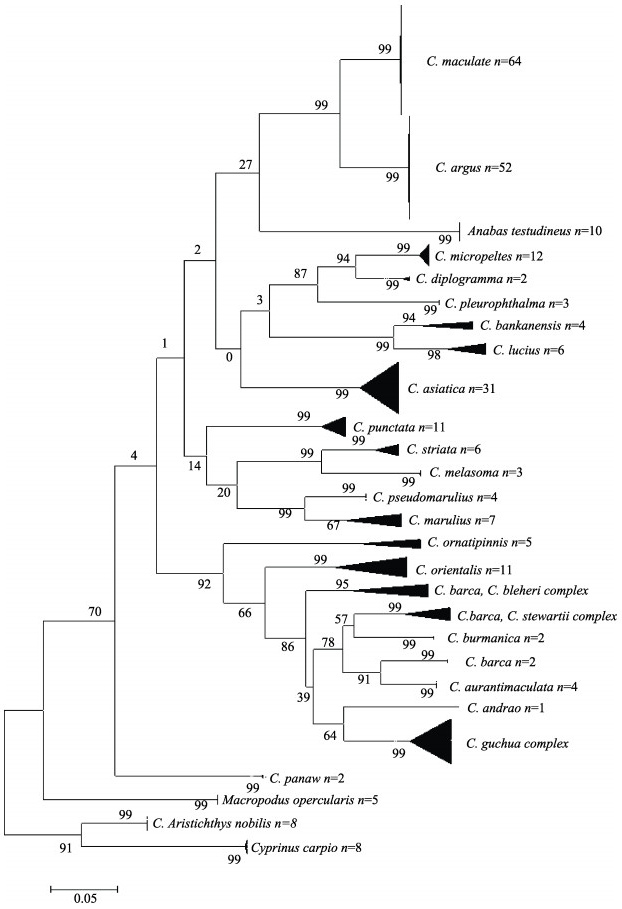
|
图 1 基于COI基因序列构建的鳢属种间NJ进化树 节点上数字显示1000次重复的bootstrap验证分析支持率, 标尺代表遗传距离单位为0.02/百万年. Fig.1 Neighbour-joining tree based on the COI gene sequences of Channa Bootstrap values in 1000 replications are shown on branches. Scale represents the genetic distance of 0.02/million years. |
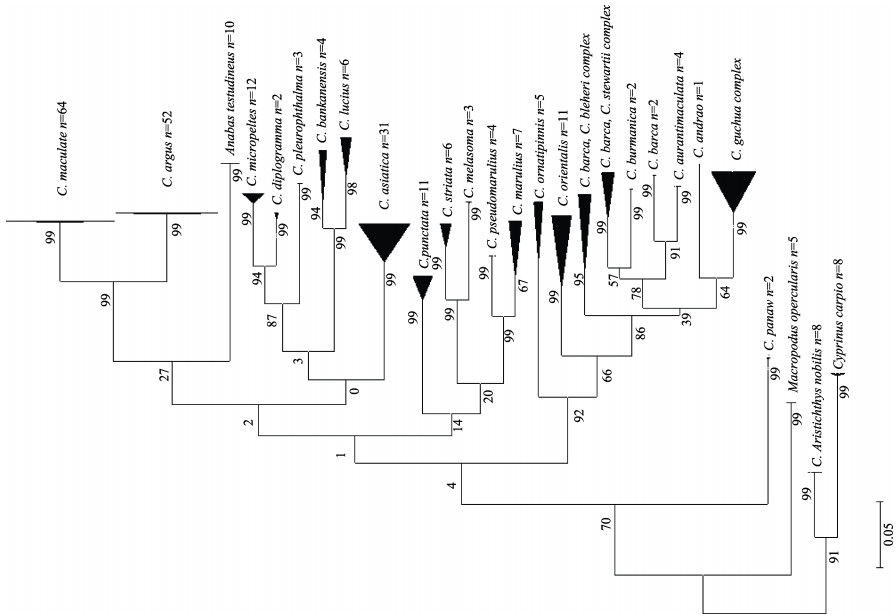
|
图 2 基于COI基因序列构建的鳢属种间ML进化树 节点上数字显示1000次重复的bootstrap验证分析支持率, 标尺代表遗传距离单位为0.05/百万年. Fig.2 Maximum likelihood tree based on the COI gene sequences of Channa Bootstrap values in 1000 replications are shown on branches. Scale represents the genetic distance of 0.05/million years. |
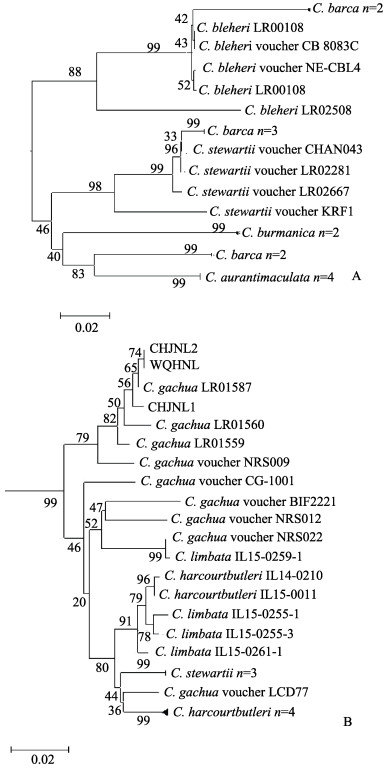
|
图 3 巴卡鳢(A)和南鳢(B)复合支系中物种的进化关系 Fig.3 Phylogenetic relationship of the C. barca and C. gachua complex lineage |
鳢科鱼类的COΙ基因序列GC含量为48.6%, 比AT含量稍少, 与线粒体全序列[19]和
基于Kimura’s 2-parameter模型的种间和种内遗传距离显示, 乌鳢和斑鳢虽在我国分布广泛, 但其种内多样性并不高, 两个物种间的遗传距离也比较小。而其他分布于东南亚和南亚的鳢, 如巴卡鳢、斯氏鳢等种内遗传距离很大, 甚至超过了一些种间的距离。造成这种情况的原因可能是由于物种鉴定的误判[12]。不同鳢种间的遗传距离较大, 有些种间遗传距离(如饰鳍鳢和黑体鳢之间达到0.302)甚至超过了与外群的遗传距离。这说明鳢科鱼类种间COI基因序列遗传多样性较高。
3.2 利用COI基因进行鳢科鱼类的系统进化和物种鉴定利用邻接法和最大似然法分别构建鳢的系统进化树, 其种内个体和部分种间的聚类支持率较高, 而且对巴卡鳢和南鳢复合支系内的支持率也较高, 这与其他同类研究的结果一致[7, 11, 23-26]。而不同种或姊妹群间的支持率较低, 而且两种方法获得的进化树的拓扑结构也存在一定的差异, 这说明利用COI基因不能很好地体现鳢科鱼类的进化关系。
Hebert等[13]提出的物种间遗传变异高于种内变异10倍, 可以作为物种鉴定的DNA条形码标准。Zhu等[7]利用COΙ序列作为DNA条码对乌鳢、斑鳢、月鳢和线鳢进行鉴定, 种间距离最小为0.091, 种内距离最大为0.027, 可以进行准确鉴定。本研究对不同地理种群的乌鳢、斑鳢、月鳢及南鳢进行分析, 其种间距离均显著大于种内距离, 在进化树中聚类支持率很高, 按照以上标准, 我国土著的鳢科鱼类通过COI序列进行物种鉴定是准确的。
由于南鳢和巴卡鳢复合支系的存在, 对相关物种的鉴定造成了影响。Conte-Grand等[12]注意到了鳢科鱼类间的物种鉴定的不一致, 进行了较大规模重新分析和鉴定, 利用较大数据量解决了一些种类的物种鉴定问题, 但对于一些样本较稀少的种类如巴卡鳢等, 数据积累还不够。本研究的结果也表明不同研究人员获得的巴卡鳢样本聚类时分别与橙斑鳢、布氏鳢和斯氏鳢聚为一支, 但它们之间的遗传距离也达到0.13以上, 遗传分化明显。由于这两个复合支系的物种在我国主要以观赏鱼存在, 在对观赏鱼市场采集的样本进行鉴别时, COI序列结果并不准确, 需要根据样本的形态分类特征进行鉴别。另一方面, 由于观赏鱼市场种质引进和保种规范性问题, 其种质可能存在混杂, 进行物种鉴定时需要进行多方面的调查和研究。
| [1] |
Li X, Musikasinthorn P, Kumazawa Y. Molecular phylogenetic analyses of snakeheads (Perciformes:Channidae) using mitochondrial DNA sequences[J]. Ichthyological Research, 2006, 53(2): 148-159. DOI:10.1007/s10228-005-0321-3 |
| [2] |
Pearl River Fisheries Research Institute, Chinese Academy of Fishery Sciences. Freshwater Fishes of Guangdong Province[M]. Guangzhou: Guangdong Sience &Technology Press, 1991. [中国水产科学研究院珠江水产研究所. 广东淡水鱼类志[M]. 广州: 广东科技出版社, 1991.]
|
| [3] |
Administration Bureau of Fisheries and Fishery management of Ministry of Agriculture. Annual Book of China Fishery Statistics 2016[M]. Beijing: China Agriculture Press, 2016. [农业部渔业渔政管理局. 2016中国渔业统计年鉴[M]. 北京: 中国农业出版社, 2016.]
|
| [4] |
Zhang C G, Musikasinthon P, Watanabe K. Channa nox, a new channid fish lacking a pelvic fin from Guangxi, China[J]. Ichthyological Research, 2002, 49(2): 140-146. DOI:10.1007/s102280200018 |
| [5] |
Britz R. Channa andrao, a new species of dwarf snakehead from West Bengal, India (Teleostei:Channidae)[J]. Zootaxa, 2013, 3731: 287-294. DOI:10.11646/zootaxa.3731.2 |
| [6] |
Britz R, Adamson E, Raghavan R, et al. Channa pseudomarulius, a valid species of snakehead from the Western Ghats region of Peninsular India (Teleostei:Channidae), with comments on Ophicephalus grandinosus, O. theophrasti and O. leucopunctatus[J]. Zootaxa, 2017, 4299: 529-545. DOI:10.11646/zootaxa.4299.4 |
| [7] |
Zhu S R, Fu J J, Wang Q, et al. Identification of Channa species using the partial cytochrome c oxidase subunit I (COI) gene as a DNA barcoding marker[J]. Biochemical Systematics and Ecology, 2013, 51: 117-122. DOI:10.1016/j.bse.2013.08.011 |
| [8] |
Zhu S R, Meng Q L, Sun Y X, et al. Phylogenetic analysis of Channa based on the mitochondrial 16S rRNA gene sequence[J]. Journal of Yangtze University (Natural Science Edition), 2015, 12(27): 25-29. [朱树人, 孟庆磊, 孙玉旋, 等. 基于线粒体16S rRNA基因序列的鳢属鱼类系统进化探讨[J]. 长江大学学报(自然科学版), 2015, 12(27): 25-29.] |
| [9] |
Chen C, He J J, Shao Q C, et al. Sequencing and phylogenetic relationship analysis of mitochondrial COI gene[J]. Journal of Jiangsu Agriculture Science, 2016, 44(8): 297-299. [陈晨, 贺晶晶, 邵青臣, 等. 乌鳢(Channa argus)线粒体COI基因序列测定及其系统发育关系分析[J]. 江苏农业科学, 2016, 44(8): 297-299.] |
| [10] |
Xue D, Zhang Q, Gao X C, et al. The analysis of genetic variation based on m tdna control region sequences of Channa gachus in the Lancang River in Yunnan Province and and the main rivers in Hainan Province of China[J]. Acta Hydrobiologyica Sinica, 2015, 39(6): 1107-1116. [薛丹, 章群, 郜星晨, 等. 基于线粒体控制区的云南澜沧江和海南岛主要水系宽额鳢遗传变异分析[J]. 水生生物学报, 2015, 39(6): 1107-1116.] |
| [11] |
Lakra W S, Goswami M. Gopalakrishnan A, et al.Genetic relatedness among fish species of genus Channa using mitochondrial DNA genes[J]. Biochemical Systematics and Ecology, 2010, 38(6): 1212-1219. DOI:10.1016/j.bse.2010.12.012 |
| [12] |
Conte-Grand C, Britz R, Dahanukar N, et al. Barcoding snakeheads (Teleostei, Channidae) revisited:Discovering greater species diversity and resolving perpetuated taxonomic confusions[J]. PLoS ONE, 2017, 12(9): e0184017. DOI:10.1371/journal.pone.0184017 |
| [13] |
Hebert P D, Ratnasingham S, deWaard J R. Barcoding animal life:Cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proceedings:Biological Sciences, 2003, 270: S96-S99. |
| [14] |
Joly S, Davies T J, Archambault A, et al. Ecology in the age of DNA barcoding:The resource, the promise and the challenges ahead[J]. Molecular Ecology Resources, 2014, 14(2): 221-232. DOI:10.1111/1755-0998.12173 |
| [15] |
Zhu S Q. Synopsis of Freshwater Fishes of China[M]. Nanjing: Phoenix Science Press, 1995: 1-549. [朱松泉. 中国淡水鱼类检索[M]. 南京: 江苏科学技术出版社, 1995: 1-549.]
|
| [16] |
Pearl River Fisheries Research Institute of Chinese Academy of Fishery Sciences, Shanghai Fisheries University, East China Sea Fisheries Research Institute of Chinese Academy of Fishery Sciences, et al. Fishes in Freshwater and Estuarine Delta of Hainan Island[M]. Guangzhou: Guangdong Sience & Technology Press, 1986. [中国水产科学研究院珠江水产研究所, 上海水产大学, 中国水产科学研究院东海水产研究所, 等. 海南岛淡水及河口鱼类志[M]. 广州: 广东科技出版社, 1986.]
|
| [17] |
Gong Y Y, Li W, Zhao J, et al. Identification and classification application of mitochondrial COI gene in ornamental fish[J]. Genomics and Applied Biology, 2017, 36(9): 3728-3734. [公月月, 李伟, 赵建, 等. 线粒体COI基因在观赏鱼中物种鉴定与分类应用[J]. 基因组学与应用生物学, 2017, 36(9): 3728-3734.] |
| [18] |
Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. Journal of Molecular Evolution, 1980, 16(2): 111-120. DOI:10.1007/BF01731581 |
| [19] |
Zhang X C, Zhu X P, Chen K C, et al. Comparison of the northern snakehead (Channa argus) and blotched snakehead (Channa maculata) and their reciprocal hybrids (C. maculata ♀×C. argus ♂ and C. argus ♀×C. maculata ♂) based on complete mitochondrial DNA sequences[J]. Mitochondrial DNA, 2015, 26(5): 805-806. DOI:10.3109/19401736.2013.855902 |
| [20] |
Chen L, Huang B D, Xue D, et al. DNA barcoding of Terapontidae and taxonomic status of a new record of Terapon puta in costal waters of China[J]. Marine Fisheries, 2017, 39(2): 121-130. [陈璐, 黄伯炎, 薛丹, 等. 中国沿海鯻科鱼类DNA条形码研究及新记录种三线鯻分类地位探讨[J]. 海洋渔业, 2017, 39(2): 121-130. DOI:10.3969/j.issn.1004-2490.2017.02.001] |
| [21] |
Chen Y X, Wu R X, Liang N, et al. Phylogenetic relationship in family sparidae of China in mitochondrial COI gene sequence[J]. Oceanologia et Limnologia Sinica, 2015, 46(3): 611-619. [陈咏霞, 吴仁协, 梁娜, 等. 基于线粒体COI基因序列的中国鲷科鱼类系统进化关系[J]. 海洋与湖沼, 2015, 46(3): 611-619.] |
| [22] |
Yi X, Wang P P, Wang J, et al. The research of COI-based DNA barcoding in Penaeidaes' identification[J]. Journal of Fisheries of China, 2018, 42(1): 1-9. [易啸, 王攀攀, 王军, 等. 基于线粒体COI的DNA条形码在对虾科种类鉴定中的研究[J]. 水产学报, 2018, 42(1): 1-9.] |
| [23] |
Adamson E A S, Hurwood D A, Mather P B A. Reappraisal of the evolution of Asian snakehead fishes (Pisces, Channidae) using molecular data from multiple genes and fossil calibration[J]. Molecular Phylogenetics and Evolution, 2010, 56(2): 707-717. DOI:10.1016/j.ympev.2010.03.027 |
| [24] |
Benziger A, Philip S, Raghavan R, et al. Unraveling a 146 years old taxonomic puzzle:Validation of Malabar snakehead, species-status and its relevance for channid systematics and evolution[J]. PLoS ONE, 2011, 6(6): e21272. DOI:10.1371/journal.pone.0021272 |
| [25] |
Serrao N R, Steinke D, Hanner R H. Calibrating snakehead diversity with DNA barcodes:expanding taxonomic coverage to enable identification of potential and established invasive species[J]. PLoS ONE, 2014, 9(6): e99546. DOI:10.1371/journal.pone.0099546 |
| [26] |
Tan M P, Jamsari A F J, Muchlisin Z A, et al. Mitochondrial genetic variation and population structure of the striped snakehead, Channa striata, in Malaysia and Sumatra, Indonesia[J]. Biochemical Systematics & Ecology, 2015, 60: 99-105. |
 2018, Vol. 25
2018, Vol. 25

