2. 农业农村部外海渔业开发重点实验室, 广东 广州 510300;
3. 中国水产科学研究院南海水产研究所, 广东 广州 510300;
4. 青岛海洋科学与技术国家实验室, 海洋渔业科学与食物产出过程功能实验室, 山东 青岛 266071;
5. 上海海洋大学 水产与生命学院, 上海 201306
2. Key Laboratory of Open-Sea Fishery Development, Ministry of Agriculture and Rural Affairs, Guangzhou 510300, China;
3. South China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Guangzhou 510300, China;
4. Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071, China;
5. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306, China
绿鳍马面鲀(Thamnaconus septentrionalis)俗称剥皮鱼、马面鱼, 为我国沿海较为常见的经济性鱼类。绿鳍马面鲀为外海暖温性底层鱼类, 栖息于水深50~120 m的海区; 喜集群, 杂食性, 主要摄食桡足类、介形类、端足类等浮游生物, 并兼食软体动物及底栖生物; 在我国主要分布于东海、南海、黄海、渤海和台湾沿海[1]。1974年绿鳍马面鲀成为我国东海的主要捕捞对象之一, 捕捞量在一段时间内仅次于带鱼(Trichiurus lepturus); 然而自20世纪90年代以来, 常年的过度捕捞以及产卵场的破坏致使其产量严重下滑, 东海区域1994年产量仅为3842 t, 资源量始终难以恢复[2-3]。
群体遗传学通过对于遗传多样性、群体间基因流和有效群体大小等遗传学参数的评估, 推断群体遗传结构、种群扩张历史等群体遗传学特征, 在渔业资源管理方面具有重要参考价值。为加强绿鳍马面鲀资源的保护和管理, 有必要对其群体结构、遗传多样性等群体遗传参数进行分析和评估。目前关于绿鳍马面鲀的研究主要集中在生理学、功能基因、摄食习性和养殖特性[4-8]等方面, 群体遗传学方面的研究较少。韩国学者An等[9]基于微卫星标记发现韩国东、西、南部沿海6个绿鳍马面鲀群体间存在着较小但显著的遗传分化。国内尚未见基于DNA分子标记手段的群体结构和遗传多样性研究报道。林新濯等[10]利用标志放流技术研究了东海、黄海、日本海绿鳍马面鲀的洄游和分布情况, 认为3个海区的绿鳍马面鲀为同一种群。丁民权[11]基于1974—1989年江苏省海洋渔业公司在东海绿鳍马面鲀的渔获量, 认为东海范围内存在东海群体、黄海群体和日本海群体3个地方群体。
线粒体DNA具有母系遗传、进化速度快、拷贝数多等特性[12-13], 已广泛应用于群体遗传学及系统发育研究。线粒体细胞色素b (cytochrome b, Cyt b)基因进化速度适中, 部分片段就可包含从种内到种间甚至到科间级别的进化遗传信息[14], 是研究鱼类群体遗传结构、遗传多样性、系统进化关系的理想分子标记[15-18]; 同时, Cyt b可作为DNA条形码, 辅助细胞色素氧化酶亚基Ⅰ基因(COI)进行物种鉴定[19-21]。本研究采集我国沿海6个群体的绿鳍马面鲀样本, 基于Cyt b基因的变异情况分析我国沿海绿鳍马面鲀的遗传多样性和群体遗传结构, 为保护绿鳍马面鲀的渔业资源提供科学依据。
1 材料与方法 1.1 样品采集实验样品于2017年3—5月采集自大连(DL)、秦皇岛(QH)、蓬莱(PL)、日照(RZ)、舟山(ZS)和汕头(ST) 6个地区的近海海域(表 1), 通过联络当地渔船购得。渔船捕获样品后, 放入-20℃冷冻室, 隔日通过邮寄的方式寄回实验室。收到样品后, 取每尾鱼背部肌肉组织保存于95%乙醇, -20℃保存备用。
|
|
表 1 绿鳍马面鲀采样信息 Tab.1 Sampling information of Thamnaconus septentrionalis |
依照天根生化科技(北京)有限公司海洋动物组织基因组DNA提取试剂盒(TIANamp Marine Animals DNA Kit)的使用说明提取DNA。利用NanoDrop Lite超微量分光光度计测量提取DNA的浓度及纯度, 于-20℃存放备用。
依据绿鳍马面鲀线粒体基因组序列[22](GenBank序列号: NC_011327), 利用Oligo7 (Molecular Biology Insights Inc.)软件设计Cyt b基因的引物, 引物由华大基因科技服务有限公司合成。上游扩增引物为: Cytb-F: 5ʹ-GTCATAATTCTCGCCCGGAT-3ʹ;下游扩增引物为: Cytb-R: 5ʹ-GGATTTTAACCCTC GGCCTC-3ʹ。利用北京擎科新业生物技术有限公司2×TSINGKETM Master Mix试剂进行PCR扩增反应, PCR反应体系为20 μL: 2×TSINGKETM Master Mix 10 μL, Cytb-F (10 µmol/L) 0.4 μL, Cytb-F (10 µmol/L) 0.4 μL, DNA模板1 μL, ddH2O 8.2 μL。反应在Bioer LifePro Thermal Cycler (TC- 96/G/H(b)A) PCR仪(杭州博日科技有限公司)上进行, 反应条件为: 94℃预变性5 min; 扩增过程35个循环, 94℃变性30 s, 50℃退火30 s, 72℃延伸60 s; 最终72℃延伸10 min。利用1%琼脂糖凝胶电泳检测扩增产物, 送青岛派森诺基因生物技术有限公司进行PCR扩增产物双向测序, 测序引物为扩增引物。
1.3 序列整理与数据分析使用BioEdit 7.0.1软件观察峰图, 校对数据, 对有杂峰、双峰的样品重新进行扩增测序。利用SeqMan 7.1.0对双向测序结果序列进行拼接。利用ClustalX 2.1[23]软件对获得的序列进行多重比对, 由DnaSP 5.10[24]软件计算变异位点以及单倍型数、各群体的核苷酸多样性(nucleotide diversity, π)、单倍型多样性(Haplotype diversity, Hd)等遗传多样性参数。以MEGA 6.06软件[25]统计序列的平均碱基组成, 计算基于Kimura双参数模型的群体间遗传距离, 自举次数(bootstrap)为1000次。同时采用Mega软件的邻接法(neighbor- joining, NJ)构建绿鳍马面鲀各单倍型间的系统发育关系树, 以黄鳍马面鲀(Thamnaconus hypargyreus)、中华单棘鲀(Monacanthus chinensis)为外类群, 以Kimura双参数法为替代模型, 自举次数(bootstrap) 1000次。利用Network 4.6软件[26]构建单倍型网络图。
借助Arlequin 3.5[27]进行Tajima’s D[28]及Fu’s Fs[29]中性检验, 基于Kimura双参数模型的群体间遗传距离估算群体间的固定指数Fst, 其显著性通过重复1000次抽样法进行检验, 并采用AMOVA分析[30]评估绿鳍马面鲀的群体遗传结构。此外, 进行核苷酸不配对分析并构建各个群体的核苷酸不配对分布图, 分析可以得到5个参数, 即SSD (the sum of squared differences)、Hir (Harpending’s raggedness indices)、θ0、θ1 (种群扩张前后的母系有效种群大小估计值)和τ值(用突变单位表示的种群扩张时间)。将此方法与Tajima’s D、Fu’s Fs相结合推断是否发生过种群扩张。通过公式τ=2ut计算实际的扩张时间, 其中u是所研究的整个序列长度的突变速率(u=kμ, k是所研究的序列长度, μ是每个核苷酸的突变速率), t是自种群扩张开始到现在的世代数。本研究采用1%~2.5%每位点每百万年作为Cyt b基因的进化速率[31-32]。绿鳍马面鲀的性成熟时间为2年, 因而选择2年作为绿鳍马面鲀的世代时间。最终依据T=τ/μk (T为种群扩张发生的时间; k为序列长度; μ为基因进化速率)推算群体发生扩张的年代。通过公式π=2Neμ计算绿鳍马面鲀的有效群体大小(π为核苷酸多样性, Ne为有效群体大小(effective population size), μ为基因进化速率)[33-34]。
2 结果 2.1 序列变异特征测序获得了176尾绿鳍马面鲀的部分Cyt b序列, 序列长度为915 bp。序列已上传GenBank, 登录号为MG845689~MG845864。在6个群体中, 共获得变异位点32个, 变异比例为3.50%。其中, 单一信息位点6个, 简约信息位点26个。变异均为转换和颠换, 无碱基的插入和缺失, 转换明显多于颠换(表 2), 转换与颠换的比值为13.46。碱基组成方面, A、T、C和G的平均频率分别为23.4%、26.5%、33.5%和16.5%, 表现出明显的反G偏倚特点。A+T出现频率为49.9%, C+G为50.1%。
|
|
表 2 绿鳍马面鲀Cyt b基因单倍型变异位点 Tab.2 The variable sites of Cyt b haplotypes in Thamnaconus septentrionalis |
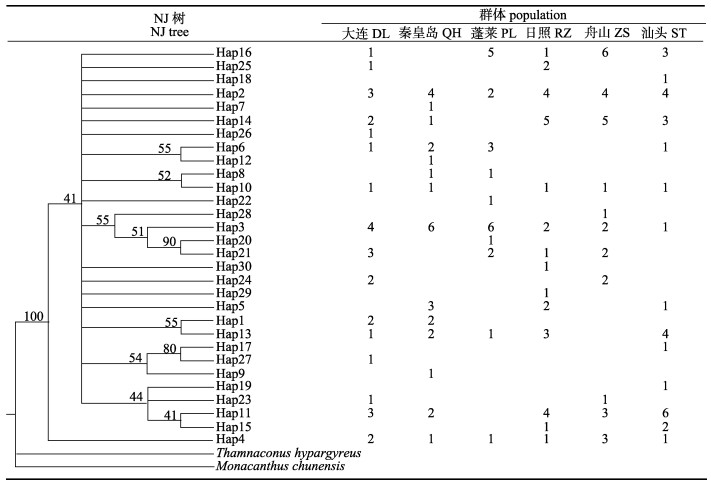
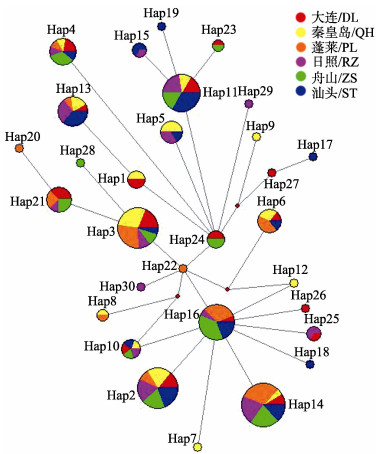
6个群体176个个体共检测到30个单倍型(图 1), 其中, Hap2、Hap3、Hap11和Hap14为优势单倍型, 分别占到个体总数的11.93%、11.93%、10.23%和13.07%。Hap2、Hap3、Hap4和Hap14这4种单倍型为6个群体所共享。Hap7、Hap9、Hap12、Hap17~Hap 20、Hap22、Hap26~Hap 30出现频率较低, 仅在某一群体中出现1次。大连群体拥有的单倍型数最多(16个), 蓬莱和舟山群体的单倍型数最少(11个)。基于检测到的单倍型构建了NJ (neighbor-joining)系统进化树(图 1), 结果显示30个单倍型并未形成明显的谱系分支, 6个群体的单倍型广泛地分布于NJ树的各个分支上, 不存在单倍型与地理群体的明确对应关系。构建的单倍型网络图(图 2)呈现相同的结果, 存在多个主体单倍型, 其他单倍型辐射状分布在主体单倍型周围, 6个群体包含的单倍型交替出现在网络图的各个分支, 并未依据各自群体而形成独立的分支。
|
图 1 绿鳍马面鲀Cyt b基因单倍型NJ系统树及其在6个群体中的分布 Fig.1 Neighbor-joining phylogenetic tree of Cyt b haplotypes and its distribution in 6 populations of Thamnaconus septentrionalis |
|
图 2 绿鳍马面鲀线粒体Cyt b基因单倍型网络图 圆圈面积与单倍型频率成正比. Fig.2 Network diagram of Cyt b haplotype in Thamnaconus septentrionalis The sizes of circles are corresponded to haplotype frequency. |
绿鳍马面鲀6个群体的遗传多样性参数列于表 3。6个群体总体的单倍型多样性较高, 为0.927±0.007, 而核苷酸多样性处于较低水平, 为0.0037±0.0001。各个群体间遗传多样性水平差异较小, 且均显示出高单倍型多样性、低核苷酸多样性的特征。
|
|
表 3 绿鳍马面鲀6个群体的遗传多样性参数 Tab.3 Genetic diversity indices of six populations in Thamnaconus septentrionalis |
由群体间的固定指数(表 4)可知, 大部分群体间的固定指数较小, 并未达到显著水平。蓬莱群体除与舟山群体外, 与其他各群体的固定指数均达到显著水平, 与汕头群体的遗传分化达到极显著水平。虽然蓬莱群体与其他群体(除舟山群体)遗传分化显著, 但固定指数较小(0.034~0.089), 群体间为中低度分化。
|
|
表 4 绿鳍马面鲀6个群体间固定指数Fst Tab.4 The Fst value of six populations in Thamnaconus septentrionalis |
AMOVA分析结果列于表 5。将所有群体归为一个组群进行分析时, 群体间的遗传变异占总变异的1.43%, 群体内的遗传变异占总变异的98.57%, 遗传变异主要来源于群体内, 群体间遗传分化未达到显著水平(Fst=0.014, P > 0.05)。
|
|
表 5 绿鳍马面鲀6个群体分子变异分析(AMOVA) Tab.5 Analysis of molecular variance (AMOVA) of all the 6 populations of Thamnaconus septentrionalis |
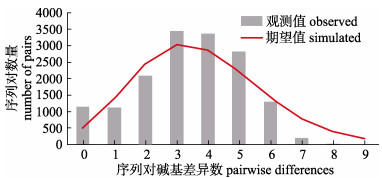
表 6列出了绿鳍马面鲀6个群体的Tajima’s D (D)、Fu’s Fs(Fs)和不配对分布检验所得到的参数值。结果显示, Tajima’s D及Fu’s Fs检验值均为负值, Fs值统计检验达到了显著水平; 不配对分布检验显示Harpending’s指数(Hir)和SSD的统计检验值均未显著偏离种群扩张模型(growth expansion model) (P > 0.05);同时, 在不配对分布图中(图 3), 绿鳍马面鲀6个群体形成了明显的单峰, 并与种群扩张模型下的预期分布相吻合, 以上检验结果说明本研究的绿鳍马面鲀群体历史上经历过种群扩张。基于τ值, 推算群体的扩张时间为42.2万~16.9万年前, 处于中更新世中晚期。由公式π=2Neμ计算绿鳍马面鲀的有效群体大小为74000~185000。
|
|
表 6 绿鳍马面鲀6个群体的Tajima’s D (D)、Fu’s Fs(Fs)和不配对分布检验 Tab.6 Tajima's D (D), Fu's Fs (Fs), mismatch distribution test (SSD), corresponding P-value and the mismatch distribution parameter estimations for the six populations of Thamnaconus septentrionalis |
|
图 3 绿鳍马面鲀6个群体核苷酸不配对分布图 Fig.3 Mismatch distributions for the six populations of Thamnaconus septentrionalis |
物种的遗传多样性是其生存适应和发展进化的前提, 丰富的遗传多样性水平将具有更高的进化潜力及环境适应能力[35]。本研究中6个绿鳍马面鲀群体遗传多样性相接近, Hd均处于较高水平, 而π处于较低水平, 相似的遗传多样性特征还出现在我国沿海的松江鲈(Trachidermus fasciatus)、大泷六线鱼(Hexagrammos otakii)和棘头梅童鱼(Collichthys lucidus)[36-38]等鱼类的研究中。
本研究中绿鳍马面鲀具有高Hd低π的遗传多样性模式, 认为可能存在以下3方面原因: (1)历史动态分析表明绿鳍马面鲀在近期经历了种群扩张, 单倍型数量显著提高, 但尚未积累明显的核苷酸差异; (2) 20世纪60年代东海、黄海主要底层经济鱼类[大黄鱼(Larimichthys crocea)、小黄鱼(Pseudosciaena polyactis)以及带鱼(Thamnaconus lepturus)]资源量衰退, 竞争者的减少使得绿鳍马面鲀的种群数量短期内猛增[39], 有利于产生更多的单倍型; (3)绿鳍马面鲀具有的种群数量大、环境适应性强、性成熟时间短(2年)等特征维持了自然种群内较高的单倍型多样性。
过度捕捞已被证实为多种海水鱼类遗传多样性下降的重要原因, 尤其当鱼类有效群体大小(Ne)较低并且群体遗传结构显著时, 过度捕捞会显著影响其遗传多样性[40]。本研究得到绿鳍马面鲀有效群体大小为74000~185000, 与我国沿海小黄鱼的有效群体大小处于相近水平(26172~261716)[41]。自20世纪70年代至今, 我国沿岸的绿鳍马面鲀经历了近50年的高强度捕捞, 虽然本研究中Hd仍然处于较高水平, 但是不应认为遗传多样性未受到捕捞影响。一方面, 绿鳍马面鲀种群数量较大, 并且不同群体间基因交流频繁, 可以一定程度上抵消或者延迟人类活动对其影响的显现; 另一方面, 本研究中Cyt b基因参考的进化速率为1%~2.5%每位点每百万年, 由捕捞导致的绿鳍马面鲀资源量大幅波动发生在最近几十年, 对于短期的群体数量波动, 基于Cyt b得到的遗传多样性水平有偏高的可能性。因此, 在绿鳍马面鲀渔业保护方面, 仍然需要严格控制捕捞规模, 加强产卵场的保护工作, 防止遗传多样性遭受破坏。
3.2 群体遗传结构和遗传分化黑潮是沿北太平洋西部边缘向北流动的一支强边界流, 流经中国外海海域后向东北方向继续前进, 具有高温、高盐的特征。部分研究发现黑潮对海洋生物群体结构的形成具有促进作用, 黑潮及其分支形成了群体间天然的阻隔[42-43]; 同样有部分研究结果显示黑潮对于营浮游生活生物的受精卵、幼体具有较强的输送作用, 有利于加强不同群体间的基因交流, 降低遗传分化[44-45]。本研究中6个绿鳍马面鲀群体的遗传分化程度较低, 认为黑潮起到了关键作用。绿鳍马面鲀产沉性卵, 受精卵具黏性, 常黏附于海底的海藻、礁石上, 孵化后幼体营浮游生活; 钓鱼岛西北部为东海绿鳍马面鲀重要的产卵场, 多个研究显示绿鳍马面鲀幼体可以借助流经该区域的黑潮向东海外海漂浮北上[11, 46-47], 从而实现东海、黄海和渤海各个群体间的基因交流。另外, 绿鳍马面鲀具有洄游习性, 东海绿鳍马面鲀每年4—5月在钓鱼岛海域产卵后, 成鱼洄游至东海北部及黄海索饵, 冬季进入五岛列岛和对马岛附近海域越冬, 翌年再次南下至钓鱼岛附近产卵[10], 海区范围内如此大规模的洄游同样可以降低各群体间的遗传分化。
3.3 群体历史动态更新世出现了多次冰期与间冰期的交替, 西北太平洋海平面高度涨落频繁, 导致其范围内的海洋生物种群经历明显的收缩和扩张[48]。本研究中绿鳍马面鲀群体在42.2万~16.9万年前发生种群扩张, 处于中更新世中晚期。中更新世这一时期内海平面的剧烈变化可能对绿鳍马面鲀的种群扩张和地理分布格局具有显著影响。
伴随冰期中海平面的下降, 东海、黄海和渤海区域逐渐干涸, 南海和日本海区域仍保有大量水体, 为海洋生物提供了冰期避难所。多个研究显示冰期结束后, 海平面上升, 位于避难所海区的海洋生物群体可以进入到东海、黄海和渤海区域形成新的群体, 进而演化为现有地理分布格局[49-51]。绿鳍马面鲀在南海和日本海范围内均有分布, 由于本研究的采样范围尚未涉及日本海区域, 南海区域仅收集了汕头沿海的样本, 因而不能推断绿鳍马面鲀是否具有上述的种群动态历史。为了更好地解析我国沿海绿鳍马面鲀的群体历史动态, 在未来的研究工作中需要加强这2个区域的样品收集工作。
| [1] |
Su J X, Li C S. Fauna Sinica:Osteichthyes-Tetraodontiformes, Pegasiformes, Gobiesociformes, Lophiiformes[M]. Beijing: Science Press, 2002: 125-132. [苏锦祥, 李春生. 中国动物志:硬骨鱼纲——鲀形目、海蛾鱼目、喉盘鱼目、鮟鱇目[M]. 北京: 科学出版社, 2002: 125-132.]
|
| [2] |
Chen P M, Zhan B Y. Age and growth of Thamnaconus septentrionalis and rational exploitation[J]. Journal of Fishery Sciences of China, 2000, 7(1): 35-40. [陈丕茂, 詹秉义. 绿鳍马面鲀年龄生长与合理利用[J]. 中国水产科学, 2000, 7(1): 35-40. DOI:10.3321/j.issn:1005-8737.2000.01.008] |
| [3] |
Chen W Z, Li C S, Hu F. Application and improvement of virtual population analysis (VPA) in stock assessment of Thamnaconus septentrionalis[J]. Journal of Fisheries of China, 2000, 24(6): 522-526. [陈卫忠, 李长松, 胡芬. 实际种群分析法在绿鳍马面鲀资源评估中的应用和改进[J]. 水产学报, 2000, 24(6): 522-526.] |
| [4] |
Miyajima-Taga Y, Masuda R, Yamashita Y. Feeding capability of black scraper Thamnaconus modestus on giant jellyfish Nemopilema nomurai evaluated through field observations and tank experiments[J]. Environmental Biology of Fishes, 2017, 100(10): 1237-1249. DOI:10.1007/s10641-017-0639-5 |
| [5] |
Lee J A, Lee J S, Kim J H, et al. Relationship between water temperature and oxygen consumption rate of the black scraper Thamnaconus modestus[J]. Ocean and Polar Research, 2014, 36(1): 39-47. DOI:10.4217/OPR.2014.36.1.039 |
| [6] |
Kim A R, Bae H J, Kim H G, et al. Age and growth of filefish, Thamnaconus modestus (Günther, 1877) off the Jeju Island of Korea[J]. Ocean Science Journal, 2016, 51(3): 355-362. DOI:10.1007/s12601-016-0031-y |
| [7] |
Wen H S, Ren Y Y, Zhang D Q, et al. Molecular cloning and expression patterns of the cytochrome CYP17-Ⅰ gene during the reproductive cycle in Navodon septentrionalis[J]. Journal of Fisheries of China, 2014, 38(12): 1945-1955. [温海深, 任源远, 张冬茜, 等. 绿鳍马面鲀CYP17-Ⅰ基因克隆及其在繁殖周期中的表达[J]. 水产学报, 2014, 38(12): 1945-1955.] |
| [8] |
Liu K, Zhang L L, Zhang Q W, et al. Study on Thamnaconus septentrionalis under industrial aquaculture condition[J]. Fishery Modernization, 2017, 44(3): 35-40. [刘琨, 张乐乐, 张庆文, 等. 绿鳍马面鲀工厂化养殖研究[J]. 渔业现代化, 2017, 44(3): 35-40. DOI:10.3969/j.issn.1007-9580.2017.03.006] |
| [9] |
An H S, Lee J W, Park J Y, et al. Genetic structure of the Korean black scraper Thamnaconus modestus inferred from microsatellite marker analysis[J]. Molecular Biology Reports, 2013, 40(5): 3445-3456. DOI:10.1007/s11033-012-2044-7 |
| [10] |
Lin X Z, Gan J B, Zheng Y J, et al. The migration research of Thamnaconus septentrionalis in China[J]. Marine Fisheries, 1984(3): 99-108. [林新濯, 甘金宝, 郑元甲, 等. 绿鳍马面鲀洄游分布的研究[J]. 海洋渔业, 1984(3): 99-108.] |
| [11] |
Ding M Q. On the stocks of filefish Navodon septentrionalis and their distributions in the East China Sea[J]. Journal of Fisheries of China, 1994, 18(1): 45-56. [丁民权. 东海绿鳍马面鲀群体及其分布[J]. 水产学报, 1994, 18(1): 45-56.] |
| [12] |
Brown W M, George Jr M, Wilson A C. Rapid evolution of animal mitochondrial DNA[J]. Proceedings of the National Academy of Sciences of the United States of America, 1979, 76(4): 1967-1971. DOI:10.1073/pnas.76.4.1967 |
| [13] |
Brown W M, Prager E M, Wang A, et al. Mitochondrial DNA sequences of primates:tempo and mode of evolution[J]. Journal of Molecular Evolution, 1982, 18(4): 225-239. DOI:10.1007/BF01734101 |
| [14] |
Kocher T D, Thomas W K, Meyer A, et al. Dynamics of mitochondrial DNA evolution in animals:amplification and sequencing with conserved primers[J]. Proceedings of the National Academy of Sciences of the United States of America, 1989, 86(16): 6196-6200. DOI:10.1073/pnas.86.16.6196 |
| [15] |
Zhang Z S, Hu B J, Ye X Y, et al. Genetic diversity of the pregnant's schizothoracin (schizothorax prenanti) based on partial mtDNA Cyt b sequences[J]. Acta Hydrobiologica Sinica, 2017, 41(3): 609-616. [张争世, 胡冰洁, 叶祥益, 等. 基于mtDNA Cyt b序列分析齐口裂腹鱼群体遗传多样性[J]. 水生生物学报, 2017, 41(3): 609-616.] |
| [16] |
Mcveigh H P, Davidson W S. A salmonid phylogeny inferred from mitochondrial cytochrome b gene sequences[J]. Journal of Fish Biology, 1991, 39((sA)): 277-282. |
| [17] |
Li Y F, Li Y Z, Chen G B, et al. Population analysis of Thamnaconus hypargyreus based on the mitochondrial Cyt b sequence[J]. Journal of Fisheries of China, 2014, 38(1): 23-32. [李玉芳, 李永振, 陈国宝, 等. 基于线粒体Cyt b基因的黄鳍马面鲀种群分析[J]. 水产学报, 2014, 38(1): 23-32.] |
| [18] |
Qi D L, Li T P, Zhao X Q, et al. Mitochondrial cytochrome b sequence variation and phylogenetics of the highly specialized schizothoracine fishes (Teleostei:Cyprinidae) in the Qinghai-Tibet plateau[J]. Biochemical Genetics, 2006, 44(5-6): 270-285. DOI:10.1007/s10528-006-9022-5 |
| [19] |
Page T J, Hughes J M. Comparing the performance of multiple mitochondrial genes in the analysis of Australian freshwater fishes[J]. Journal of Fish Biology, 2010, 77(9): 2093-2122. DOI:10.1111/j.1095-8649.2010.02821.x |
| [20] |
Li N, Wang J H, Han C H, et al. Application of COI and cytb gene in species identification of pufferfish[J]. Chinese Journal of Food Hygiene, 2018, 30(1): 6-11. [李楠, 王佳慧, 韩春卉, 等. COI及cytb基因对河鲀鱼鱼种鉴定的适用性研究[J]. 中国食品卫生杂志, 2018, 30(1): 6-11.] |
| [21] |
Mo B H, Qu L, Han S, et al. DNA barcoding identification Ⅰ. Research progress and applied perspective of DNA barcoding[J]. Sichuan Journal of Zoology, 2008, 27(2): 303-306. [莫帮辉, 屈莉, 韩松, 等. DNA条形码识别Ⅰ. DNA条形码研究进展及应用前景[J]. 四川动物, 2008, 27(2): 303-306.] |
| [22] |
Yamanoue Y, Miya M, Matsuura K, et al. A new perspective on phylogeny and evolution of tetraodontiform fishes (Pisces:Acanthopterygii) based on whole mitochondrial genome sequences:basal ecological diversification?[J]. BMC Evolutionary Biology, 2008, 8(1): 212. DOI:10.1186/1471-2148-8-212 |
| [23] |
Larkin M A, Blackshields G, Brown N, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23(21): 2947-2948. DOI:10.1093/bioinformatics/btm404 |
| [24] |
Librado P, Rozas J. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451-1452. DOI:10.1093/bioinformatics/btp187 |
| [25] |
Tamura K, Stecher G, Peterson D, et al. MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [26] |
Bandelt H J, Forster P, R hl A. Median-joining networks for inferring intraspecific phylogenies[J]. Molecular Biology and Evolution, 1999, 16(1): 37-48. DOI:10.1093/oxfordjournals.molbev.a026036 |
| [27] |
Excoffier L, Lischer H E. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 2010, 10(3): 564-567. DOI:10.1111/men.2010.10.issue-3 |
| [28] |
Tajima F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 1989, 123(3): 585-595. |
| [29] |
Fu Y. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection[J]. Genetics, 1997, 147(2): 915-925. |
| [30] |
Excoffier L, Smouse P E, Quattro J M. Analysis of molecular variance inferred from metric distances among DNA haplotypes:application to human mitochondrial DNA restriction data[J]. Genetics, 1992, 131(2): 479-491. |
| [31] |
Kocher T D, Stepien C A. Molecular Systematics of Fishes[M]. New York: Academic Press, 1997: 113-128.
|
| [32] |
Maggio T, Andaloro F, Hemida F, et al. A molecular analysis of some Eastern Atlantic grouper from the Epinephelus and Mycteroperca genus[J]. Journal of Experimental Marine Biology and Ecology, 2005, 321(1): 83-92. DOI:10.1016/j.jembe.2005.01.004 |
| [33] |
Rogers A R, Harpending H. Population growth makes waves in the distribution of pairwise genetic differences[J]. Molecular Biology and Evolution, 1992, 9(3): 552-569. |
| [34] |
Rogers A R. Genetic evidence for a pleistocene population explosion[J]. Evolution, 1995, 49(4): 608-615. DOI:10.1111/evo.1995.49.issue-4 |
| [35] |
Shen H, Liu D Y. Summary of genetic diversity[J]. Journal of Biology, 2001, 18(3): 5-7. [沈浩, 刘登义. 遗传多样性概述[J]. 生物学杂志, 2001, 18(3): 5-7.] |
| [36] |
Gao T X, Bi X X, Zhao L L, et al. Population genetic structure of roughskin sculpin trachidermus fasciatus based on the mitochondrial Cytb sequence[J]. Acta Hydrobiologica Sinica, 2013, 37(2): 199-207. [高天翔, 毕潇潇, 赵林林, 等. 基于线粒体Cytb基因全序列的松江鲈群体遗传结构分析[J]. 水生生物学报, 2013, 37(2): 199-207.] |
| [37] |
Zhao M, Song W, Ma C Y, et al. Population genetic structure of Collichthys lucidus based on the mitochondrial cytochrome oxidase subunit Ⅰ sequence[J]. Journal of Fishery Sciences of China, 2015, 22(2): 233-242. [赵明, 宋炜, 马春艳, 等. 基于线粒体COI基因序列的棘头梅童鱼7个野生群体遗传结构分析[J]. 中国水产科学, 2015, 22(2): 233-242.] |
| [38] |
Shen Z, Guan H B, Zheng F R, et al. Analysis of genetic diversity between four populations of Hexagrammos otakii based on partial mtDNA cytb and D-loop[J]. Advances in Marine Science, 2017, 35(4): 524-534. [沈朕, 关洪斌, 郑风荣, 等. 基于cytb和D-loop的4个大泷六线鱼群体遗传多样性分析[J]. 海洋科学进展, 2017, 35(4): 524-534. DOI:10.3969/j.issn.1671-6647.2017.04.009] |
| [39] |
Xu X L, Zheng Y J, Liu S. Estimation of stock size of filefish Thamaconus Modestus in the East China Sea and Yellow Sea[J]. Oceanologia et Limnologia Sinica, 1992, 23(6): 651-656. [许学龙, 郑元甲, 刘松. 东、黄海绿鳍马面鲀的资源评估[J]. 海洋与湖沼, 1992, 23(6): 651-656.] |
| [40] |
Pinsky M L, Palumbi S R. Meta-analysis reveals lower genetic diversity in overfished populations[J]. Molecular Ecology, 2014, 23(1): 29-39. |
| [41] |
Zhang B D, Xue D X, Wang J, et al. Development and preliminary evaluation of a genomewide single nucleotide polymorphisms resource generated by RAD-seq for the small yellow croaker (Larimichthys polyactis)[J]. Molecular Ecology Resources, 2015, 16(3): 755-768. |
| [42] |
He L, Takahiko M, Hou C K, et al. Biogeographical role of the Kuroshio Current in the amphibious mudskipper Periophthalmus modestus indicated by mitochondrial DNA data[J]. Scientific Reports, 2015, 5: 15645. DOI:10.1038/srep15645 |
| [43] |
Kuriiwa K, Chiba S N, Motomura H, et al. Phylogeography of Blacktip Grouper, Epinephelus fasciatus (Perciformes:Serranidae), and influence of the Kuroshio Current on cryptic lineages and genetic population structure[J]. Ichthyological Research, 2014, 61(4): 361-374. DOI:10.1007/s10228-014-0408-9 |
| [44] |
Xie S, Watanabe Y. Transport-determined early growth and development of jack mackerel Trachurus japonicus juveniles immigrating into Sagami Bay, Japan[J]. Marine and Freshwater Research, 2007, 58(11): 1048-1055. DOI:10.1071/MF06165 |
| [45] |
Kasai A, Komatsu K, Sassa C, et al. Transport and survival processes of eggs and larvae of jack mackerel Trachurus japonicus in the East China Sea[J]. Fisheries Science, 2008, 74(1): 8-18. DOI:10.1111/fis.2008.74.issue-1 |
| [46] |
Chen L F, Zheng Y J. On the early development, the spawning groud and spawning season of Navodon Septentrionalis (Günther) in the Dong Hai[J]. Acta Ecologica Sinica, 1984, 4(1): 73-79. [陈莲芳, 郑元甲. 东海区绿鳍马面鲀的早期发育和产卵场、产卵期的探讨[J]. 生态学报, 1984, 4(1): 73-79.] |
| [47] |
Fang R S, Zheng Y J. A study on the eddy off Diaoyu Island and its relationship to the fishing ground of filefish (Navodon Septentrionalis Günther)[J]. Journal of Fisheries of China, 1986, 10(2): 161-176. [方瑞生, 郑元甲. 钓鱼岛近海的涡旋及其与马面鲀渔场的关系[J]. 水产学报, 1986, 10(2): 161-176.] |
| [48] |
Hewitt G. The genetic legacy of the Quaternary ice ages[J]. Nature, 2000, 405(6789): 907-913. DOI:10.1038/35016000 |
| [49] |
Wang Z, Zhang Y, Zhao L, et al. Shallow mitochondrial phylogeographical pattern and high levels of genetic connectivity of Thamnaconus hypargyreus in the South China Sea and the East China Sea[J]. Biochemical Systematics and Ecology, 2016, 67: 110-118. DOI:10.1016/j.bse.2016.05.013 |
| [50] |
Yan S, Catanese G, Brown C L, et al. Phylogeographic study on the chub mackerel (Scomber japonicus) in the Northwestern Pacific indicates the late Pleistocene population isolation[J]. Marine Ecology, 2015, 36(3): 753-765. DOI:10.1111/maec.2015.36.issue-3 |
| [51] |
Liu J X, Gao T X, Wu S F, et al. Pleistocene isolation in the Northwestern Pacific marginal seas and limited dispersal in a marine fish, Chelon haematocheilus (Temminck & Schlegel, 1845)[J]. Molecular Ecology, 2007, 16(2): 275-288. |
 2018, Vol. 25
2018, Vol. 25

