2. 海洋渔业科学与食物产出过程功能实验室, 青岛海洋科学与技术国家实验室, 山东 青岛 266273;
3. 水产科学国家级实验教学示范中心, 上海海洋大学, 上海 201306
2. Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Science and Techuology, Qingdao 266273, China;
3. National Demonstration Center for Experimental Fisheries Science Education, Shanghai Ocean University, Shanghai 201306, China
现今地球上估计有1500多万物种, 但已知的生物只有174万种, 其中动物有110多万种。而传统的物种鉴定主要通过形态学、解剖学和生理学等方法, 但由于表型可塑性和遗传变异性, 常导致传统分类无法准确鉴定[1]。2003年, 加拿大生物学家Paul Herbert首次提出了以基因序列作为鉴别不同物种的“条形码”, 即DNA条形码[2-4]。DNA条形码一经提出, 得到了许多学者的肯定, 并在很多物种中得到应用, 在物种鉴定、发现隐存种等方面为传统分类方法提供了科学的辅助数据。细胞色素C氧化酶I (cytochrome coxidase subunit I, CO I)基因, 作为重要的线粒体基因, 具有母系遗传、进化速度快、核苷酸替代率高等特点, 在物种鉴定、种群遗传以及系统发育等研究领域得到广泛应用。作为DNA条形码的发展之初, Herbert等[8]分析了除刺细胞门外, 11个门13320个物种的COI基因序列, 发现其序列间的差异可以很好的区分开所有研究的物种; 而且在物种系统发育过程中, 它的变异速度刚好能使种内差异小于种间差异[9-11], 因此在对大多数物种的研究中采用COI作为DNA条形码的首选基因[12]。
软体动物门是动物界里仅次于节肢动物门的第二大门类, 据不完全统计, 迄今已记载现生贝类超过十万种, 化石种类约3.5万种[13]。收集、保存贝类的实物和信息资源, 是人类认识贝类、合理保护和开发贝类资源的重要基础。软体动物按其体制是否对称, 贝壳、鳃、外套膜和神经等特征的异同进行分类, 但软体动物种类丰富, 许多种形态相似, 且环境对其形态影响较大(表型可塑性), 建立并保存不会随环境的变化而变化的DNA条形码信息对研究其分类地位和系统发育具有重要科学意义。
为进一步丰富COI在贝类分类及鉴定中的科学数据, 本研究通过PCR扩增获得了所收集的353个体贝类(隶属3个纲, 13个目, 33个科, 60个种)的COI基因的部分序列, 分析了其系统发育关系, 以验证COI基因在揭示贝类系统进化关系与传统分类关系的一致性, 同时对同一物种的不同地理群体进行了聚类分析, 为贝类DNA条码数据库的建设提供参考数据。
1 材料与方法 1.1 样品采集实验贝类主要采自于中国沿海, 均通过形态特征进行鉴定, 共426个个体, 属于13个目33个科60个物种。样品采集后, 剪取DNA提取组织并置于酒精中, 与标本材料低温运回实验室, –80℃保存备用。具体物种信息及采样地点如表 1所示。
|
|
表 1 用于分析COI序列的物种及其采样地点 Tab.1 Information on classification and collection of species used for COI sequences analysis |
DNA提取参照有机溶剂抽提法(苯酚氯仿法)[14-15]方法稍作修改。具体是在1.5 mL灭菌离心管中加入400 μL TE抽提液, 无菌解剖刀取50~ 100 mg组织, 装入离心管中并将其剪碎; 每管加入40 μL 10% SDS, 10 μL 20 mg/mL蛋白酶K终浓度400 μg/mL, 充分混匀, 放入55~65℃恒温水浴锅中温浴3 h, 消化至溶液澄清;每管加300 μL 6 mol/L NaCl后, 在旋涡混匀器上快速离心混匀30 s, 12000 rpm离心20 min, 移取上清到另一离心管中;每管加入等体积的酚:氯仿:异戊醇(25:24:1), 缓慢颠倒混匀约8 min, 12000 rpm离心10 min, 将上清液移至另一离心管中, 重复抽提一次;每管加1/10体积的3 mol/L NaCl和2倍体积的无水乙醇, 混匀后置于–20℃中沉淀1 h, 12000 rpm离心15 min后弃上清;加入700 μL 70%乙醇, 缓慢颠倒30 min, 离心10 min, 洗两次;倒掉乙醇后, 在超净台上吹干, 然后加入灭菌水溶解沉淀。利用琼脂糖凝胶电泳检测提取DNA的完整性, NANO DROP 1000分光光度计检测其浓度和纯度。用三蒸水将DNA浓度稀释至50 μg/μL, 置于–20℃保存备用。
PCR扩增在Eppendorf仪器上进行。所用的通用COI-F和COI-R引物[16-17]分别为引物L1490: GGTCAACAAATCATAAAGATATTGG, 以及H2198: TAAACTTCAGGGTGACCA AAAAATCA;PCR反应体系为25 μL, 包括ddH2O 16.8 μL, COI-F 1 μL, COI-R 1 μL, 模板1 μL, Taq Enzyme 0.2 μL, dNTP 2.5 μL, buffer 2.5 μL。反应程序为: 94℃预变性5 min;95℃变性45 s, 55℃退火45 s, 72℃延伸1 min, 35个循环;最后72℃延伸10 min;4℃保存。PCR产物用1%的琼脂糖凝胶电泳检测。
1.3 数据分析将PCR产物送至上海生工有限公司进行测序, 序列拼接后, NCBI人工比对, 验证测序准确性。将每个物种随机选取一条序列, 通过MEGA 6.0截取了275 bp的同源序列, 采用基于距离的矩阵法构建了NJ进化树。为研究不同地理群体的遗传差异, 对不同地理群体的序列截取其同源序列并构建了NJ进化树。
2 实验结果 2.1 引物通用性本研究共利用COI通用引物L1490和F2198扩增60个物种426个个体, 共获得了353条序列, 有效扩增效率为82.7%。其中帘蛤目、蚶目、海螂目和贻贝目各物种的扩增效率较高, 达到90%以上;原始腹足目扩增效率较低, 仅有50%;在珍珠贝目中基本无法扩增获得片段, 只有10%。总体上, COI通用引物的扩增效率较高, 能够适用于多数贝类种类的扩增。
2.2 聚类进化关系如图 1所示, 由进化树可以看出利用COI基因序列进行的聚类关系基本与传统的分类一致。首先, 除沼蛤、大竹蛏(Solen grandis)、长竹蛏(Solen strictus)、魁蚶、缢蛏(Sinonovacula constricta)、栉孔扇贝(Chlamys farreri)、虾夷扇贝(Patinopecten yessoensis)和太平洋潜泥蛤外, 双壳纲各物种聚为一支;其次, 腹足纲下的新腹足目、中腹足目和原始腹足目被分裂开, 存在交叉现象, 未能明显区分各目间遗传距离差异, 整个纲聚为两个分支, 而且八腕目与蛾螺科的褐管蛾螺、略胀管蛾螺聚为一支;除大竹蛏、长竹蛏和缢蛏外, 其他帘蛤目的物种聚为一支;最为特别的是太平洋潜泥蛤, 其传统个体分类为双壳纲海螂目的一种, 但其与中腹足目的斑玉螺聚为一支。
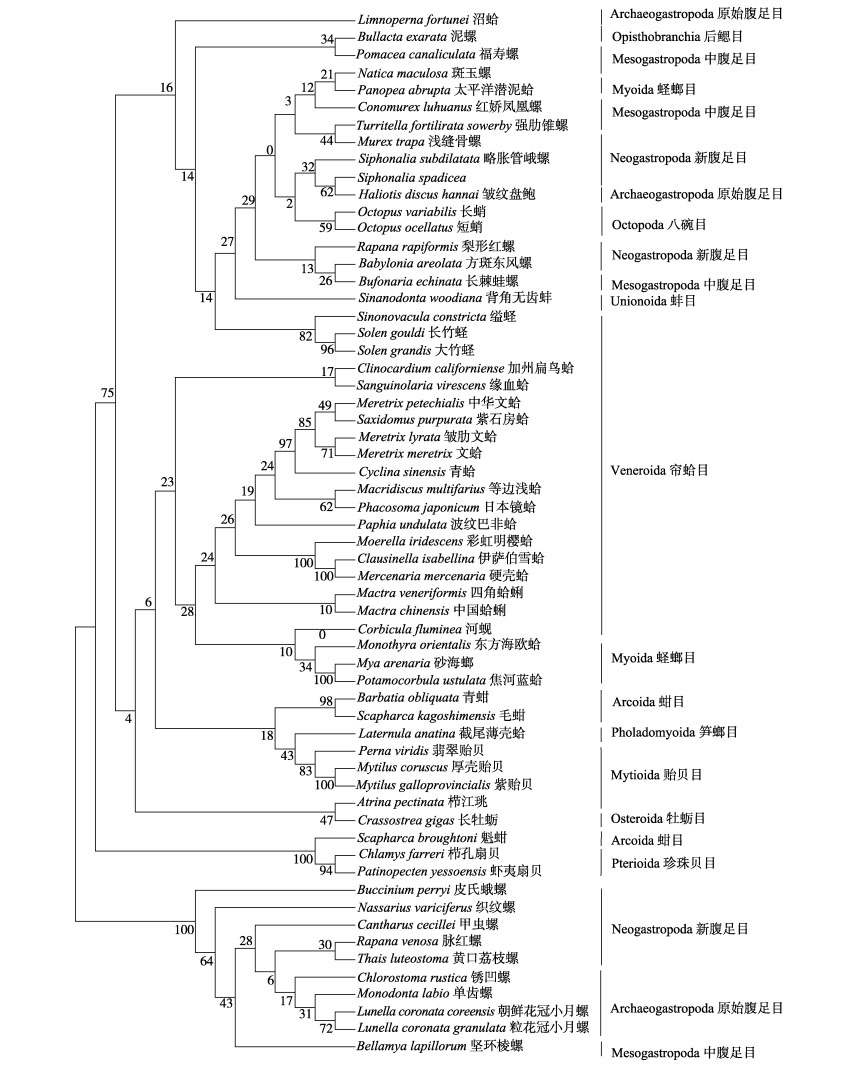
|
图 1 基于60种贝类COI序列构建的进化树 Fig.1 The phylogenetic tree of COI sequences from 60 species in molluscs |
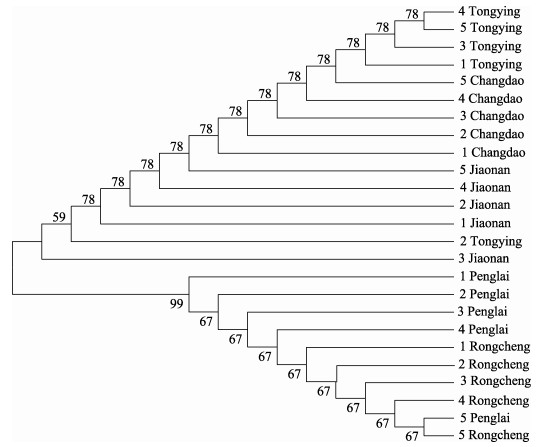
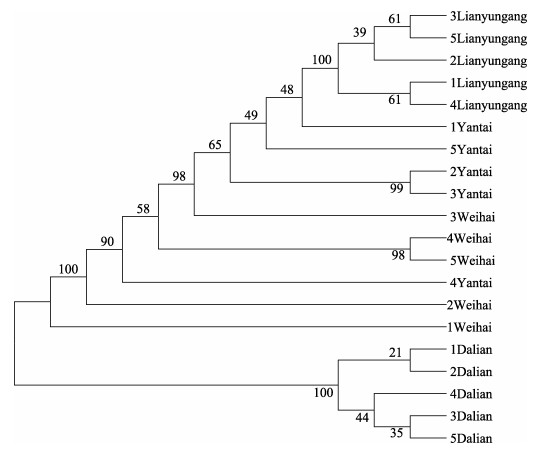
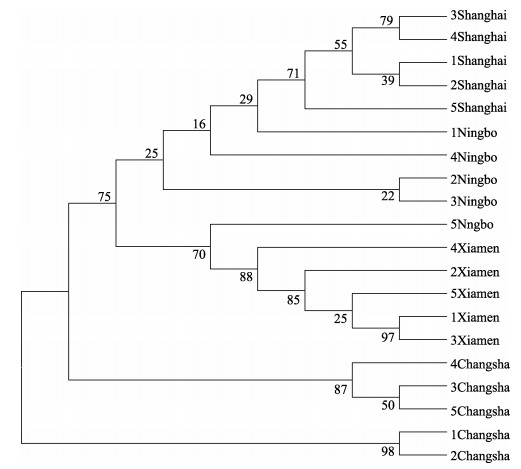
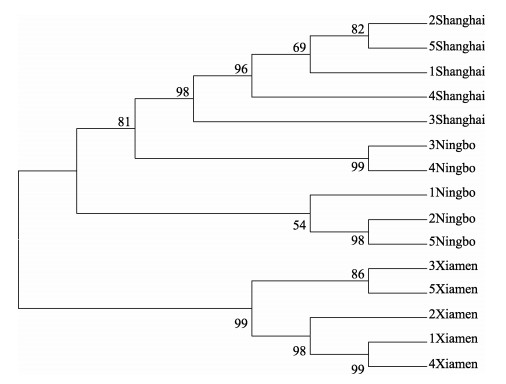
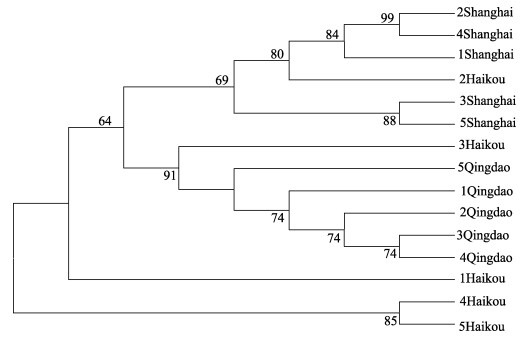
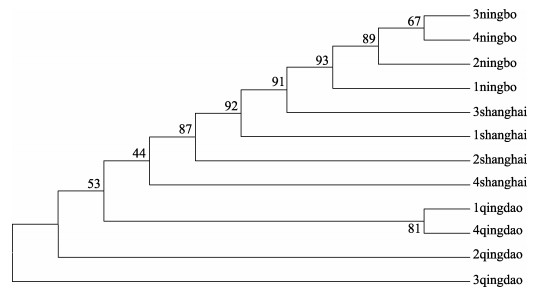
选取了不同地理群体的7种贝类, 通过聚类关系研究了其进化关系。物种名称和采集地点如表 2所示。通过MEGA 6.0构建了NJ进化树, 图 2~图 8分别为方斑东风螺(Babylonia areolata)、魁蚶、毛蚶(Scapharca kagoshimensis)、青蚶(Barbatia obliquata)、厚壳贻贝(Mytilus coruscus)、缢蛏、青蛤(Cyclina sinensis)不同地理群体聚类图。方斑东风螺全部个体主要聚为两个分支, 上海群体单独聚为一支, 厦门和海口群体整体聚为一支, 其内存在群体交叉现象;魁蚶蓬莱和荣成群体聚为一支, 其中5号蓬莱个体和5号荣成个体聚为一支, 其他蓬莱和荣成个体聚类, 统营、长岛和胶南群体聚为一支, 除2号统营个体外, 其余群体聚在一起;毛蚶大连群体单独聚为一支, 连云港、烟台和威海群体聚为一支, 除4号烟台个体外, 首先连云港群体聚为一支, 再与烟台群体聚在一起, 最后与威海群体聚在一起;青蚶上海和厦门群体聚为两支, 宁波群体的个体分别与上海和厦门群体聚在一起, 最后和长沙群体相聚;厚壳贻贝厦门群体单独聚为一支, 上海群体先聚为一支, 再与宁波群体聚在一起;缢蛏青岛和上海群体分别聚为两支, 海口群体的2号个体与上海群体聚为一支, 3号个体与青岛群体聚为一支。青蛤宁波群体聚为一支, 再与上海群体相聚, 最后与青岛群体相聚。所有这些结果表明, COI基因基本能对同一物种不同地理群体进行聚类。
|
|
表 2 用于分析不同地理群体遗传距离的物种名和采集地点 Tab.2 Species used to analyze genetic distance of different geographical populations and its extracted. |
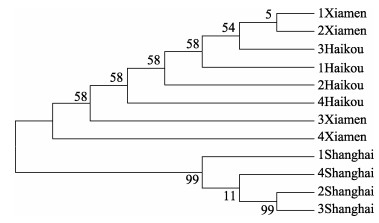
|
图 2 方斑东风螺不同地理群体构建的进化树 1、2、3和4表示不同地理群体中个体的编号. Fig.2 The phylogenetic tree of Babylonia areolata from different geographical populations 1, 2, 3 and 4 indicate the numbers of individuals in different geographical groups. |
|
图 3 魁蚶不同地理群体构建的进化树 1、2、3、4和5表示不同地理群体中个体的编号. Fig.3 The phylogenetic tree of Scapharca broughtoni from different geographical populations 1, 2, 3, 4 and 5 indicate the numbers of individuals in different geographical groups. |
|
图 4 毛蚶不同地理群体构建的进化树 1、2、3和4表示不同地理群体中个体的编号. Fig.4 The phylogenetic tree of Scapharca kagoshimensis from different geographical populations. 1, 2, 3 and 4 indicate the numbers of individuals in different geographical groups. |
|
图 5 青蚶不同地理群体构建的进化树 1、2、3、4和5表示不同地理群体中个体的编号. Fig.5 The phylogenetic tree of Barbatia obliquata from different geographical populations. 1, 2, 3, 4 and 5 indicate the numbers of individuals in different geographical groups. |
|
图 6 厚壳贻贝不同地理群体构建的进化树 1、2、3、4和5表示不同地理群体中个体的编号. Fig.6 The phylogenetic tree of Mytilus coruscus from different geographical populations 1, 2, 3, 4 and 5 indicate the numbers of individuals in different geographical groups. |
|
图 7 缢蛏不同地理群体构建的进化树 1、2、3、4和5表示不同地理群体中个体的编号. Fig.7 The phylogenetic tree of Sinonovacula constricta from different geographical populations 1, 2, 3, 4 and 5 indicate the numbers of individuals in different geographical groups. |
|
图 8 青蛤不同地理群体构建的进化树 1、2、3和4表示不同地理群体中个体的编号. Fig.8 The phylogenetic tree of Cyclina sinensis from different geographical populations 1, 2, 3 and 4 indicate the numbers of individuals in different geographical groups. |
遗传多样性(genetic diversity)是生物所携带的遗传信息的总和, 是生态多样性和物种多样性的基础, 而物种多样性也就显示了基因遗传的多样性, 基因遗传多样性代表生物种群之内和种群之间的遗传结构的变异[18-19]。DNA碱基序列分析可以准确的检测出遗传结构的变异(碱基互换、插入和缺失等)信息, 可以准确地揭示种群间的进化关系, 更准确地鉴别不同个体[20-21]。DNA条形码就是使用一段短DNA标准序列来实现物种鉴别的, 线粒体基因因为没有内含子, 不会发生等位基因重组现象, 其遗传信息完全来自母本, 因此比核基因更具优势, 所以对动物而言, 一般采用线粒体细胞色素C氧化酶I作为DNA条形码标准基因。据已有的报道, COI基因序列对龙虾属[22]和文蛤属[24]等的分类鉴定具有一定的可行性。
由于贝类鉴别难度大, 而且贝类幼体较小, 发育过程中存在变态现象, 更难用传统方法鉴定其物种信息, 而条形码DNA序列分析能更好地解决这个问题。通过对上述60个物种的COI基因序列的总结研究, 发现COI基因对贝类通用性较好, 扩增效率高, 方便各种贝类COI基因序列的获得, 这为贝类物种的鉴定提供了先提条件;其次进化树中大多数物种聚类关系与传统分类一致, 虽然腹足纲同目之间未能明显聚类, 但这可能是由于整个研究物种跨度较大, 导致截取的同源片段较少, 从而导致其中包含的遗传信息有限, 不足以区分所有的物种, 或由于地理环境的不同, 导致同一物种在表观形态上有一定的差异, 使原始分类鉴定时不够准确, 太平洋潜泥蛤未能明显聚类, 原因还需要进一步研究。由不同地理群体聚类分析, 同一地理群体基本能够聚在一起, 之后再与其他群体聚类, 这可能由于长期的地理隔离, 缺少基因交流, 从而产生了一定的基因差异。但有的地理群体间存在群体交叉现象, 此现象的原因可能为受到人为因素的干扰或距离较近未存在明显的遗传分化, 如进口外来苗种培育时产生的幼苗逃逸而产生外来物种的污染, 或在自然海区中存在基因交流。本研究推测魁蚶的胶南和统营群体存在交叉互换, 可能由于该地魁蚶育种时所选的种贝同为韩国进口品种, 而毛蚶的烟台威海群体存在群体交叉就可能由于两地距离较近未产生明显分化。上述情况出现的原因还需要进一步探究, 虽然COI基因在鉴别同一物种不同地理群体间有一定的作用, 但尚不能作为鉴别不同地理群体的依据。
对于COI基因的探索和分析已经有了很多的报道, 如孟学平等[25]发现COI基因可以鉴定福建西施舌, 但不能鉴定其余群体;张迪等[26]发现COI基因适于银鱼科鱼类物种鉴别和系统发育研究。但物种鉴定中的很多方面还处于初级阶段, 仍有许多后续深入研究等待开展。一个基因序列没有变化在分类鉴别上是没有意义的, 而快速变异的基因序列虽然在鉴定近源物种具有一定的优势, 但其由于碱基的突变导致不能区分远源物种, COI序列片段包含一定的变异速度相对较快的系统发育信息, 因而可以用以探讨属级以下的系统发育关系, 对低阶元的系统发育研究具有重要意义。一个位点有4种碱基组成, 只需要14 bp的序列就可以产生2.68亿种组合方式, 虽然有些位点因为物种进化选择压力将会保持稳定, 但适当延长DNA片段长度便足以鉴别大多数物种。本研究需要进一步的总结分析, 发现其中的规律和特殊性, 为以后鉴别动物物种的种类和系统发育规律提供一定的依据, 从而探索更多的隐存种, 了解更多的遗传多样性。
| [1] |
Tautz D, Arctander P, Minelli A, et al. DNA points the way ahead in taxonomy[J]. Nature, 2002, 418(6897): 472-479. DOI:10.1038/418472a |
| [2] |
Hebert P D N, Stoeckle M Y, Zemlak T S, et al. Identification of birds through DNA barcodes[J]. PLoS Biology, 2004, 2(10): 1657-1663. |
| [3] |
Ward R D, Hanner R, Hebert P D N, et al. The campaign to DNA barcode all fishes[J]. Fish Biology, 2009, 74(2): 329-356. DOI:10.1111/jfb.2009.74.issue-2 |
| [4] |
Hebert P D N, Ratnas S, Waard J R, et al. Barcoding animal life:cytochrome coxidase subunit divergences among closely related species[J]. Philosophical Transactions-Royal Society, 2003, 270(1): 96-99. |
| [5] |
Peng J L, Wang X Z, He S P, et al. The progress and application of DNA barcoding[J]. Acta Hydrobiologica Since, 2008, 32(6): 916-919. [彭居俐, 王绪帧, 何舜平. DNA条形码技术的研究进展及其应用[J]. 水生生物学报, 2008, 32(6): 916-919.] |
| [6] |
Chen J, Li Q, Kong L F, et al. CO I-based DNA barcoding in Tapetinae species (Mollusca, Bivalvia, Veneridae) along the coast of China[J]. Zoological Research, 2010, 31(4): 345-352. [陈军, 李琪, 孔令锋, 等. 基于CO I序列的DNA条形码在中国沿海缀锦蛤亚科贝类中的应用分析[J]. 动物学研究, 2010, 31(4): 345-352.] |
| [7] |
Wang L N, Yan X W, Qin Y J, et al. Research on the development of 16 kinds of economic shellfish DNA barcodes and molecular systems in China[J]. Journal of Dalian Fisheries University, 2013, 28(5): 431-437. [王琳楠, 闫喜武, 秦艳杰, 等. 中国帘蛤目16种经济贝类DNA条形码及分子系统发育的研究[J]. 大连海洋大学学报, 2013, 28(5): 431-437. DOI:10.3969/j.issn.2095-1388.2013.05.004] |
| [8] |
Hou Y, Tian J T, Liu Z H, et al. Genetic diversity of the population of Scapharca subcrenata based on mitochondrial CO I gene[J]. Marine Science, 2016, 40(1): 1-9. [侯丫, 田吉腾, 刘志鸿, 等. 基于线粒体CO I基因的毛蚶群体遗传多样性[J]. 海洋科学, 2016, 40(1): 1-9. DOI:10.3969/j.issn.1671-6647.2016.01.001] |
| [9] |
Chen A H, Li Z X, Feng G N. Phylogenetic relationships of the genus Meretrix (Mollusca:Veneridae) based on mitochondrial COI gene sequences[J]. Zoological Research, 2009, 30(3): 233-239. DOI:10.3724/SP.J.1141.2009.03233 |
| [10] |
Zheng W J, Zhu S H, Shen X Q, et al. Genetic differentiation of Tegillarca granosa based on mitochondrial COI gene sequences[J]. Zoological Research, 2009, 30(1): 17-23. [郑文娟, 朱世华, 沈锡权, 等. 基于线粒体COI基因序列探讨泥蚶的遗传分化[J]. 动物学研究, 2009, 30(1): 17-23.] |
| [11] |
Meng W, Yang T Y, Hai S, et al. Study of DNA barcoding based on the mitochondrial COI gene sequences in Salmo trutta fraio[J]. Chinese Journal of Fisheries, 2010, 23(1): 6-10. [孟玮, 杨天燕, 海萨, 等. 基于线粒体COI基因序列的亚东鲑DNA条形码研究[J]. 水产学杂志, 2010, 23(1): 6-10. DOI:10.3969/j.issn.1005-3832.2010.01.002] |
| [12] |
Li Q, Zou S M, Zheng X D, et al. DNA barcode and its application in Marine life[J]. Journal of Ocean University of China (Natural Science Edition), 2010, 40(8): 43-47. [李琪, 邹山梅, 郑小东, 等. DNA条形码及其在海洋生物中的应用[J]. 中国海洋大学学报(自然科学版), 2010, 40(8): 43-47.] |
| [13] |
Ball S L, Armstrong K F. DNA barcodes for insect pest identification:a test case of tussock moths (Lepidoptera:Lymantriidae)[J]. Canadian Journal of Forest Research, 2006, 36(2): 337-350. DOI:10.1139/x05-276 |
| [14] |
Yang X Q, Ye Q T, Xin T R, et al. Population genetic structure of Cheyletus malaccensis (Acari:Cheyletidae) in China based on mitochondrial COI and 12S rRNA genes[J]. Experimental and Applied Acarology, 2016, 69(2): 117-128. DOI:10.1007/s10493-016-0028-3 |
| [15] |
Shen Y B, Li J L, Feng B B. Genetic analysis of cultured and wild populations of Mytilus coruscus based on mitochondrial DNA[J]. Zoological Research, 2009, 30(3): 240-246. DOI:10.3724/SP.J.1141.2009.03240 |
| [16] |
Feng Y W. DNA barcoding and molecular phylogeny of the Pteriina and Arcoida[D]. Qingdao: Ocean University of China, 2012. [冯艳微.珍珠贝亚目和蚶目DNA条形码与系统发生学研究[D].青岛: 中国海洋大学, 2012.] http://cdmd.cnki.com.cn/Article/CDMD-10423-1012505786.htm
|
| [17] |
Chiesa S, Lucentini L, Freitas R, et al. A history of invasion:COI phylogeny of Manila clam Ruditapes philippinarum in Europe[J]. Fisheries Research, 2017, 186(1): 25-35. |
| [18] |
Mona K, Oshaghi M A, Sedaghat M M, et al. Sequence analysis of mtDNA COI barcode region revealed three haplotypes within Culex pipiens assemblage[J]. Experimental Parasitology, 2017, 181: 102-110. DOI:10.1016/j.exppara.2017.08.003 |
| [19] |
Alcock A L, Barratt I, Eleaume M, et al. Cryptic specitation and the circumpolarity debate:a case study on endemic Southern Ocean Octopuses using the COI barcode of life[J]. Deep Sea Research Part Ⅱ:Topical Studies in Ocean ography, 2010, 58(1): 242-249. |
| [20] |
Zou S M. DNA barcoding and phylogeny of Neogastropoda[D]. Qingdao: Ocean University of China, 2013. [邹山梅.新腹足目贝类DNA条形码系统构建及系统发育研究[D].青岛: 中国海洋大学, 2013.] http://cdmd.cnki.com.cn/Article/CDMD-10423-1013354649.htm
|
| [21] |
Davison A, Rose I, Blackie E, et al. DNA barcoding of stylommatophoran land snails:a test of existing sequences[J]. Molecular Ecology Resources, 2009, 9(4): 1092-1101. DOI:10.1111/men.2009.9.issue-4 |
| [22] |
Liang H F, Xu X P, Huang Z J, et al. The molecular phylogeny on eight species of Panulirus Genusfrom the Chinese coast based on COI gene sequence[J]. Acta Scientiarum Naturalium Universitatis Sunyatseni, 2011, 50(6): 94-98. [梁华芳, 徐晓鹏, 黄志坚, 等. 中国沿海龙虾属8种龙虾COI基因序列的分子系统学研究[J]. 中山大学学报(自然科学版), 2011, 50(6): 94-98.] |
| [23] |
Chen A H, Li C X, Feng G N. Phylogenetic relationships of the genus Meretrix (Mollusca:Veneridae) based on mitochondrial COI gene sequences[J]. Zoological Research, 2009, 30(3): 233-239. [陈爱辉, 李朝霞, 封功能. 线粒体COI、COⅡ和CYTB基因在鲍属物种鉴定中的适用性分析[J]. 动物学研究, 2009, 30(3): 233-239.] |
| [24] |
Meng X P, Gao R C, Shen X, et al. DNA barcodes of clam Coelomactata antiquata (Bivalvia:Veneroida) in China based on COI gene[J]. Fisheries Science, 2011, 30(10): 627-630. [孟学平, 高如承, 申欣, 等. 基于COI的中国西施舌DNA条形码[J]. 水产科学, 2011, 30(10): 627-630.] |
| [25] |
Zhang D, Lei G C, Gong C, et al. Genetic diversity of Neosalanx taihuensis based on mitochondrial COI sequences[J]. Journal of Lake Sciences, 2012, 24(2): 299-306. [张迪, 雷光春, 龚成, 等. 基于COI基因序列的太湖新银鱼遗传多样性[J]. 湖泊科学, 2012, 24(2): 299-306. DOI:10.3969/j.issn.1003-5427.2012.02.020] |
 2018, Vol. 25
2018, Vol. 25

