2. 海南大学海洋学院, 海南 海口 570228;
3. 广州迪澳科技有限公司, 广东 广州 510663
2. The Ocean College, Hainan University, Haikou 570228, China;
3. Guangzhou Deaou Bio-Tech Co., Guangzhou 510663, China
哈维氏弧菌(Vibrio harveyi)为水产养殖中常见的条件性致病菌, 可以感染鲑(Centropomus undecimalis) [1]、居氏鲷(Acanthopagrus cuvieri)[2]、高体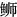
2016年通过对海南后水湾深水网箱养殖卵形鲳鲹(Trachinotus ovatus)“烂身病”研究发现, 哈维氏弧菌也是引发该病的主要病原, 每年因该病暴发对海南省养殖业造成上亿元的损失[11]。然而在养殖生产过程中如何快速诊断该病原菌还存在一定的困难, 虽然有时可通过外观症状进行初步判断, 但在海水养殖鱼类疾病中, 包括溶藻弧菌(V. alginolyticus)和创伤弧菌(V. vulnificus)等病原均可引起养殖动物体表溃烂等“烂身”症状, 因此仅凭外观症状无法准确确定病原种类。另外, 为了有效控制疾病的传播与危害, 往往需要在疾病发生早期典型症状尚未出现时就要快速准确诊断出病原, 以便尽早采取针对性的防控措施。因此, 迫切需要建立简单、快速、准确的病原检测技术。
目前, 对养殖鱼类细菌病原的检测方法很多, 但是各有优缺点。其中, 传统的病原分离和生理生化鉴定方法虽然操作简单, 但是耗时较长, 而且检测结果准确度不高, 对一些近似菌种也难以区分; 免疫诊断技术需要制作抗体, 成本高, 灵敏度低; 而16S rRNA基因的序列分析只能鉴定到科属水平[12-13]。就哈维氏弧菌检测而言, 虽然多位点序列分析以及基因组测序法能有效区分哈维氏弧菌与其他近缘弧菌, 但操作步骤烦琐, 且测序分析费昂贵, 对仪器设备要求也较高, 只能适用于科研和专门检测机构, 在生产一线难以推广应用[14-15]。近年来, 针对哈维氏弧菌特异性基因的鉴定和筛选成为研究热点, 并建立了多种快速检测方法, 包括多重PCR法、LAMP法等[16-19], 不过, 前者需要PCR仪和凝胶电泳仪等较昂贵的检测仪器; 后者采用浊度计检测或显色法对扩增结果进行检测, 存在灵敏度低和假阳性率高等问题。因此, 进一步研究开发简单、快速、准确的哈维氏弧菌检测技术已十分必要。
本研究将根据LAMP技术原理, 以哈维氏弧菌菌株QT520的基因序列设计特异性引物和加入特异荧光染料, 在已有相关研究方法基础上进行改良[16, 18], 旨在为生产一线提供一种简单、快速、准确的哈维氏弧菌检测方法—哈维氏弧菌实时荧光环介导等温扩增(Real-time loop-mediated isothermal amplification, RT-LAMP)技术。
1 材料与方法 1.1 实验菌株大菱鲆弧菌(V. scophtalmi)、副溶血性弧菌(V. parahaemolyticus ATCC 33847)、哈维氏弧菌(V. harveyi CCTCC AB2010414)、溶藻弧菌(V. alginolyticus)、霍乱弧菌(V. cholerae CCTCC AB2010414)、拟态弧菌(V. mimicus ATCC 33653)、创伤弧菌(V. vulnificus ATCC 27562)和坎氏弧菌(V. campbellii ATCC 33863)均来自中国科学院南海海洋研究所; 沙门氏菌(Salmonella sp.)、金黄色葡萄球菌(Staphylococcus aureus)、铜绿假单胞菌(Pseudomonas aeruginosa)、单增李斯特菌(Listeria monocytogenes)、副溶血性弧菌(V. Parahemolyticus)和创伤弧菌(V. vulnificus)购自广州迪澳生物科技有限公司; 哈维氏弧菌LG353和哈维氏弧菌QT520由海南大学和海南省海洋与渔业科学院分离自患病卵形鲳鲹并–80℃保存(表 1)。所有以上菌株都进行了16S rRNA鉴定[20]。
|
|
表 1 实验菌株信息 Tab.1 Experimental strain information |
Bst DNA聚合酶大片段(New England Biolabs公司); 甜菜碱(Betaine)、(NH4)2SO4、Tris-HCl、KCl、MgSO4和Tween-20 (Sigma公司); Taq DNA聚合酶和dNTPs(宝生物工程(大连)有限公司); SYTO-9荧光染料(Life Technologies公司); 正向外引物F3、正向内引物FIP、反向内引物BIP和反向外引物B3(生工生物工程(上海)有限公司); 细菌基因组DNA快速提取试剂盒(广州迪澳生物科技有限公司); 普通PCR试剂盒(Takara); SuperReal荧光定量预混试剂盒(天根)。
1.3 实验仪器与设备–80℃冻存柜, DAYA-024 (Thermo Fisher Scientific, 美国); 高速台式离心机, PICO17(Thermo Fisher Scientific, 美国); 漩涡混合器, MS2 (IKA, 德国); 微量移液(Eppendorf, 德国); 分析天平, AL104(梅特勒-托利多仪器有限公司, 瑞士); 超净工作台, SW-CJ-1FD(苏州安泰空气技术有限公司, 江苏); 实时荧光PCR仪(ABI, 美国); 微量紫外分光光度计(Thermo Fisher Scientific, 美国)。
1.4 实验方法 1.4.1 细菌培养和基因组DNA的制备选用的弧菌划线接种于TCBS固体培养基, 30℃培养12~16 h后, 挑取单菌落于2216E培养基中震荡培养至所需浓度; 沙门氏菌、铜绿假单胞菌、单增李斯特、金黄色葡萄球菌划线接种于BHI固体培养基, 30℃或者37℃培养后, 挑取单菌落于BHI液体培养基中震荡培养至所需浓度。细菌基因组的提取选用细菌基因组DNA快速提取试剂盒, 操作步骤参考说明书。
1.4.2 引物的设计以哈维氏弧菌QT520 ToxR (跨膜转录激活蛋白)基因一段特异DNA序列(GenBank: CP018680.2 Range 1: 3543839 to 3544714)作为哈维氏弧菌的特异靶标序列[21], 使用软件LAMP DESIGNER (http://www.optigene.co.uk/wp-content/uploads/2012/11/LAMP-Designer-flyer.pdf)设计TOX-1、TOX-2、TOX-3和TOX-4共4套特异性引物(表 2)。每套引物主要包括两个外引物和两个内引物, 特异结合靶序列上的6个特异区域。内引物FIP包含F1c(与Fl区域互补)和F2序列, 内引物BIP包含B1c (与B1区域互补)和B2序列, 外引物为F3和B3序列(图 1)[22-23]。
|
|
表 2 引物序列 Tab.2 Primer sequence |
|
图 1 引物组成及对应区域 Fig.1 Primer composition and corresponding regions |
根据文献[16, 18]略做修改, 确定25 μL的反应体系, 其成分包括:内引物FIP和BIP各1.6 μmol/L, 外引物F3和B3各0.2 μmol/L, 20 mmol/L Tris-HCl (pH 8.8), 10 mmol/L KCl, 8 mmol/L MgSO4, 10 mmol/L (NH4)2SO4, 0.1% Tween-20, 1 mol/L甜菜碱, 6 mmol/L MgSO4, 1.6 mmol/L dNTP, 8 U Bst大片段DNA聚合酶, 0.2 μmol/L SYTO-9。
1.4.4 筛选引物实验以哈维氏弧菌标准菌株E385的DNA为模板, 用设计的4套引物在API7500仪器上进行实时荧光扩增反应, 反应条件为63℃, 60 min和70 min, 以便筛选出无引物自身扩增、出峰时间早、扩增曲线标准的最佳引物。
1.4.5 特异性实验以各菌株的DNA作为特异性反应模板, 用引物TOX-4进行RT-LAMP反应, 63℃运行60 min, 验证反应的特异性, 利用实时荧光仪ABI 7500观察反应结果。
1.4.6 灵敏度实验将提取的DNA用Tris-HCL (pH 8.0)进行10倍梯度的稀释, 分别为10 ng/μL、1 ng/L、100 pg/μL、10 pg/μL、1 pg/μL、100 fg/μL、10 fg/μL和1 fg/μL, 作为DNA灵敏度检测实验的模板。同时, 将过夜培养的哈维氏弧菌QT520菌液(浓度为0.8×109 cfu/mL)用无菌生理盐水进行10倍梯度稀释, 分别为10 cfu/mL、102 cfu/mL、103 cfu/mL、104 cfu/mL和105 cfu/mL, 作为菌液灵敏度检测实验的模板。
哈维氏弧菌DNA灵敏度
以各梯度的DNA作为模板, TOX-4为引物, 按照优化的反应体系进行RT-LAMP反应, 每个梯度2次重复。反应程序: 63℃ 20 s; 63℃ 30 s, 63℃ 30 s, 60个循环。
以各梯度的DNA作为模板, 选用F3-4/B3-4为引物, 进行普通PCR反应, 每个梯度2次重复。反应体系为10×PCR缓冲液2.5 μL; Taq聚合酶(5 U/μL) 0.25 μL; dNTP(各2.5 mmol/L) 2μL; 引物F3-4(10 μmol/L)和引物B3-4(10 μmol/L)各2.5 μL; 超纯水13.25 μL; DNA模板2 μL。反应程序: 94℃预变性5min; 94℃变性30 s, 55℃退火30 s, 72℃延伸1 min, 30个循环; 最后, 72℃延伸1 min。反应产物利用1.5%琼脂糖凝胶电泳检测(100 V, 40 min)。
同时, 以各梯度的DNA作为模板, 以引物ToxR F1: CCACTGCTGAGACAAAAGCA, ToxR R1: GTGATTCTGCAGGGTTGGTT, ToxR P1: CAGCCGTCGAACAAGCACCG[24], 进行Real-time PCR反应, 每个梯度2次重复。反应体系为2× SuperRealPreMix(Probe) 12.5 μL; 引物ToxR F1 (10 μmol/L) 0.8 μL; 引物ToxR R1 (10 μmol/L) 0.8 μL; ToxR P1 0.4 μL; 超纯水8.5 μL以及DNA模板2 μL。反应程序: 95℃预变性10 min, 95℃变性15 s, 60℃退火1 min, 40个循环。
哈维氏弧菌菌液灵敏度
以各梯度的菌液的DNA为模板, TOX-4为引物, 进行RT-LAMP反应, 每个梯度2次重复。
同时, 以各梯度菌液的DNA作为模板, 选用F3-4/B3-4为引物, 进行普通PCR反应, 每个梯度2次重复。
以各梯度菌液的DNA为模板, 以ToxR F1、ToxR R1和ToxR P1为引物, 进行RT-PCR反应, 每个梯度2次重复。
1.5 实际样品检测将22个取自卵形鲳鲹鱼类组织及24个分离出的菌株(具体信息见表 3)进行哈维氏弧菌RT-LAMP检测。使用DNA提取试剂盒提取实际样品的基因组DNA, 以提取的DNA为模板, 用引物TOX-4进行RT-LAMP反应。
|
|
表 3 RT-LAMP检测与16S rDNA测序对不同样品中哈维氏弧菌的检测结果比较 Tab.3 Comparison of the test results of Vibrio harveyi in different samples using the RT-LAMP and 16S rDNA sequencing methods |
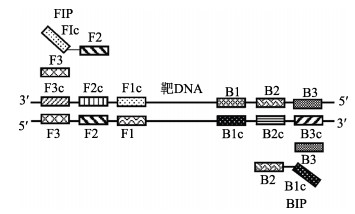
4套引物均能扩增出相应产物, 但是产物出现的时间和曲线规则程度有差异。由图 2可知, 在荧光仪ABI7500上63℃反应60 min后, 4套引物都没有出现引物自结合的非特异性扩增, 且引物组TOX-4出峰时间最早、扩增效率最高; 由图 3可知, 在Deaou-308C上63℃运行反应70 min后, 4套引物也都没有出现引物自结合的非特异扩增, 且引物组TOX-4不仅出峰时间最早、扩增效率最高, 且扩增曲线最规则。因此, 引物组TOX-4为设计的四套引物中的最佳引物, 后续分析均选用该引物组。
|
图 2 以哈维氏弧菌E385 DNA为模板利用不同引物RT-LAMP法扩增结果(ABI 7500) Fig.2 The results of RT-LAMP using different primers and DNA from Vibrio harveyi E385 as the template in ABI 7500 |
|
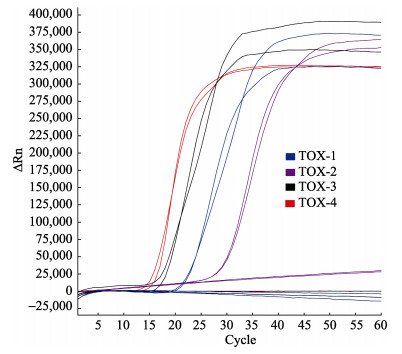
图 3 以16株弧菌DNA为模板检测哈维氏弧菌RT-LAMP法的特异性检测 Fig.3 The specificity of RT-LAMP was tested using DNA templates extracted from 16 strains of Vibrio sp. |
哈维氏弧菌菌株E385、LG353和QT520菌均出现比较规则的扩增曲线, 而坎氏弧菌1.1596菌株以及其他弧菌或细菌均没有扩增反应。由此可见, 以引物组TOX-4为引物对哈维氏弧菌的扩增特异性良好, 且能够区分哈维氏弧菌与坎氏弧菌等其他弧菌。
2.3 灵敏度实验 2.3.1 哈维氏弧菌DNA灵敏度检测以10 ng/μL、1 ng/μL、100 pg/μL、10 pg/μL、1 pg/μL和100 fg/μL的DNA为模板的扩增反应均有出现标准S形曲线, 而以10 fg/μL和1 fg/μL的DNA为模板的扩增反应均没有扩增反应(图 4A); 普通PCR反应检出限为100 pg/μL (图 4B); RT-PCR方法对哈维氏弧菌的DNA检测的灵敏度为100 fg/μL, 当浓度为10 fg/μL时检测结果为弱阳性(图 4C)。由此可见, 以Tox-4为引物的RT-LAMP法对哈维氏弧菌DNA的检测灵敏度与RT-PCR法相当, 要比普通PCR高1000倍。
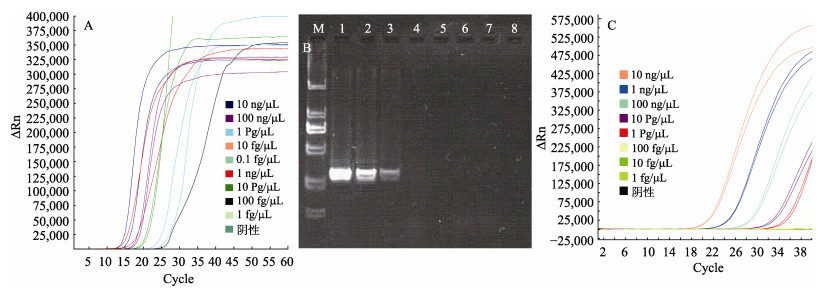
|
图 4 以10倍梯度稀释的哈维氏弧菌QT520 DNA为模板RT-LAMP(A)、普通PCR法(B)以及RT-PCR法(C)的检测灵敏度 M: DL2000 Marker; 1: 10 ng/μL; 2: 1 ng/μL; 3: 100 pg/μL; 4: 10 pg/μL; 5: 1 pg/μL; 6: 100 fg/μL; 7:10 fg/μL; 8: 1 fg/μL Fig.4 Sensitivity test for RT-LAMP(A), general PCR(B) and RT-PCR(C) using the 10-fold series dilutions of DNA templates from Vibrio harveyi QT520 M: DL2000 Marker; 1: 10 ng/μL; 2: 1 ng/μL; 3: 100 pg/μL; 4: 10 pg/μL; 5: 1 pg/μL; 6: 100 fg/μL; 7:10 fg/μL; 8: 1 fg/μL |
RT-LAMP法对菌液DNA检测的灵敏度可达103 cfu/mL, 当浓度为102 cfu/mL时, 2个重复有一个检出(图 5A); 普通PCR反应检测的灵敏度为104 cfu/mL(图 5B); RT-PCR反应检测的灵敏度为103 cfu/mL(图 5C)。由此可见, 对于哈维氏弧菌菌液, RT-LAMP法的检出限为103 cfu/mL, 其灵敏度与RT-PCR法相当, 要比普通PCR高10倍。
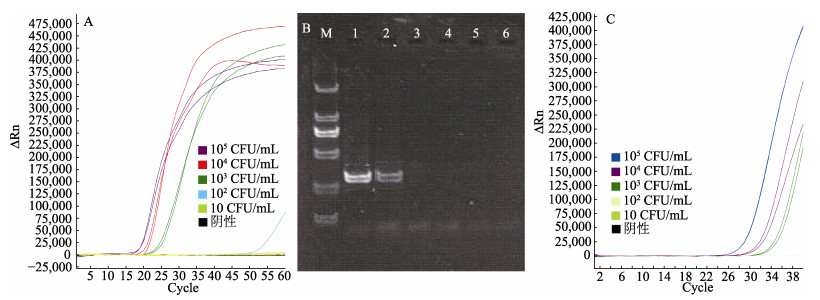
|
图 5 以10倍梯度稀释的哈维氏弧菌菌液DNA为模板RT-LAMP(A)、普通PCR法(B)以及RT-PCR法(C)灵敏度检测 M: DL2000 Marker, 1: 105 cfu/mL, 2: 104 cfu/mL, 3: 103 cfu/mL, 4: 102 cfu/mL, 5: 10 cfu/mL, 6:阴性对照. Fig.5 Sensitivity test for RT-LAMP(A), general PCR(B) and RT-PCR(C) using the 10-fold series dilutions of DNA templates from Vibrio harveyi QT520. M: DL2000 Marker, 1: 105 cfu/Ml, 2: 104 cfu/mL, 3: 103 CFU/mL, 4: 102 cfu/mL, 5: 101 cfu/mL, 6: negative control |
对2016年8—10月采集到的22份卵形鲳鲹组织样品及24份菌株样品进行哈维氏弧菌RT-LAMP检测, 结果如表 3所示, 共检测到25份阳性样品, 其中阳性组织样品17份, 阳性菌液8份, 与16S rDNA测序鉴定结果一致[11] (华大基因公司, 深圳)。
3 讨论哈维氏弧菌是引起深水网箱养殖卵形鲳鲹“烂身病”的主要病原菌, 为了能够在发病早期及时检测出该病原菌, 以减少疾病暴发造成的损失, 急需建立一种快速的、准确的检测方法。
LAMP技术是一种新的恒温扩增方法, 自2000年首次报道以来, 已经在核酸研究、疾病诊断、性别鉴定、转基因及病原检测等领域广泛应用[25]。近年在水产养殖领域应用发展较快, 2004年LAMP技术首次被应用于牙鲆病原菌爱德华氏菌的检测[26], 此后鱼诺卡氏菌(Nocard iaseriolae)、柱状黄杆菌(Flavobacterium columnare)、嗜水气单胞菌(Aeromonas hydrophila)、溶藻弧菌等病原菌的LAMP法检测相继报道[27-30]。
Imai等[31]认为环介导等温扩增技术具有比PCR更加高的灵敏度; Cao等[16]提出检测哈维氏弧菌的LAMP法, 根据ToxR基因设计了4条引物, 以SYBR GreenⅠ为荧光染料, 通过显色法对样品哈维氏弧菌进行检测, 灵敏度为17.2 cfu/ reaction, 比普通PCR高10倍, 其缺点在于荧光染料SYBR GreenⅠ会一定程度抑制LAMP反应的进行, 不能提前直接加入反应体系, 需要等反映后开盖加入荧光染料, 容易造成扩增产物污染, 出现假阳性。LAMP法和试纸条法结合起来, 以溶血素基因序列设计引物, 可以将该方法的灵敏度提高到2 cfu/reaction和0.6 cfu/reaction[32-33]。
本研究建立的RT-LAMP法, 主要以分离到的病原菌QT520的ToxR基因序列设计引物, 灵敏度为103 cfu/mL或者20 cfu/reaction, 与Cao等[16]提出哈维氏弧菌的LAMP法相当。不过, 本研究建立的RT-LAMP法以SYTO-9为荧光染料, 可以不开盖实时监测到扩增结果, 消除了LAMP产物污染源, 克服了上述提到的假阳性问题。以SYTO-9为荧光染料的RT-LAMP法应用于对创伤弧菌的检测, 也取得良好的效果[34]。
在检测耗时方面, Cao等[16]、Thongkao等[32]和程蝶等[33]利用LAMP进行检测的时间分别为50 min, 60 min和90 min, 而本研究建立的RT-LAMP法, 可实时监测到检测结果, 仅需40 min, 就可以知道全部检测结果。
监测仪器选择方面, 因为LAMP法是基于恒温扩增, 不仅ABI 7500荧光定量PCR仪器适用, 其他的恒温荧光检测仪, 如Deaou-308C、ESE Quant Tube Scanner仪器同样适用于本检测方法, 不仅价格低廉, 而且携带方便, 非常适合在基层防疫部门和养殖生产基地推广应用。
总之, 本方法具有高特异性, 高灵敏度, 耗时短且操作简单, 能实时观测结果等优点, 非常适合在生产现场进行哈维氏弧菌的检测, 为其引起的相关疾病的监控与预防奠定了良好基础。
4 小结选择菌株QT520的一段特异性ToxR序列(GenBank: CP018680.2 Range 1: 3543839 to 3544714)进行RT-LAMP引物设计; 经引物筛选后, 确定引物组TOX-4为本研究最适用的引物。
RT-LAMP法检测哈维氏弧菌的基因组DNA及菌液灵敏度分别为100 fg/μL和103 cfu/mL或者20 cfu/reaction, 其灵敏度与RT-PCR法相当, 比普通PCR的检测灵敏度要分别高1000倍和10倍。此外, RT-LAMP法检测哈维氏弧菌消除了LAMP产物污染源, 且检测时间只需要40 min, 少于LAMP法的50~90 min, 具有耗时短、特异性和灵敏度高、仪器简单、安全高效等优点, 非常适合在生产现场进行哈维氏弧菌的检测。
| [1] |
Kraxbergerbeatty T, Mcgarey D J, Grier H J, et al. Vibrio harveyi, an opportunistic pathogen of common snook, Centropomus undecimalis (Bloch), held in captivity[J]. Journal of Fish Diseases, 1990, 13(6): 557-560. DOI:10.1111/jfd.1990.13.issue-6 |
| [2] |
Saeed M. Association of Vibrio harveyi with mortalities in cultured marine fish in Kuwait[J]. Aquaculture, 1995, 136: 21-29. DOI:10.1016/0044-8486(95)01045-9 |
| [3] |
Wu H B, Pan J P. Pathological studies on the vibriosis of marine cage cultured Seriola dumerili[J]. Journal of Fisheries of China, 1999, 23(2): 206-209. [吴后波, 潘金培. 海水网箱养殖高体鰤弧菌病病理学研究[J]. 水产学报, 1999, 23(2): 206-209.] |
| [4] |
Chabrillón M, Rico R M, Arijo S, et al. Interactions of microorganisms isolated from gilthead sea bream, Sparus aurata L., on Vibrio harveyi, a pathogen of farmed Senegalese sole, Solea senegalensis (Kaup)[J]. Journal of Fish Diseases, 2005, 28(9): 531-537. DOI:10.1111/jfd.2005.28.issue-9 |
| [5] |
Pujalte M J, Sitjà-Bobadilla A, Macián M C, et al. Virulence and molecular typing of Vibrio harveyi strains isolated from cultured dentex, gilthead sea bream and european sea bass[J]. Systematic and Applied Microbiology, 2003, 26(2): 284-292. DOI:10.1078/072320203322346146 |
| [6] |
Mei B, Zhou Y C, Xu X D, et al. Isolation and identification of bacteria pathogens from Epinephelus coioides with tail-rotted disease[J]. Journal of Tropical Oceanography, 2010, 29(6): 118-124. [梅冰, 周永灿, 徐先栋, 等. 斜带石斑鱼烂尾病病原菌的分离与鉴定[J]. 热带海洋学报, 2010, 29(6): 118-124. DOI:10.3969/j.issn.1009-5470.2010.06.018] |
| [7] |
Chen X G, Wu S Q, Shi C B, et al. Isolation and identification of pathogenetic Vibrio harveyi from estuary cod Epinephelus coioides[J]. Journal of Fishery Sciences of China, 2004, 11(4): 313-317. [陈献稿, 吴淑勤, 石存斌, 等. 斜带石斑鱼病原菌(哈维氏弧菌)的分离与鉴定[J]. 中国水产科学, 2004, 11(4): 313-317. DOI:10.3321/j.issn:1005-8737.2004.04.007] |
| [8] |
Cui J, Fan X T, Liu W Z, et al. Isolation and identification of vibriosis pathongens of marine cultured fishes in southern China[J]. Natural Science Journal of Hainan University, 2014, 32(3): 244-251. [崔婧, 范雪亭, 刘文竹, 等. 华南地区海水养殖鱼类主要弧菌病原的分离与鉴定[J]. 海南大学学报:自然科学版, 2014, 32(3): 244-251.] |
| [9] |
Cui J. The typing of fish pathogen Vibrio harveyi in South China and the research of its prevention strategies[D]. Haikou: Hainan University, 2014. [崔婧.华南地区鱼类病原哈维氏弧菌分型及其防治策略研究[D].海口: 海南大学, 2014.] http://cdmd.cnki.com.cn/Article/CDMD-10589-1014240076.htm
|
| [10] |
Shi Y S, Tong G Z, Xue C B, et al. Identification and drug sensitive test of Vibrio harveyi isolated from the tail-rotted Pseudosciaena crocea in Zhoushan[J]. Chinese of Journal of Health Laboratory Technology, 2005, 15(3): 267-269. [石亚素, 童国忠, 薛超波, 等. 舟山养殖大黄鱼烂尾病中哈氏弧菌的分离鉴定及药敏试验[J]. 中国卫生检验杂志, 2005, 15(3): 267-269. DOI:10.3969/j.issn.1004-8685.2005.03.005] |
| [11] |
Tu Z G. Studies on microbial diversity and rot body disease of Trachinotus ovatus in deep-sea cage aquaculture area in Houshui Bay, Hainan[D]. Haikou: Hainan University, 2017. [涂志刚.海南后水湾深水网箱养殖区微生物多样性及其卵形鲳鲹"烂身病"研究[D].海口: 海南大学, 2017.]
|
| [12] |
Gomezgil B, Sotorodriguez S, Garciagasca A, et al. Molecular identification of Vibrio harveyi-related isolates associated with diseased aquatic organisms[J]. Microbiology, 2004, 150(6): 1769-1777. DOI:10.1099/mic.0.26797-0 |
| [13] |
Canogomez A, Bourne D G, Hall M R, et al. Molecular identification, typing and tracking of Vibrio harveyi in aquaculture systems:current methods and future prospects[J]. Aquaculture, 2009, 287(1): 1-10. |
| [14] |
Gurtler V, Mayall B C. Genomic approaches to typing, taxonomy and evolution of bacterial isolates[J]. International Journal of Systematic and Evolutionary Microbiology, 2001, 51(1): 3-16. DOI:10.1099/00207713-51-1-3 |
| [15] |
Thompson C C, Vicente A C P, Souza R C, et al. Genomic taxonomy of vibrios[J]. BMC Evolutionary Biology, 2009, 9(1): 258. DOI:10.1186/1471-2148-9-258 |
| [16] |
Cao Y T, Wu Z, Jian J, et al. Evaluation of a loop-mediated isothermal amplification method for the rapid detection of Vibrio harveyi in cultured marine shellfish[J]. Letters in Applied Microbiology, 2010, 51(1): 24-29. |
| [17] |
Haldar S, Neogi S B, Kogure K, et al. Development of a haemolysin gene-based multiplex PCR for simultaneous detection of Vibrio campbellii, Vibrio harveyi and Vibrio parahaemolyticus[J]. Letters in Applied Microbiology, 2010, 50(2): 146-152. DOI:10.1111/lam.2009.50.issue-2 |
| [18] |
Zhang J, Shi H, Xie J J, et al. Rapid detection of pathogenic Vibrio harveyi by loop-mediated isothermal amplification (LAMP) method[J]. Journal of Shanghai Ocean University, 2010, 19(5): 601-607. [张静, 施慧, 谢建军, 等. 利用LAMP法快速检测致病性哈维氏弧菌[J]. 上海海洋大学学报, 2010, 19(5): 601-607.] |
| [19] |
Canogomez A, Hoj L, Owens L, et al. A multiplex PCR-based protocol for identification and quantification of Vibrio harveyi-related species[J]. Aquaculture, 2015, 437: 195-200. DOI:10.1016/j.aquaculture.2014.10.050 |
| [20] |
Gauger E J, Gomezchiarri M. 16S ribosomal DNA sequencing confirms the synonymy of Vibrio harveyi and V. carchariae[J]. Diseases of Aquatic Organisms, 2002, 52(1): 39-46. |
| [21] |
Tu Z, Li H, Zhang X, et al. Complete genome sequence and comparative genomics of the golden pompano (Trachinotus ovatus) pathogen, Vibrio harveyi strain QT520[J]. PeerJ, 2017, 5(12): e4127. |
| [22] |
Kuang Y Y, Ye W X, Lv J Z, et al. Loop mediated isothermal amplification (Lamp) method for detection of Vibrio cholerae[J]. Chinese Journal of Public Health, 2009, 25(11): 1310-1312. [匡燕云, 叶卫翔, 吕敬章, 等. 霍乱弧菌环介导等温扩增LAMP技术检测[J]. 中国公共卫生, 2009, 25(11): 1310-1312. DOI:10.11847/zgggws2009-25-11-18] |
| [23] |
He L. Study on a loop mediated isothermal amplification (LAMP) for the rapid detection of pathogens in aquatic animals[D]. Hangzhou: Zhejiang University, 2012. [何琳.环介导等温扩增技术快速检测水产动物病原的研究[D].杭州: 浙江大学, 2012.] http://cdmd.cnki.com.cn/Article/CDMD-10335-1013186907.htm
|
| [24] |
Schikorski D, Renault T, Paillard C, et al. Development of TaqMan real-time PCR assays for monitoring Vibrio harveyi infection and a plasmid harbored by virulent strains in European abalone Haliotis tuberculata aquaculture[J]. Aquaculture, 2013, 392-395(10): 106-112. |
| [25] |
Notomi T, Okayama H, Masubuchi H, et al. Loop-mediated isothermal amplification of DNA[J]. Nucleic Acids Research, 2000, 28(12): e63. DOI:10.1093/nar/28.12.e63 |
| [26] |
Savan R, Igarashi A, Matsuoka S, et al. Sensitive and rapid detection of edwardsiellosis in fish by a loop-mediated isothermal amplification method[J]. Applied and Environmental Microbiology, 2004, 70(1): 621-624. DOI:10.1128/AEM.70.1.621-624.2004 |
| [27] |
Wang G L, Liu L, Li S Y. Development of a SYBR GreenⅠreal-time PCR assay for detection of Nocardia seriolae and its application[J]. Journal of Fisheries of China, 2012, 36(4): 509-513. [王国良, 刘璐, 李思源. 蛳鱼诺卡氏菌SYBRGreenⅠ实时荧光定量PCR检测方法的建立与应用[J]. 水产学报, 2012, 36(4): 509-513.] |
| [28] |
Wang G L, Liu L, Xu Y J. Development and application of loop-mediated isothermal amplication for detection of fish pathogenic Nocardia seriolae[J]. Oceanologia et Limnologia Sinica, 2011, 42(1): 27-31. [王国良, 刘璐, 徐益军. 鱼类致病鰤鱼诺卡氏菌(Nocardia seriolae)的LAMP检测技术建立与应用[J]. 海洋与湖沼, 2011, 42(1): 27-31.] |
| [29] |
Cheng T Y, Liu X, Chang X B. Establishment and application of loop-mediated isothermal amplification for detection of Aeromonas hydrophila[J]. Veterinary Science in China, 2007, 37(12): 1013-1016. [程天印, 刘洵, 常小斌. 嗜水气单胞菌Lamp检测方法的建立及应用[J]. 中国兽医科学, 2007, 37(12): 1013-1016.] |
| [30] |
Ding W C, Hu J R, Shi Y H, et al. Establishment of loop-mediated isothermal amplification for detection of Vibrio alginolyticus[J]. Journal of Molecular Cell Biology, 2009, 42(1): 70-76. [丁文超, 胡健饶, 史雨红, 等. 环介导恒温扩增技术快速检测溶藻弧菌[J]. 分子细胞生物学报, 2009, 42(1): 70-76.] |
| [31] |
Imai M, Ninomiya A, Minekawa H, et al. Development of H5-RT-LAMP (loop-mediated isothermal amplification) system for rapid diagnosis of H5 avian influenza virus infection[J]. Vaccine, 2006, 24(44): 6679-6682. |
| [32] |
Thongkao K, Longyant S, Silprasit K, et al. Rapid and sensitive detection of Vibrio harveyi by loop-mediated isothermal amplification combined with lateral flow dipstick targeted tovhh P2 gene[J]. Aquaculture Research, 2015, 46(5): 1122-1131. DOI:10.1111/are.2015.46.issue-5 |
| [33] |
Cheng D, Chai F C, Cai Y, et al. Visual detection of Vibrio harveyi based on loop-mediated isothermal amplification combined with a lateral flow dipstick[J]. Biotechnology Bulletin, 2016, 32(6): 60-68. [程蝶, 柴方超, 蔡怡, 等. 环介导等温扩增联合横向流动试纸条可视化检测哈维氏弧菌的研究[J]. 生物技术通报, 2016, 32(6): 60-68.] |
| [34] |
Han F, Ge B. Quantitative detection of Vibrio vulnificus in raw oysters by real-time loop-mediated isothermal amplification[J]. International Journal of Food Microbiology, 2010, 142(1): 60-66. |
 2018, Vol. 25
2018, Vol. 25

