2. 中国水产科学研究院黑龙江水产研究所, 淡水鱼类育种国家地方联合工程实验室, 黑龙江 哈尔滨 150070
2. National Local Joint Engineering Laboratory for Freshwater Fish Breeding, Heilongjiang River Fisheries Research Institute, Chinese Academy of Fishery Sciences, Harbin 150070, China
激素敏感性脂肪酶(hormone sensitive lipase, HSL或LIPE)是由脂肪组织泌的一种中性脂肪酶, 能够催化动物脂肪组织中的甘油三酯水解成游离脂肪酸, 是影响动物脂肪沉积的关键酶之一[1-2]。1988年, HSL基因全长cDNA首次由Holm等[3]从大鼠(Rattus norvegicus)的脂肪组织表达文库中获得。随后, 在人(Homo sapiens)[4]、小鼠(Mus musculus)[5]、猪(Sus scrofa)[6]等哺乳动物中也检测到该基因。近年来, 诸多学者开展了HSL基因多态性、mRNA表达与脂肪性状的相关性研究, 证实了HSL基因在动物脂质生成及代谢过程中具有重要调节作用。任善茂等[7]研究发现, HSL基因多态性与苏姜猪(Sus scrofa Sujiang)肌肉品质高度相关, AA基因型个体的平均背膘厚和肌内脂肪含量极显著高于BB基因型(P < 0.01), 并指出可通过提高AA基因型的频率来增加肌内脂肪含量, 从而改善猪肉的品质。Qiao等[8]研究发现哈萨克羊(Ovis aries Kazak) HSL基因表达水平与肌内脂肪含量显著相关。目前HSL基因被证明是畜禽肉质性状的重要候选基因[9]。
水产动物中, 牙鲆(Paralichthys olivaceus)[10]、虹鳟(Oncorhynchus mykiss)[11]、黄颡鱼(Pelteobagrus fulvidraco)[12]、真鲷(Pagrus major)[13]等多种鱼类中已经成功克隆到HSL基因, 并对其在鱼体各组织中的mRNA表达模式进行了初步研究。结果显示HSL基因在组织中表达广泛, 但不同组织中表达量存在较大差异, 通常在脂肪组织中表达量高于其他组织。此外, 饲料中营养素对于HSL基因表达的调控研究也有报道。Han等[14]研究显示, 高脂饲料能够促进杂交罗非鱼(Oreochromis niloticus× O. aureus)肝和肌肉中HSL基因表达, 但抑制了腹腔脂肪中HSL基因的表达; 艾庆辉等[15]研究显示, 高脂饲料组(21.88%)能显著促进半滑舌鳎(Cynoglossus semilaevis)稚鱼内脏团HSL基因的表达。但关于HSL基因与鱼类脂肪沉积以及肉质性状相关性的研究报道较少。
鱼体脂肪含量和脂肪酸的组成是评价鱼肉品质的重要标准之一[16]。研究表明, 肌内脂肪含量提高可以有效地改善肉质口感[17], 而腹腔(内脏周围)等非可食部位脂肪的大量沉积不仅影响鱼类肉质、口感和营养价值, 而且对鱼体健康、养殖效率和养殖成本均造成不利影响[18]。因此, 了解脂肪沉积机理及规律可为鱼类肉质改善、生产效率提高提供理论依据, 具有较大的实际意义。鲤(Cyprinus carpio)是中国重要的淡水经济鱼类之一, 目前中国已培育出25个鲤新品种, 是培育品种最多的水产养殖种, 极大地满足了中国鲤养殖产业的良种需求, 促进了国内淡水水产养殖业的迅速发展。随着人们对优质鱼类需求的不断增长, 加之养殖中片面追求高产量、高生长速度, 不同程度上造成了养殖鱼类体内脂肪堆积、品质下降、口感不佳等问题, 严重影响了鱼肉的品质。本研究从调控脂肪代谢关键酶基因入手, 克隆了lipea基因全长cDNA序列, 并与其他物种进行了同源性比对和系统发育进化树构建; 研究了其在鲤各组织中的表达分布、不同发育阶段的表达特征, 并对其表达量与脂肪沉积相关性进行了初步分析, 旨在为进一步研究鲤脂肪沉积分子调控机制奠定基础。
1 材料与方法 1.1 样品制备实验用鲤均由黑龙江水产研究所呼兰试验场培育, 选取同一池塘中商业饲料饲养条件下5尾体重200~250 g的1龄鱼, 禁食24 h, 取肝、肠、肾、脑、心脏、腹腔脂肪、脾、腹部肌肉(腹鳍基部上方约1 cm处肌肉)、背部肌肉(侧线上方、第2至第3背鳍之间肌肉)和血液样本, 迅速投入液氮冷冻备用。取鲤受精卵期、8细胞期、囊胚晚期、原肠胚晚期、7体节期、心跳时期、体循环期、出膜期、开口期和出苗5 d等10个不同发育时期的胚胎或仔鱼, 置于RNA later中用于制备RNA样本。脂肪含量极大10尾个体和极小10尾个体, 百分比分别为(9.56±0.72)%和(1.89±0.25)%, 选自于全基因组关联分析群体[19]。其来源为同一家系同一池塘养殖环境下的220尾鲤个体, 在采集背部肌肉用于脂肪含量测定的同时, 将背部肌肉(侧线上方、第2至第3背鳍之间肌肉)和腹腔脂肪组织保存于液氮备用。采样前, 用MS-222对实验用鱼进行麻醉。以上样品以手持匀浆器(QIAGEN)匀浆至无颗粒状态, 按照RNeasy Lipid Tissue Mini Kit说明书提取各组织样品总RNA。利用1.2%琼脂糖电泳与NanoDrop 8000对RNA样品进行质量和浓度控制。
1.2 基因全长cDNA序列获得使用PrimeScriptTM RT reagent Kit with gDNA Eraser(Perfect Real Time)试剂盒合成cDNA第一链(Takara RR047A)。根据NCBI数据库中斑马鱼lipea基因以及其他硬骨鱼类HSL基因的cDNA序列, 选择保守区域与鲤转录组数据库进行序列比对, 获得鲤lipea基因的部分cDNA序列并设计引物进行序列验证(表 1)。按照SMARTer®RACE5′/3′试剂盒(Clontech)说明书构建RACE反应模板, 根据已获得鲤lipea基因的保守区域的序列设计RACE扩增引物(表 1), SeqAmpTM DNA Polymerase (Clontech)试剂盒进行RACE扩增。
|
|
表 1 引物序列 Tab.1 Primers used in this study |
PCR产物使用1.2%琼脂糖凝胶电泳进行检测, 扩增产物的回收纯化参照琼脂糖胶回收试剂盒纯化(康为世纪), 纯化产物连接到载体, 转化至感受态细胞, 筛选阳性克隆, 送至苏州金唯智生物科技有限公司测序。将测序结果使用BLAST (https://www.ncbi.nlm.nih.gov/)比对, DNAMAN软件分析, Staden 1.7软件进行拼接, 最终获得lipea基因的全长cDNA序列。
1.3 基因序列特征分析及系统进化树构建用DNAMAN软件查找基因的开放阅读框(Open Reading Frame, ORF); 通过EXPASY (http://www.expasy.org/proteomics)等在线分析软件中的工具进行信号肽、蛋白疏水性、跨膜结构、蛋白的二级结构等分析。使用MEGA 7.0软件, 采用邻接法(Neighbor-Joining method, NJ)进行系统进化树的构建, 其中Bootsrap (1000次)得出各分支置信度。
1.4 基因表达规律分析利用已经获得的lipea基因全长cDNA序列, 设计荧光定量引物(表 1)。以鲤18S RNA基因为内参基因, 以上各组织样品cDNA为模板, 使用SYBR® Premix Ex Taq TM Ⅱ (Tli RNase H Plus)试剂盒(Takara), ABI 7500荧光定量PCR仪, 检测鲤lipea基因在不同组织、不同发育时期和脂肪含量极端差异个体背部肌肉和腹腔脂肪的相对表达量, 每个样品至少设计3个平行。使用2–∆∆Ct相对定量方法计算基因的相对表达量, 数据分析采用SPSS 17.0软件中单因素方差分析(one-way ANOVA)方法, 差异显著性以0.05为标准。
2 结果与分析 2.1 鲤lipea全长cDNA序列分析通过基因保守序列的同源扩增获得2082 bp的鲤lipea cDNA的核心序列, 并通过RACE扩增分别获得了1280 bp和1037 bp的5′和3′端序列, 进而通过拼接得到了3379 bp的基因cDNA全长序列(GenBank登录号: MF061228.2), 其中开放阅读框(open reading frame, ORF)长度为2079 bp, 编码693个氨基酸; 5′非翻译区(5′-UTR) 230 bp; 3′非翻译区(3′-UTR)为1067 bp, 包含25 bp的polyA尾巴, 两个“AATAAA”加尾信号分别位于PloyA上游969 bp和29 bp处。
利用ExPASY的ProtParam工具预测得知蛋白质相对分子量7.60 kD, 理论PI值为6.29, 亮氨酸Leu的含量最高, 为12.6%, 且不含氨基酸Pyl和Sec。带负电荷的残基总数(Asp+Glu)为65个, 而正电荷的残基总数(Arg+Lys)为58个; 理论计算得到的分子式为C3389H5346N916O1007S31, 总原子数为10689个。蛋白质的不稳定系数为45.72, 说明该蛋白不稳定。脂肪系数为92.60, 亲水性平均系数为-0.046。ExPASY的SOPMA工具得知鲤lipea蛋白质的二级结构以α螺旋为主, 为43.87%; Signal 4.1预测结果显示鲤lipea不具有信号肽结构, 为非分泌型蛋白(图 1上); SMART工具显示该蛋白含有3个功能域分别具有脂肪酶活性、乙酰基水解酶活性和水解酶活性(图 1下); Motif Scan预测脂酶功能域的活性位点(图 2); 使用TMHMM预测鲤lipea没有跨膜结构。
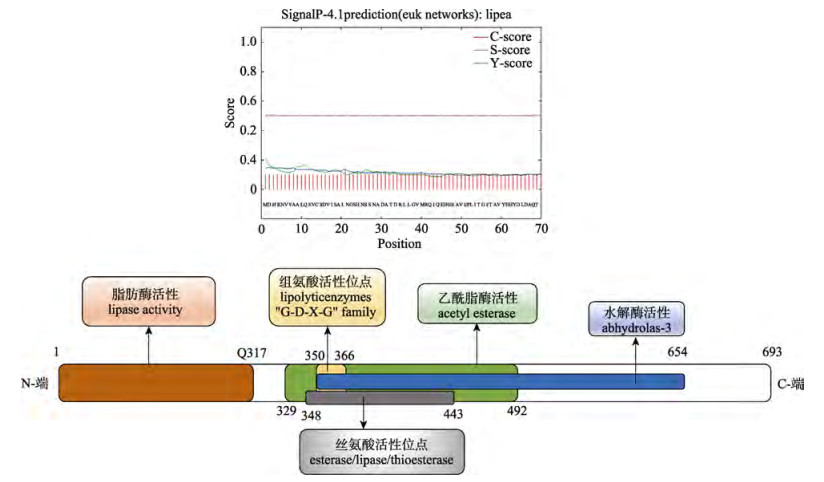
|
图 1 鲤lipea蛋白质信号肽预测(上)及结构示意图(下) Fig.1 The signal peptide location (up) and the structure of lipea protein (down) in common carp |

|
图 2 鲤lipea推测氨基酸序列的同源性多重比较
竖线表示lipea蛋白质N-端(Gln316)和C-端(Asp317); “●”为Ser-Asn-His组成的三联体催化活性中心的磷酸化位点; 灰色表示亲核Ser残基周围有GXSXG保守序列; “△”为酪蛋白激酶Ⅱ磷酸化位点; “#”为酰基化位点; “★”为蛋白激酶C磷酸化位; “ ”为酪氨酸激酶磷酸化位点。
Fig.2 Multiple alignment of deduced amino acid sequences of lipea in common carp Cyprinus carpio
Conserved boundary between N-terminal and C-terminal regions is indicated by a vertical line; the catalytic cores GXSXG were highlighted with a gray shadow; the catalytic triad is marked by black circles; casein kinase Ⅱ phosphorylation sites were shown as △; N-myristoylation sites were expressed by #; protein kinase C phosphorylation sites were presented ★; tyrosine kinase phosphorylation sites were shown as ”为酪氨酸激酶磷酸化位点。
Fig.2 Multiple alignment of deduced amino acid sequences of lipea in common carp Cyprinus carpio
Conserved boundary between N-terminal and C-terminal regions is indicated by a vertical line; the catalytic cores GXSXG were highlighted with a gray shadow; the catalytic triad is marked by black circles; casein kinase Ⅱ phosphorylation sites were shown as △; N-myristoylation sites were expressed by #; protein kinase C phosphorylation sites were presented ★; tyrosine kinase phosphorylation sites were shown as  . .
|
使用CLUSTAL 2.1软件进行鲤lipea基因编码蛋白的同源性氨基酸序列搜索, 结果显示其与斑马鱼、牙鲆和虹鳟的相似性分别为88.17%, 64.94%和63.35%, 不同物种间lipea氨基酸序列及同源性的差异主要集中在C-端的多肽上(图 2)。基于lipea基因的氨基酸序列, 利用MEGA7.0, 应用N-J法构建的13个物种的系统进化树结果表明, 鱼类聚为一大支, 其中同为鲤科鱼类的鲤、金线鲃和斑马鱼聚为一支, 鲆鲽类和鲑鳟类各聚为一支; 哺乳动物则聚为另一大支(图 3)。
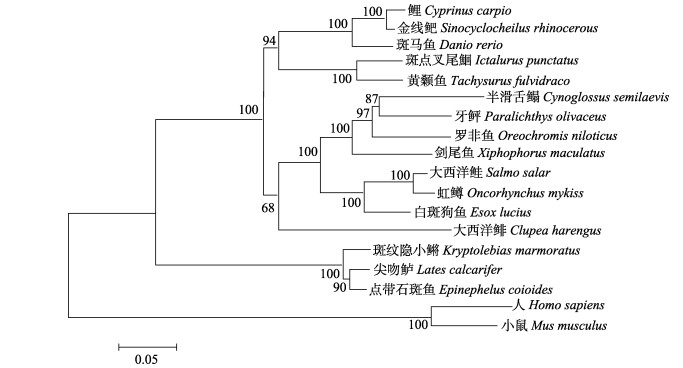
|
图 3 基于不同物种HSL氨基酸序列构建的NJ系统进化树 物种及其HSL的GenBank序列登录号分别为: Sinocyclocheilus rhinocerous, 金线鲃, XP_016300015; Danio rerio, 斑马鱼, NP_001303654; Ictalurus punctatus, 斑点叉尾鮰, XP_017336708.1; Tachysurus fulvidraco, 黄颡鱼, AIF72763.1; Cynoglossus semilaevis, 半滑舌鳎, XP_008329302.1; Paralichthys olivaceus, 牙鲆, XP_019959978.1; Oreochromis niloticus, 尼罗罗非鱼, XP_003447301.1; Xiphophorus maculatus, 剑尾鱼, XP_005811845.1; Salmo salar, 大西洋鲑, XP_013997642.1; Oncorhynchus mykiss, 虹鳟, NP_001184139.1; Esox lucius, 白斑狗鱼, XP_010899186.1; Clupea harengus, 大西洋鲱, XP_012684999.1; Kryptolebias marmoratus, 斑纹隐小鳉, XP_017271065.1; Lates calcarifer, 尖吻鲈, XP_018527967.1; Epinephelus coioides, 点带石斑鱼, AGU42438.1; Homo sapiens, 人, XP_005258994.1; Mus musculus, 小鼠, XP_006539634.1. Fig.3 Phylogenetic derived from multiple alignments of HSL amino acid sequences from various species The Accession No. of each HSL are as follows: Sinocyclocheilus rhinocerous, XP_016300015; Danio rerio, NP_001303654; Ictalurus punctatus, XP_017336708.1; Tachysurus fulvidraco, AIF72763.1; Cynoglossus semilaevis, XP_008329302.1; Paralichthys olivaceus, XP_019959978.1; Oreochromis niloticus, XP_003447301.1; Xiphophorus maculatus, XP_005811845.1; Salmo salar, XP_013997642.1; Oncorhynchus mykiss, NP_001184139.1; Esox lucius, XP_010899186.1; Clupea harengus, XP_012684999.1; Kryptolebias marmoratus, XP_017271065.1; Lates calcarifer, XP_018527967.1; Epinephelus coioides, AGU42438.1; Homo sapiens, XP_005258994.1; Mus musculus, XP_006539634.1. |
利用实时荧光定量PCR技术检测lipea基因在鲤10个不同组织中的表达模式和分布情况(图 4)。结果显示, 该基因在所有检测组织中均有表达, 但存在明显组织差异性。10个组织中基因相对表达量由高到低依次为腹腔脂肪、腹部肌肉、肠、脑、背部肌肉、心脏、脾、肝、肾和血液, 脂肪组织的表达量相对高于其他组织且差异极显著(P < 0.01), 腹部肌肉、肠和脑组织中的表达量相当, 差异不显著性(P > 0.05)。
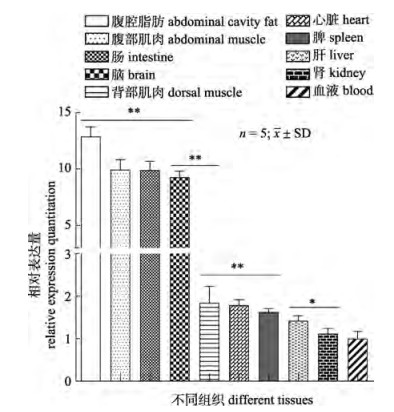
|
图 4 lipea基因在鲤不同组织中的相对表达 “**”表示极显著性差异(P < 0.01); “*”表示显著性差异(P < 0.05). Fig.4 The relative expression level of lipea in different tissues of common carp Cyprinus carpio "**" indicates the very significant difference (P < 0.01); "*" indicates significant difference (P < 0.05). |
lipea基因在鲤胚胎不同发育时期表达变化情况(图 5):随着胚胎的发育, lipea基因的表达水平呈现下降趋势, 8细胞期表达量最高, 相对其他时期相比具有显著性差异(P < 0.05);出膜期与出苗5 d的表达量相对较低, 两个时期表达量差异显著(P < 0.05);开口期的表达量最低。
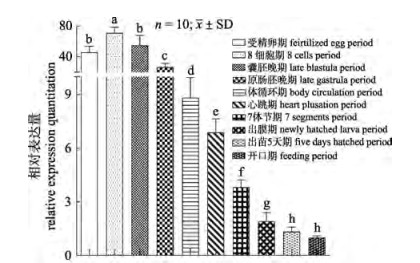
|
图 5 lipea基因鲤胚胎不同发育时期的表达规律 小写字母不同表示差异显著(P < 0.05). Fig.5 The expression of lipea in different development stages of common carp Cyprinus carpio embryos Different lowercase letters indicate significant differences (P < 0.05). |
lipea基因在脂肪含量极端差异个体表达情况(图 6)。在腹腔脂肪组织中, lipea基因表达量与腹腔脂肪量呈负相关且差异极显著性(P < 0.01);而lipea基因在背部肌肉组织中的表达模式与腹腔脂肪中不同, lipea基因表达量与背部肌肉脂肪含量呈正相关, 但差异不显著(P > 0.05)。
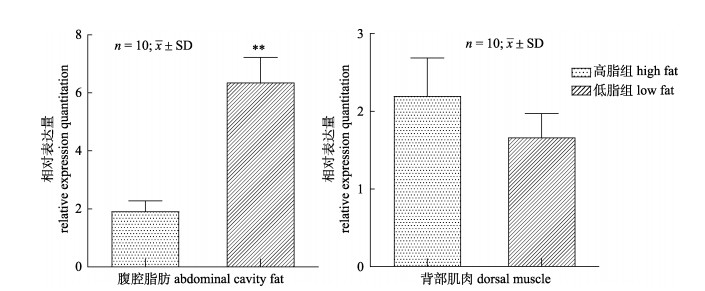
|
图 6 鲤lipea基因在不同脂肪含量组间的表达分析 “**”表示极显著性差异(P < 0.01);高脂组和低脂组平均脂肪含量分别为(9.56±0.72)%和(1.89±0.25)%. Fig.6 The expression analysis of lipea in different fat content groups of common carp Cyprinus carpio "**" indicates the very significant difference (P < 0.01), the average fat content of high and low fat group were (9.56±0.72)% and (1.89±0.25)%. |
本研究用RACE的方法获得了鲤的HSL1基因, 沿用斑马鱼同源基因的命名方式为命名为lipea。研究表明, HSL基因在诸多鱼类中存在两个拷贝, 且表达模式和功能也在差异。Kittilson等[11]首次从虹鳟鱼中克隆出两个HSL基因即HSL1和HSL2, 发现当鱼体饥饿的时候HSL1在肠系膜脂肪组织的表达量最高, HSL2则在幽门盲囊表达量最高, 虽然脂肪组织中的两个HSL基因表达量均会升高, 但HSL1与HSL2的表达模式却不同。牙鲆同样发现了HSL1和HSL2两个基因且表达模式也不相同。qRT-PCR检测结果显示, 牙鲆HSL1基因在附肢肌(脂肪存储部位)中的表达量最高而HSL基因在肝中表达量最高; 利用原位杂交技术也证实HSL1基因在附肢肌的表达量远高于HSL2基因[10]。同样, 对黄颡鱼[12]和真鲷[13]等的研究中均获得两个HSL基因并验证了其表达模式也不相同。由此推测, 在鱼类HSL1基因主要参与脂类代谢过程而HSL2相比脂解作用则另有其他功能。本文主要针对鲤HSL1基因的时空表达特征及其与脂肪沉淀的关系开展研究。
利用qRT-PCR方法检测了lipea基因在鲤不同组织中的表达分布和不同发育时期的表达规律。lipea基因在鲤各组织中均有表达, 包括脂肪、肌肉、脑、肠、肝等组织, 在脂肪组织中表达量最高, 这与已报道的物种具有一致性[11-14]。虽然HSL在非脂肪组织中的作用尚不清楚, 但HSL在小鼠[20]和虹鳟[11]骨骼肌中发挥脂解作用。本研究中lipea在鲤腹部肌肉中均有较高表达量, 其中腹部肌肉表达量仅次于脂肪组织, 推测lipea在鲤肌肉中同样发挥脂解作用。HSL在胚胎发育时期的表达规律研究尚未见报道。本研究结果显示, lipea在胚胎发育早期表达量较高, 随后逐渐下降。8细胞时期表达量最高, 原肠胚晚期后表达量迅速下降, 是因为卵黄囊中含有大量母源性脂类为胚胎发育初期提供细胞结构成分和初级供能[21]。原肠胚时期是胚胎发生中一个极为重要的阶段, 这个时期初步形成了内、中、外三个胚层, 大量的膜结构形成, 原肠胚时期前需要积累大量的游离脂肪酸和能量, 推测在胚胎早期发育阶段所需的大量的游离脂肪酸可能是由HSL基因的脂解作用下产生的。胡先成等[22]发现河川沙塘鳢(Odontobutis potamophila)胚胎及仔鱼发育过程中总脂肪含量呈下降趋势。Desvilettes[23]等研究了白斑狗鱼胚胎发育过程中脂类的变化, 结果显示在白斑狗鱼胚胎发育过程中脂质下降了41.3%, 甘油三酯下降了58.1%。本研究尚未测定胚胎及仔鱼发育过程中脂肪含量变化情况, 因此关于胚胎不同发育时期lipea基因的表达与脂肪含量间的调控关系的还需进一步研究。
HSL是调控脂肪组织分解的最关键因素, 也是影响动物脂肪沉积的关键酶之一[1]。Kazala等[24]研究表明, 牛肌肉中HSL活性与肌内脂肪含量存在相关性, 可作为牛肌内脂肪含量选育的指标。因HSL基因在脂肪代谢过程中的重要作用, 近年来通常作为脂肪沉积的候选基因。诸多学者开展了HSL基因表达量与肌内脂肪含量的相关性研究, 但不同物种研究结果不尽相同[9]。Qiao等[8]研究发现, HSL基因表达量与哈萨克羊肌内脂肪含量呈显著负相关(P < 0.05), 但与新疆细毛羊(Ovis aries Xinjiang)肌内脂肪含量无显著的相性(P > 0.05)。栾兆进等[25]在绵羊中研究显示, HSL表达量与肌内脂肪含量呈负相关, 但差异不显著(P > 0.05)。然而, 李广经[26]在黑鲷(Acanthopagrus schlegelii)中的研究显示, HSL基因表达量与脂肪含量呈正相关。陈杰等[27]研究结果显示, HSL基因表达水平与苏太猪肌内脂肪含量之间相关性不显著(P > 0.05)。本研究中, lipea基因表达量与鲤背部肌肉脂肪含量呈现正相关, 但两组间基因的表达差异不显著性(P > 0.05), 与其他物种中的报道既有一致性也存在一定差异。不同物种间结果差异可能是不同的物种利用脂肪酸的方式和途径有所不同导致。
甘油三酯(TG)是动物体内脂肪的主要成分, 动物的脂肪组织是其脂肪沉积的主要场所, HSL是脂肪水解为游离脂肪酸和甘油过程中的关键限速酶, 其表达量的升高会显著降低TG在脂肪细胞中的沉积[28]。苗志国等[29]研究发现较低的HSL活性的淮南猪体内脂肪的沉积能力强, 本研究中也发现lipea基因表达水平与腹腔脂肪量呈负相关, 且差异极显著(P < 0.01)。脂肪沉积是一个复杂的生理和生化过程, 由代谢酶调节, 受到脂肪摄取、合成和分解等信号通路中相关基因的调控[9]。同时, 脂肪沉积受多个因素的影响, 如遗传因素、营养水平、环境因素和性别等[30], 在育种工作中需要注重各种影响因素的作用机理、各因素间的协同与互作关系, 尤其是基因间的功能和互作的探索, 以期找到最佳的控制鱼类脂肪沉积的管理模式。
| [1] |
Yeaman S J. Hormone-sensitive lipase—a multipurpose enzyme in lipid metabolism[J]. Biochimica et Biophysica Acta: Molecular Cell Research, 1990, 1052(1): 128-132. DOI:10.1016/0167-4889(90)90067-N |
| [2] |
Kraemer F B, Shen W J. Hormone-sensitive lipase knockouts[J]. Nutrition & Metabolism, 2006, 3(1): 12-19. |
| [3] |
Holm C, Kirchgessner T G, Svenson K L, et al. Hormone-sensitive lipase: sequence, expression and chromosomal localization to 19 cent-q13.3[J]. Science, 1988, 241(4872): 1503-1506. DOI:10.1126/science.3420405 |
| [4] |
Langin D, Laurell H, Hols L S, et al. Gene organization and primary structure of human hormone-sensitive lipase: possible significance of a sequence homology with a lipase of Moraxella TA144, an antarctic bacterium[J]. Proceedings of the National Academy of Sciences of the United States of America, 1993, 90(11): 4897-4901. DOI:10.1073/pnas.90.11.4897 |
| [5] |
Li Z, Sumida M, Birchbauer A, et al. Isolation and characterization of the gene for mouse hormone-sensitive lipase[J]. Genomics, 1994, 24(2): 259-265. DOI:10.1006/geno.1994.1614 |
| [6] |
Harbitz I, Langset M, Ege A G, et al. The porcine hormone-sensitive lipase gene: sequence, structure, polymorphisms and linkage mapping[J]. Animal Genetics, 1999, 30(1): 10-15. DOI:10.1046/j.1365-2052.1999.00412.x |
| [7] |
Ren S M, Zhao X T, Zhou C B, et al. Correlation of HSL gene polymorphisms with carcass traits and meat quality in pig breed Sujiang and its crossbreeds[J]. Hunan Agricultural Sciences, 2014(13): 55-57. [任善茂, 赵旭庭, 周春宝, 等. 猪HSL基因多态性与胴体性状及肌肉品质的相关性分析[J]. 湖南农业科学, 2014(13): 55-57. DOI:10.3969/j.issn.1006-060X.2014.13.018] |
| [8] |
Qiao Y, Huang Z G, Li Q F, et al. Developmental changes of the FAS and HSL mRNA expression and their effects on the content of intramuscular fat in Kazak and Xinjiang sheep[J]. Journal of Genetics and Genomics, 2007, 34(10): 909-917. DOI:10.1016/S1673-8527(07)60102-7 |
| [9] |
Wang Y P, Xu C C, Luo H L. Research progress in relevant candidate genes of animal intramuscular fat deposition[J]. Chinese Journal of Animal Nutrition, 2017, 29(5): 1475-1480. [王怡平, 徐晨晨, 罗海玲. 动物肌内脂肪沉积相关候选基因的研究进展[J]. 动物营养学报, 2017, 29(5): 1475-1480.] |
| [10] |
Khieokhajonkhet A, Kaneko G, Ohara K, et al. Hormone-sensitive lipase in Japanese flounder Paralichthys olivaceus: the potential function of the inclinator muscle of fin as a lipid storage site[J]. Fisheries Science, 2014, 80(2): 341-351. DOI:10.1007/s12562-013-0695-5 |
| [11] |
Kittilson J D, Reindl K M, Sheridan M A. Rainbow trout (Oncorhynchus mykiss) possess two hormone-sensitive lipase-encoding mRNAs that are differentially expressed and independently regulated by nutritional state[J]. Comparative Biochemistry and Physiology Part A: Molecular & Integrative Physiology, 2011, 158(1): 52-60. |
| [12] |
Chen Q L, Luo Z, Song Y F, et al. Hormone-sensitive lipase in yellow catfish Pelteobagrus fulvidraco: Molecular characterization, mRNA tissue expression and transcriptional regulation by leptin in vivo and in vitro[J]. General and Comparative Endocrinology, 2014, 206: 130-138. DOI:10.1016/j.ygcen.2014.06.031 |
| [13] |
Khieokhajonkhet A, Kaneko G, Hirano Y, et al. Different effects of growth hormone and fasting on the induction patterns of two hormone-sensitive lipase genes in red seabream Pagrus major[J]. General and Comparative Endocrinology, 2016, 236: 121-130. DOI:10.1016/j.ygcen.2016.06.025 |
| [14] |
Han C Y, Wen X B, Zheng Q M, et al. Effects of dietary lipid levels on lipid deposition and activities of lipid metabolic enzymes in hybrid tilapia (Oreochromis niloticus ×O. aureus)[J]. Journal of Animal Physiology and Animal Nutrition, 2011, 95(5): 609-615. DOI:10.1111/jpn.2011.95.issue-5 |
| [15] |
Ai Q H, Yuan Y H, Mai K S, et al. Lipolysis related genes clone and regulation by dietary lipid level in half-smooth tongue sole (Cynoglossus Semilaevis) larvae[J]. Periodical of Ocean University of China, 2014, 44(10): 65-71. [艾庆辉, 袁禹惠, 麦康森, 等. 半滑舌鳎稚鱼脂肪分解关键酶基因的克隆及饲料中脂肪水平对其表达的调控[J]. 中国海洋大学学报:自然科学版, 2014, 44(10): 65-71.] |
| [16] |
Zhang Y G, Wang Q R, Wang Z Y. Research advance on flesh quality improvement and evaluation of farmed fish[J]. Journal of Fujian Fisheries, 2015, 37(2): 172-178. [张亚光, 王秋荣, 王志勇. 养殖鱼类肉质改良与评价的研究进展[J]. 福建水产, 2015, 37(2): 172-178.] |
| [17] |
Robb D, Kestin S C, Warriss P D, et al. Muscle lipid content determines the eating quality of smoked and cooked Atlantic salmon (Salmo salar)[J]. Aquaculture, 2002, 205(3-4): 345-358. DOI:10.1016/S0044-8486(01)00710-4 |
| [18] |
Du Z Y. Causes of fatty liver in farmed fish: a review and new perspectives[J]. Journal of Fisheries of China, 2014, 38(9): 1628-1638. [杜震宇. 养殖鱼类脂肪肝成因及相关思考[J]. 水产学报, 2014, 38(9): 1628-1638.] |
| [19] |
Zheng X H, Kuang Y Y, Lv W H, et al. Genome-wide association study for muscle fat content and abdominal fat traits in common carp (Cyprinus carpio)[J]. PLoS ONE, 2016, 11(12): e0169127. DOI:10.1371/journal.pone.0169127 |
| [20] |
Haemmerle G, Zimmermann R, Strauss J G, et al. Hormone-sensitive lipase deficiency in mice changes the plasma lipid profile by affecting the tissue-specific expression pattern of lipoprotein lipase in adipose tissue and muscle[J]. Journal of Biological Chemistry, 2002, 277(15): 12946-12952. DOI:10.1074/jbc.M108640200 |
| [21] |
Fraher D, Sanigorski A, Mellett N A, et al. Zebrafish embryonic lipidomic analysis reveals that the yolk cell is metabolically active in processing lipid[J]. Cell Reports, 2016, 14(6): 1317-1329. DOI:10.1016/j.celrep.2016.01.016 |
| [22] |
Hu X C, Zhao Y L. Variation in the lipid content and fatty acid composition during embryonic and larval development of Odontobutis potamophila[J]. Freshwater Fisheries, 2008, 38(3): 46-50. [胡先成, 赵云龙. 河川沙塘鳢胚胎、仔鱼发育过程中脂类含量及脂肪酸组成的变化[J]. 淡水渔业, 2008, 38(3): 46-50. DOI:10.3969/j.issn.1000-6907.2008.03.009] |
| [23] |
Desvilettes C, Bourdier G, Breton J C. Changes in lipid class and fatty acid composition during development in pike (Esox lucius L) eggs and larvae[J]. Fish Physiology and Biochemistry, 1997, 16(5): 381-393. DOI:10.1023/A:1007764130651 |
| [24] |
Kazala E C, Petrak J L, Lozeman F J, et al. Hormone-sensitive lipase activity in relation to fat content of muscle in wagyu hybrid cattle[J]. Livestock Production Science, 2003, 79(1): 87-96. DOI:10.1016/S0301-6226(02)00141-0 |
| [25] |
Luan Z J, Liu K D, He J N, et al. The FAM134B, PPARγ, HSL and FAS gene expression patterns and their asociation with intramuscular fat content in shep[J]. Acta Veterinaria et Zotechnica Sinica, 2016, 47(12): 2379-2389. [栾兆进, 刘开东, 贺建宁, 等. 绵羊FAM134B、PPARγ、HSL和FAS基因表达量及与肌内脂肪含量的关系[J]. 畜牧兽医学报, 2016, 47(12): 2379-2389.] |
| [26] |
Li G J. Effect of dietary and carbohydrates on enzymes activities and relative gene expression of FAS and HSL in juvenile black sea bream, Acanthopagrus schlegelii[D]. Hangzhou: Zhejiang University, 2015. [李广经.不同脂肪和碳水化合物水平对黑鲷脂肪代谢关键酶FAS和HSL活性和基因表达的影响研究[D].杭州: 浙江大学, 2015.] http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y2882528
|
| [27] |
Chen J, Yang X J, Tong H, et al. Expressions of FAS and HSL mRNA in longissimus dorsi muscle and their relation to intramuscular fat contents in pig[J]. Journal of Agricultural Biotechnology, 2004, 12(4): 422-426. [陈杰, 杨晓静, 佟辉, 等. FAS和HSL mRNA在猪背最长肌的表达及其与肌内脂肪含量的关系[J]. 农业生物技术学报, 2004, 12(4): 422-426. DOI:10.3969/j.issn.1674-7968.2004.04.014] |
| [28] |
Sztalryd C, Komaromy M C, Kraemer F B. Overexpression of hormone-sensitive lipase prevents triglyceride accumulation in adipocytes[J]. Journal of Clinical Investigation, 1995, 95(6): 2652-2661. DOI:10.1172/JCI117967 |
| [29] |
Miao Z G, Xie H B, Wang Y Q, et al. Difference in fat metabolic enzymes and serum adiponectin between Huainan and Landrace pig[J]. Guizhou Agricultural Sciences, 2018, 46(1): 61-63. [苗志国, 谢红兵, 王永强, 等. 淮南猪与长白猪脂肪代谢酶及脂联素的发育差异[J]. 贵州农业科学, 2018, 46(1): 61-63. DOI:10.3969/j.issn.1001-3601.2018.01.017] |
| [30] |
Liu J Z, Xing J Y, Cao S D, et al. Analysis of genetic and nutritious factors affecting domestic animal fat deposition[J]. Livestock and Poultry Industry, 2008(12): 26-29. [刘京贞, 邢晋祎, 曹善东, 等. 影响畜禽脂肪沉积的遗传和营养因素分析[J]. 畜禽业, 2008(12): 26-29. DOI:10.3969/j.issn.1008-0414.2008.12.009] |
 2019, Vol. 26
2019, Vol. 26

