2. 水产科学国家级实验教学示范中心, 上海海洋大学, 上海 201306;
3. 海洋生物科学国际联合研究中心, 中国科学技术部, 上海海洋大学, 上海 201306
2. National Demonstration Center for Experimental Fisheries Science Education, Shanghai Ocean University, Shanghai 201306, China;
3. International Research Center for Marine Biosciences at Shanghai Ocean University, Ministry of Science and Tech-nology, Shanghai 201306, China
DNA甲基化是发生于DNA胞嘧啶第五个碳原子上的一种共价修饰, 作为一种表观遗传标记, DNA甲基化已经被证明与基因组功能、基因转录以及X染色体失活相关[1-2]。在脊椎动物中, DNA甲基化主要发生在鸟嘌呤前的胞嘧啶位点上或者在CpG岛上, 研究发现在人类和小鼠胚胎干细胞中, DNA非CpG岛区域也存在甲基化修饰, 然而这种非CpG位点的甲基化在成熟组织中往往会被丢失[3]。越来越多的研究发现环境压力能够改变基因组甲基化水平, 如磷饥饿[4]以及热压力[5]。在植物中, DNA甲基化调控胁迫应答基因的表达使植物适应外界环境变化[6-7], 在昆虫中研究发现, DNA甲基化的改变使得昆虫适应温度的变化[8]。然而, 在鱼类中DNA甲基化与冷适应之间的关系尚未研究清楚。
活性氧(reactive oxygen species, ROS)如超氧阴离子(O2–)和过氧化氢(H2O2)等, 是需氧细胞在代谢中产生的电子还原产物。在高浓度时, ROS很容易与蛋白质、脂类、碳水化合物以及核苷酸反应, 这些反应通常包括不可逆的功能的改变, 甚至是彻底的破坏[9]。已有研究证明, 短期低温压力下斑马鱼ZF4细胞中ROS水平明显上升并且产生DNA双链断裂[10]。在植物中研究发现, 低温压力下ROS作为一种信号分子能激活细胞转录调控从而调控下游基因的表达, 最终使得植物适应低温[11]。在人结肠癌细胞中研究发现ROS能在启动子CpG岛产生氧化损伤, 损伤部位能够募集DNA甲基转移酶1(DNMT1)等表观酶类[12], 导致DNA发生甲基化, 从而抑制下游基因的转录[13]。ATM是应答DNA双链断裂的一个重要的调控因子, 控制着一个复杂的生化网络, 能够直接或间接影响DNA损伤修复[14]。研究证明ROS能够激活共济失调–毛细血管扩张症突变蛋白(ataxia- telangiectasia mutated, ATM)[15]。但是活性氧导致的氧化损伤与低温鱼类细胞甲基化水平改变是否有关尚不明确。
斑马鱼(Danio rerio)由于易饲养、繁殖速度快且基因组全序列已经公布成为一种广泛应用于分子生物学、遗传学的模式生物[16]。斑马鱼成纤维细胞ZF4细胞系是来源于发育1 d的斑马鱼胚胎, 常用于研究硬骨鱼类冷适应过程中基因表达调控[17]以及甲基化水平变化[18]。本研究探究了不同时间点(3 d、5 d和30 d), 和不同温度条件下(18℃和10℃), 斑马鱼ZF4细胞DNA甲基化水平变化及其与低温下氧化损伤之间的关系, 为后期斑马鱼细胞低温胁迫分子机制的研究奠定基础。
1 材料与方法 1.1 细胞斑马鱼胚胎成纤维细胞ZF4购于American Type Culture Collection (ATCC), 采用DMEM/F- 12培养基[(含2.50 mmol/L谷氨酰胺(Hyclone), 青霉素(100 U/mL)和链霉素(100 mg/mL)(Hyclone)、10%的胎牛血清(Gibco)], 于28℃、95%湿度、5%CO2的培养箱中进行培养。每隔2 d换液, 当细胞长至90%用不含有EDTA的0.25%胰蛋白酶(Gibco)进行消化, 1 : 2传代后加入培养基继续培养, 实验所用的细胞均为4~6代。
1.2 试剂和仪器DMEM/F-12液体培养基(Hyclone, SH30023.01B)、胎牛血清和胰蛋白酶购于Gibco公司; 谷氨酰胺L-glutamine (Hyclone, SH30034.01)和杜氏磷酸缓冲液DPBS购于Thermo公司; KU- 55933和二甲基亚砜DMSO购于Sigma公司; 限制性内切酶MspⅠ和HpaⅡ购于NEB公司; 甲基化检测试剂盒5-mC DNA ELISA Kit (D5325)购于Zymo Research公司; 其他常用试剂均为国产分析纯。
主要仪器:倒置荧光显微镜(Zeiss)、细胞低温培养箱(Galaxy170R, Eppendorf)、多功能酶标仪(BioTek)、电泳仪(Bio-Rad)。
1.3 细胞低温及药物处理 1.3.1 细胞低温处理取生长状态良好的野生型ZF4细胞, 实验前1 d按汇合度70%接种于100 mm培养皿中, 28℃培养24 h后, 将细胞放到低温培养箱中, 18℃低温培养3 d、5 d、30 d, 10℃低温培养3 d、5 d。利用倒置显微镜观察细胞形态并拍照。
1.3.2 N-乙酰半胱氨酸(NAC)处理细胞取对数期ZF4细胞提前1 d接种至100 mm培养皿中, 28℃培养24 h, 待汇合度至70%~90%, 加入终浓度2 mmol/L的NAC于28℃预处理2 h后放到低温培养箱, 18℃处理3 d、5 d, 每天都需要补充NAC。
1.3.3 KU-55933处理细胞取对数期ZF4细胞提前1 d接种至100 mm培养皿中, 28℃培养24 h, 待汇合度至70%~90%, 参照Chwastek等[19]的研究结果, 选取终浓度为1 μmol/L和10 μmol/L的KU-55933于28℃预处理1 h后放到低温培养箱18℃处理3 d、5 d, 每天都需要补充KU- 55933。
1.4 细胞浓度与比生长速率测定28℃培养的细胞分为两组, 接种于12孔板中, 24 h后一组置于28℃培养作为常温对照组, 另一组置于18℃培养7 d作为18℃短期培养组; 另外将18℃低温驯化30 d的细胞也接种至12孔板中, 24 h后置于18℃培养箱中培养7 d作为18℃长期培养组。每天将细胞用胰酶消化收集, 用磷酸缓冲液(PBS)稀释至适当浓度, 加入0.4%(wt/v)台盼蓝进行活细胞染色, 吹打均匀后用血球计数板进行计数。
1.5 限制性内切酶检测DNA甲基化水平利用两种限制性内切酶MspⅠ和HpaⅡ对基因组DNA进行酶切, MspⅠ和HpaⅡ DNA识别位点都是CCGG, 但是具有不同的甲基化敏感性: HpaⅡ不能对发生甲基化的DNA进行酶切而MspⅠ可以。用酚氯仿法提取出细胞基因组DNA, 取1 μg基因组DNA, 分别用MspⅠ和HpaⅡ于37℃水浴锅中酶切过夜。酶切后的DNA用0.8%琼脂糖凝胶进行电泳分析。利用灰度分析软件, 通过比较HpaⅡ/MspⅠ的比值分析各个样品间的相对甲基化水平[20]。
1.6 酶联免疫吸附剂测定DNA甲基化水平利用酚氯仿法提取细胞基因组DNA, 后按照Zymo Research公司的5-mC DNA ELISA试剂盒(5-mC DNA ELISA Kit, D5325, Zymo Research)说明书检测基因组DNA甲基化水平。
1.7 数据统计分析比生长速率(μ)通过以下公式计算:
| $ \mu = \frac{1}{{{C_{\rm{v}}}}} \times \frac{{{\rm{d}}{C_{\rm{v}}}}}{{{\rm{d}}t}} $ |
其中, Cv为活细胞密度(106 cells/mL), t为培养时间(day)。
所有实验结果均是来自至少3次独立实验, 图中所示结果均为均值±标准差(x±SD)。t-test法用于检测低温处理的显著性, 两两之间的显著差异(P < 0.05)利用Turkey’s多重比较来检测。
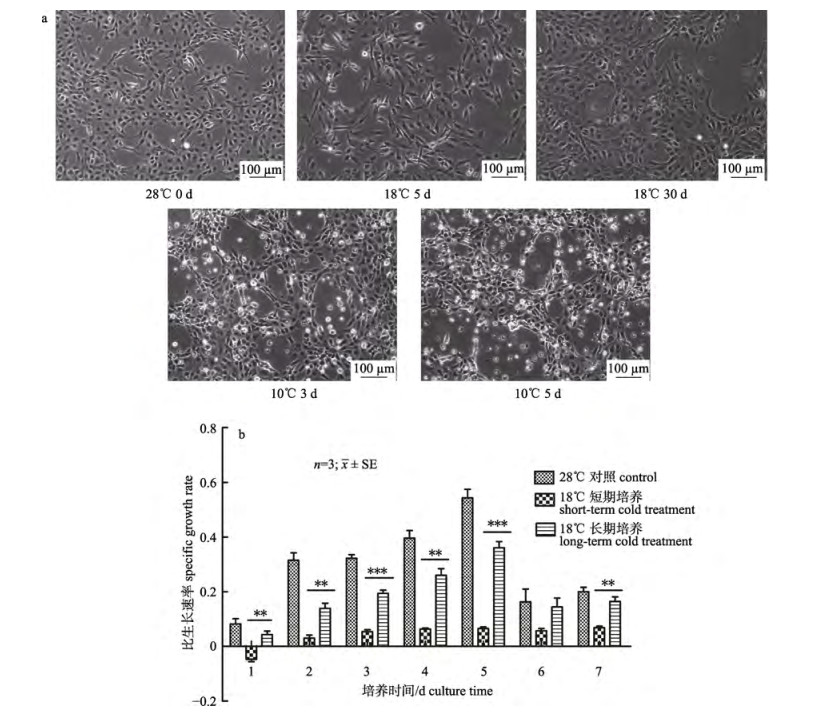
2 结果与分析 2.1 细胞低温处理后形态以及比生长率变化用倒置相差显微镜观察细胞可以发现, 在正常培养温度下(28℃)ZF4细胞呈不规则三角形, 细胞核为卵圆形, 胞质突起, 细胞边缘清晰。在18℃低温处理5 d后, 细胞形态无显著变化, 有少量细胞脱落。在18℃低温处理30 d后, 可以看见细胞边缘模糊, 大部分细胞不再是不规则三角形, 细胞核形态不规则。在10℃处理处理3 d后, ZF4细胞形状从不规则三角形收缩为长条状, 细胞间空隙变大, 部分细胞死亡。而在10℃处理5 d后, 细胞大量死亡漂浮在培养基中(图 1a)。
|
图 1 细胞在不同温度和时间点下细胞形态图以及比生长速率 a.细胞在常温28℃、低温18℃处理5 d、30 d以及低温10℃处理3 d、5 d的细胞形态图; b.细胞在常温(28℃)培养以及短期和长期低温(18℃)培养下的比生长速率. *表示短期培养组与长期培养组相比差异显著, *表示P < 0.05, **表示P < 0.01, ***表示P < 0.001. Fig.1 Cell morphology and specific growth rate at different temperatures and time points a. The cellular morphology at normal temperature (28℃), low temperature (18℃) for 5 d, 30 d, and 10℃ for 3 d, 5 d. b. Changes of specific growth rate (µ) of cells cultured at normal temperature (28℃) or low temperature (18℃) for short and long periods. * denotes significant differences between short-term cold treatment group and long-term cold treatment group; *means P < 0.05; ** means P < 0.01; *** means P < 0.001. |
对低温18℃下细胞进行比生长速率测定发现, 28℃细胞以及18℃下处理30 d的细胞在接种后4~5 d都能达到最大比生长速率, 分别为0.568和0.355。而18℃下短期处理的细胞贴壁后第1天有部分细胞死亡, 比生长速率为负, 之后的第2~7天比生长速率也较低, 在0.06左右。并且从图中可以看出, 相同时间点下, 18℃长期培养的细胞的比生长速率显著高于18℃短期培养组(P < 0.05) (图 1b)。
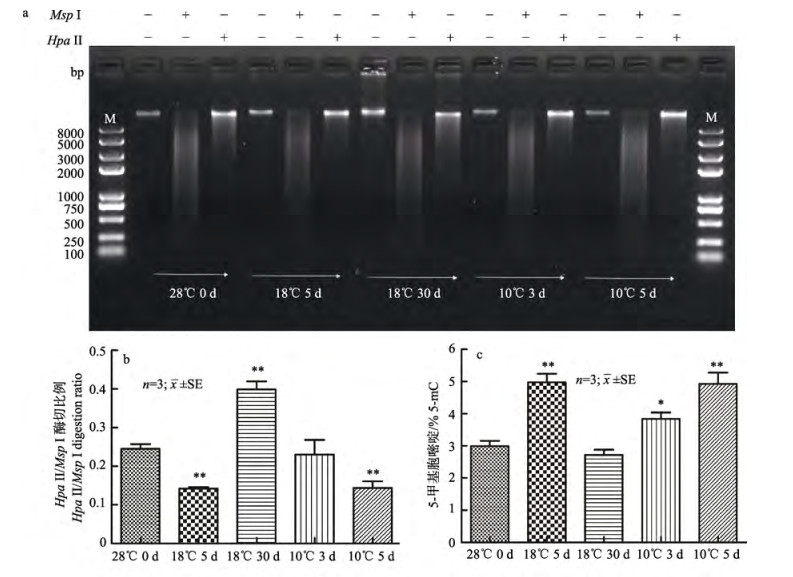
2.2 短期低温下基因组DNA甲基化水平上升与28℃下培养的细胞相比, 无论是在18℃还是10℃, 短期低温压力都能导致细胞基因组甲基化水平上升, 而在18℃低温处理30 d后, 细胞基因组甲基化水平明显下降(P < 0.05)(图 2a、图 2b)。用5-mC DNA ELISA试剂盒检测同样证明相比于对照组(28℃), 18℃处理5 d细胞其DNA甲基化水平上升而18℃处理30 d细胞的DNA甲基化水平下降(图 2c)。
|
图 2 不同温度条件下DNA甲基化水平变化 a.各个温度和时间点下限制性内切酶MspⅠ和HpaⅡ酶切电泳图; M: Trans 2K® Plus Ⅱ DNA Ladder; b.各个温度和时间点下, DNA甲基化水平量化为甲基化敏感酶HpaⅡ相对于甲基化不敏感同裂酶MspⅠ酶切比例; c.不同实验条件下, 利用5-mC甲基化试剂盒检测DNA甲基化水平变化. *表示低温处理组(18℃ 5 d, 18℃ 30 d, 10℃ 3 d, 10℃ 5 d)与对照组(28℃)相比差异显著, *表示P < 0.05, **表示P < 0.01, ***表示P < 0.001. Fig.2 Changes of DNA methylation level at different temperatures a. Electrophoretogram of digestion of genomic DNA with restriction endonucleases Msp Ⅰ and Hpa Ⅱ; M: Trans 2K® Plus Ⅱ DNA Ladder; b. For ZF4 cells under different temperatures and time points, DNA methylation levels are quantified as the relative amount of DNA digestion by the methylation-sensitive enzyme Hpa Ⅱ relative to its methylation-insensitive isoschizomer Msp Ⅰ. c. Using 5-mC methylation kit to detect intracellular DNA methylation levels under different experimental conditions. *denotes significant differences between low temperature treatment groups (18℃ 5 d, 18℃ 30 d, 10℃ 3 d, 10℃ 5 d) and the control group (28℃); *means P < 0.05; ** means P < 0.01; *** means P < 0.001. |
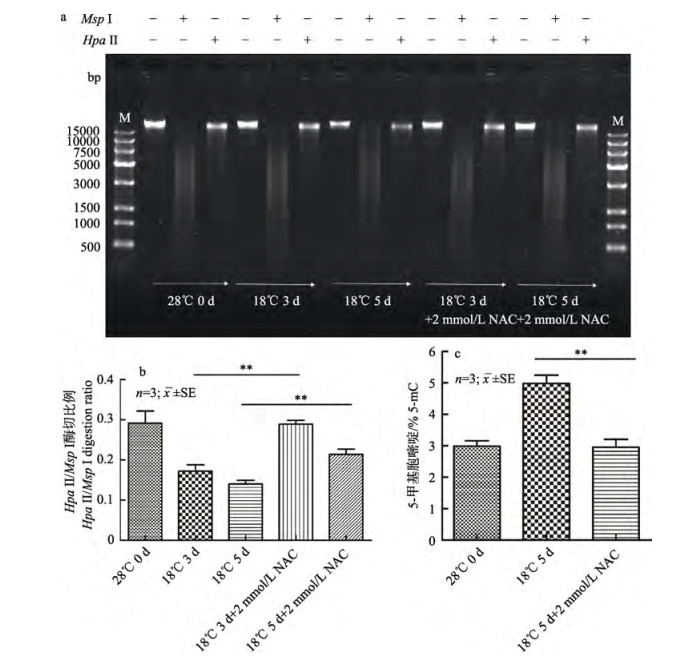
前期研究已经证明低温能够诱导斑马鱼ZF4细胞产生活性氧, 并且在低温(10℃和18℃)处理5 d时ROS水平上升最为显著。为了证明短期低温下基因组DNA甲基化水平上升与ROS有关, 本研究用抗氧化剂N-乙酰半胱氨酸(NAC)对低温处理的细胞进行预处理。从图中可以看出, 相对于未预处理NAC的细胞, 预处理NAC后细胞的甲基化水平显著下降(P < 0.05)(图 3a、图 3b)。用5-mC DNA ELISA试剂盒检测同样也可以发现, 相对于未用NAC预处理的18℃ 5 d细胞, 预处理NAC后, DNA甲基化水平明显下降(图 3c)。这说明短期低温压力下细胞内DNA甲基化水平上升与活性氧产生相关。
|
图 3 短期低温下NAC预处理后DNA甲基化水平变化 a.不同实验条件下限制性内切酶MspⅠ和HpaⅡ酶切电泳图; M: Trans 15K DNA Marker; b.不同实验条件下, 甲基化敏感酶HpaⅡ相对于甲基化不敏感同裂酶MspⅠ酶切比例; c.不同实验条件下, 利用5-mC甲基化试剂盒检测DNA甲基化水平变化. *表示低温处理加药组与未加药组相比差异显著, *表示P < 0.05, **表示P < 0.01, ***表示P < 0.001. Fig.3 NAC pretreatment inhibited DNA methylation induced under short-term cold stress a. Electrophoretogram of digestion of genomic DNA with restriction endonucleases Msp Ⅰ and Hpa Ⅱ; M: Trans 15K DNA Marker; b. For ZF4 cells under different experimental conditions, DNA methylation levels are quantified as the relative amount of DNA digestion by the methylation-sensitive enzyme Hpa Ⅱ relative to its methylation-insensitive isoschizomer Msp Ⅰ; c. Using 5-mC methylation kit to detect intracellular DNA methylation levels under different experimental conditions. *denotes significant differences between the drug treatment group and the untreated group under short-term cold stress; *means P < 0.05; ** means P < 0.01; *** means P < 0.001. |
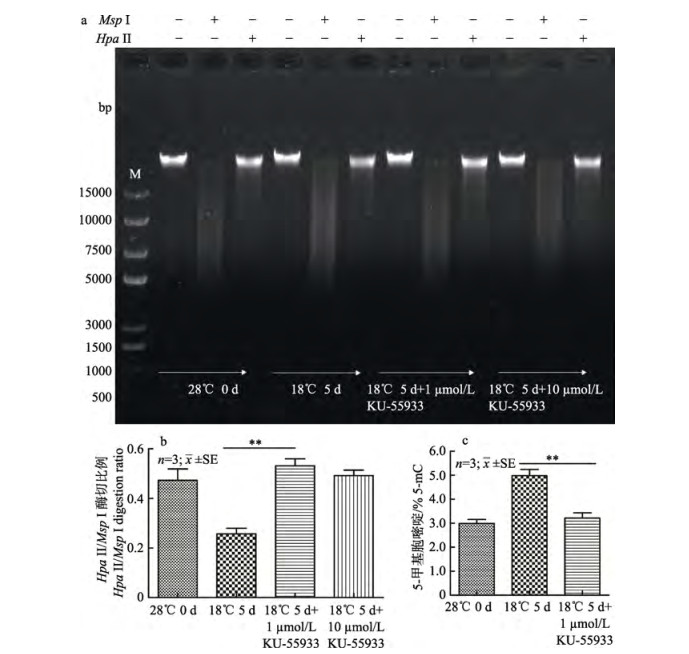
共济失调–毛细血管扩张突变蛋白(ATM)属于磷脂酰肌醇-3-激酶相关激酶家族, 是DNA损伤的感应器。ATM能够将DNA损伤信息传递到下游修复蛋白, 从而启动DNA修复机制, 维持细胞基因组的完整性。为了探究短期低温下DNA甲基化与DNA损伤修复的直接关系, 在细胞低温处理前用一种特异的ATM抑制剂KU-55933对细胞进行预处理。从图中可以看出, 短期低温胁迫下(18℃ 5 d), 相对于没有用KU-55933进行预处理的细胞, 预处理1 mmol/L KU-55933后, 细胞内DNA甲基化水平显著降低(P < 0.05)(图 4a、图 4b), 另外使用10 μmol/L的KU-55933处理与1 μmol/L相比DNA甲基化水平没有显著差异, 说明对于ZF4细胞系1 μmol/L KU-55933即可有效抑制ATM(图 4a)。同样地, 用5-mC DNA ELISA试剂盒检测发现, 同样实验温度和时间点下, 预处理KU-55933的细胞DNA甲基化水平相比于对照组明显下降(图 4c)。这说明了短期低温压力下细胞内甲基化水平上升与DNA损伤修复相关。
|
图 4 短期低温下KU-55933预处理后细胞内DNA甲基化水平变化 a.不同实验条件下限制性内切酶MspⅠ和HpaⅡ酶切电泳图; M: Trans 15K DNA Marker; b.不同实验条件下, 甲基化敏感酶HpaⅡ相对于甲基化不敏感同裂酶MspⅠ酶切比例; c.不同实验条件下, 利用5-mC甲基化试剂盒检测DNA甲基化水平变化. *表示短期低温胁迫下, 加药组与未加药组相比差异显著, *表示P < 0.05, **表示P < 0.01, ***表示P < 0.001. Fig.4 Changes of DNA methylation level of KU-55933 pretreatment under short-term low temperature stress a. Electrophoretogram of digestion of genomic DNA with restriction endonucleases Msp Ⅰ and Hpa Ⅱ; M: Trans 15K DNA Marker; b: For ZF4 cells under different experimental conditions, DNA methylation levels are quantified as the relative amount of DNA digestion by the methylation-sensitive enzyme Hpa Ⅱ relative to its methylation-insensitive isoschizomer Msp Ⅰ; c. Using 5-mC methylation kit to detect intracellular DNA methylation levels under different experimental conditions. *denotes significant differences between the drug treatment group and the untreated group under short-term cold stress; * means P < 0.05; ** means P < 0.01; *** means P < 0.001. |
之前研究中, 研究者们对斑马鱼实施18℃[21]、12℃[22]或者10℃[23]等的低温胁迫, 进而研究鱼类中一些与温度耐受相关基因的表达。许琼琼等[10]分别在18℃和10℃下处理斑马鱼细胞1 d、3 d和5 d发现低温胁迫能够诱导斑马鱼细胞ROS产生。另外, Han等[18]发现, 斑马鱼细胞在18℃短期培养下DNA甲基化水平上升, 而18℃长期培养下DNA甲基化水平下降。已有研究证实, ROS能够诱导DNA损伤进而发生DNA甲基化[13]。因此, 本研究参照之前的研究体系[17], 选取了18℃和10℃这两个温度点对斑马鱼细胞进行低温处理, 探究低温下DNA甲基化水平上升与ROS产生之间的联系。18℃和10℃这两个温度点能够典型的反映出斑马鱼ZF4细胞在低温胁迫下的生理状态:斑马鱼ZF4细胞在18℃低温胁迫下能够很好地适应, 而在10℃下培养5 d后有大多数细胞死亡漂浮在培养基中(图 1a)。并且, 计算斑马鱼细胞在18℃下比生长速率可以发现, 在18℃长期培养的细胞的比生长速率远远高于短期培养的细胞(图 1b), 反映出在18℃长期培养过程中, 细胞表现出对外界低温压力的适应性。
利用限制性内切酶对DNA进行酶切发现, 相对于28℃培养的细胞, 18℃处理5 d的细胞DNA甲基化水平明显上升, 而18℃处理30 d的细胞DNA甲基化水平反而下降(图 2), 这些都说明斑马鱼细胞在18℃下培养时存在一个低温适应的过程, 并且DNA甲基化可能参与了细胞低温适应过程。为研究低温胁迫下DNA甲基化与ROS之间的关系, 利用ROS抑制剂NAC对细胞进行预处理, 发现短期低温压力下抑制了ROS产生的同时DNA甲基化水平也会相应降低(图 3), 这说明了在斑马鱼低温适应过程中DNA甲基化水平的升高和ROS产生密切相关。
ATM是作为直接感受DNA双链断裂损伤并起始诸多DNA损伤信号反应通路的主开关分子, 调控了DNA损伤修复过程中一系列信号通路。有研究发现ROS能够激活ATM[15], 而ATM能够通过调控视网膜母细胞瘤肿瘤抑制基因(Rb)从而引起DNA甲基化[24]。同样在斑马鱼ZF4细胞中, 研究发现短期低温压力下利用KU-55933抑制ATM后, DNA甲基化水平出现下降(图 4), 这说明短期低温压力下细胞内甲基化水平上升与DNA损伤修复相关。另外, 利用5-mC DNA甲基化试剂盒重复上述研究得出的结果与MspⅠ/ HpaⅡ酶切法相同, 说明研究结果可靠。有研究推测, 在DNA修复期间, DNA甲基化水平的变化有利于稳定染色体抑制性标记如组蛋白修饰H3K9me2/3以及导致染色质的浓缩, 在DNA损伤修复期间抑制基因的表达, 起到稳定基因组的作用[25]。综合以上发现, 推测短期低温胁迫下, 由于活性氧的产生导致DNA损伤, DNA损伤激活了DNA损伤修复机制, 进一步导致了DNA甲基化水平上升, 从而可能对ZF4细胞基因组起到稳定作用。而这其中所涉及的ROS通路和ATM信号通路仍有待进一步研究。
综上所述, 本研究通过对斑马鱼细胞进行低温胁迫, 研究不同温度和时间点下斑马鱼细胞DNA甲基化水平的变化, 发现细胞在短期低温胁迫下DNA甲基化水平上升, 而细胞长期在低温下培养后DNA甲基化水平下降。并且短期低温胁迫下DNA甲基化水平上升与ROS产生以及DNA损伤修复有关, 为后期斑马鱼细胞低温胁迫分子机制的研究奠定基础。
| [1] |
Singer-Sam J, Riggs A D. DNA Methylation: Molecular Biology and Biological Significance[M]. New York: Springer- Verlag, 1993: 358-384.
|
| [2] |
Attwood J T, Yung R L, Richardson B C. DNA methylation and the regulation of gene transcription[J]. Cell and Molecular Life Science, 2002, 59(2): 241-257. DOI:10.1007/s00018-002-8420-z |
| [3] |
Lister R, Pelizzola M, Dowen R H, et al. Human DNA methylomes at base resolution show widespread epigenomic differences[J]. Nature, 2009, 462(7271): 315-322. DOI:10.1038/nature08514 |
| [4] |
Secco D, Wang C, Shou H X, et al. Stress induced gene expression drives transient DNA methylation changes at adjacent repetitive elements[J]. eLife, 2015, 4: e09343. DOI:10.7554/eLife.09343 |
| [5] |
Kisliouk T, Cramer T, Meiri N. Methyl CpG level at distal part of heat-shock protein promoter HSP70 exhibits epigenetic memory for heat stress by modulating recruitment of POU2F1- associated nucleosome-remodeling deacetylase (NuRD) com plex[J]. Journal of Neurochemistry, 2017, 141(3): 358-372. DOI:10.1111/jnc.2017.141.issue-3 |
| [6] |
Wang M, Qin L M, Xie C, et al. Induced and constitutive DNA methylation in a salinity-tolerant wheat introgression line[J]. Plant and Cell Physiology, 2014, 55(7): 1354-1365. DOI:10.1093/pcp/pcu059 |
| [7] |
Pecinka A, Dinh H Q, Baubec T, et al. Epigenetic regulation of repetitive elements is attenuated by prolonged heat stress in Arabidopsis[J]. Plant Cell, 2010, 22(9): 3118-3129. DOI:10.1105/tpc.110.078493 |
| [8] |
Dai T M, Lü Z C, Wang Y S, et al. Molecular characterizations of DNA methyltransferase 3 and its roles in temperature tolerance in the whitefly, Bemisia tabaci Mediterranean[J]. Insect Molecular Biology, 2018, 27(1): 123-132. DOI:10.1111/imb.12354 |
| [9] |
Hernández J A, Ferrer M A, Jiménez A, et al. Antioxidant systems and O2–·/H2O2 production in the apoplast of pea leaves. Its relation with salt-induced necrotic lesions in minor veins[J]. Plant Physiology, 2001, 127(3): 817-831. DOI:10.1104/pp.010188 |
| [10] |
Xu Q Q, Han B S, Luo J T, et al. Effects of cold stress on ROS production and expression of MAPK proteins in zebrafish ZF4 cells[J]. Journal of Fishery Sciences of China, 2016, 23(4): 771-776. [许琼琼, 韩兵社, 罗军涛, 等. 低温胁迫诱导斑马鱼ZF4细胞ROS及MAPK相关蛋白表达的影响[J]. 中国水产科学, 2016, 23(4): 771-776.] |
| [11] |
Liu H, Ouyang B, Zhang J H, et al. Differential modulation of photosynthesis, signaling, and transcriptional regulation between tolerant and sensitive tomato genotypes under cold stress[J]. PLoS ONE, 2012, 7(11): e50785. DOI:10.1371/journal.pone.0050785 |
| [12] |
Mortusewicz O, Schermelleh L, Walter J, et al. Recruitment of DNA methyltransferase Ⅰ to DNA repair sites[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(25): 8905-8909. DOI:10.1073/pnas.0501034102 |
| [13] |
Ding N, Bonham E M, Hannon B E, et al. Mismatch repair proteins recruit DNA methyltransferase 1 to sites of oxidative DNA damage[J]. Journal of Molecular Cell Biology, 2016, 8(3): 244-254. DOI:10.1093/jmcb/mjv050 |
| [14] |
Berger N D, Stanley F K T, Moore S, et al. ATM-dependent pathways of chromatin remodelling and oxidative DNA damage responses[J]. Philosophical Transactions of the Royal Society of London Series B-Biological Sciences, 2017, 372(1731): 20160283. DOI:10.1098/rstb.2016.0283 |
| [15] |
Guo Z, Kozlov S, Lavin M F, et al. ATM activation by oxidative stress[J]. Science, 2010, 330(6003): 517-521. DOI:10.1126/science.1192912 |
| [16] |
Howe K, Clark M D, Torroja C F, et al. The zebrafish reference genome sequence and its relationship to the human genome[J]. Nature, 2013, 496(7446): 498-503. DOI:10.1038/nature12111 |
| [17] |
Hu P, Liu M L, Zhang D, et al. Global identification of the genetic networks and cis-regulatory elements of the cold response in zebrafish[J]. Nucleic Acids Research, 2015, 43(19): 9198-9213. DOI:10.1093/nar/gkv780 |
| [18] |
Han B S, Li W H, Chen Z Z, et al. Variation of DNA methylome of zebrafish cells under cold pressure[J]. PLoS ONE, 2016, 11(8): e0160358. DOI:10.1371/journal.pone.0160358 |
| [19] |
Chwastek J, Jantas D, Lasoń W. The ATM kinase inhibitor KU-55933 provides neuroprotection against hydrogen peroxide-induced cell damage via a γH2AX/p-p53/caspase- 3-independent mechanism: Inhibition of calpain and cathepsin D[J]. The International Journal of Biochemistry & Cell Biology, 2017, 87: 38-53. |
| [20] |
Ugarte F, Sousae R, Cinquin B, et al. Progressive chromatin condensation and H3K9 methylation regulate the differentiation of embryonic and hematopoietic stem cells[J]. Stem Cell Reports, 2015, 5(5): 728-740. DOI:10.1016/j.stemcr.2015.09.009 |
| [21] |
Wang G F, Kang N, Gong H M, et al. Upregulation of uncoupling protein Ucp2 through acute cold exposure increases post-thaw sperm quality in zebrafish[J]. Cryobiology, 2015, 71(3): 464-471. DOI:10.1016/j.cryobiol.2015.08.016 |
| [22] |
Wu S M, Liu J H, Shu L H, et al. Anti-oxidative responses of zebrafish (Danio rerio) gill, liver and brain tissues upon acute cold shock[J]. Comparative Biochemistry and Physiology Part A: Molecular & Integrative Physiology, 2015, 187: 202-213. |
| [23] |
Chen S E, Yu M C, Chu X, et al. Cold-induced retrotransposition of fish LINEs[J]. Journal of Genetics and Genomics, 2017, 44(8): 385-394. DOI:10.1016/j.jgg.2017.07.002 |
| [24] |
Shamma A, Suzuki M, Hayashi N, et al. ATM mediates pRB function to control DNMT1 protein stability and DNA methylation[J]. Molecular and Cellular Biology, 2013, 33(16): 3113-3124. DOI:10.1128/MCB.01597-12 |
| [25] |
Russo G, Landi R, Pezone A, et al. DNA damage and Repair Modify DNA methylation and Chromatin Domain of the Targeted Locus: Mechanism of allele methylation polymorphism[J]. Scientific Reports, 2016, 6: 33222. DOI:10.1038/srep33222 |
 2019, Vol. 26
2019, Vol. 26

