2. 中国科学院海洋研究所, 山东 青岛 266003
2. Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266003, China
海参纲(Holothuroidea)隶属于棘皮动物门(Echinodermata), 在海洋中分布广泛, 全世界约有1400种[1], 中国共有15科58属147种[2], 以印度-西太平洋区系种类为主。中国有21种可供食用[3], 除仿刺参(Apostichopus japonicus)分布于北方黄渤海域, 其余20种可食用海参均分布于中国东海与南海。海参中含有丰富的氨基酸、胶原蛋白以及多种人体所需的微量元素, 具有多种生理药理活性[4-5], 有很高的经济价值和开发潜力。
海参骨片是埋藏于海参真皮表层的内骨骼, 是海参的重要分类特征。廖玉麟[6]记录描绘了中国134种海参的外形及骨片。赵世民[7]描述了台湾沿岸潮间带30种海参及骨片的超显微结构。李赟等[8]的研究结果表明仅通过比较海参背部真皮层的骨片来鉴定海参是可行的。
随着研究的不断深入, 人们对海参纲分类系统的认识也在转变。基于形态学特征分为6个目:平足目(Elasipodida)、楯手目(Aspidochirotida)、枝手目(Dendrochirotida)、指手目(Dactylochirotida)、芋参目(Molpadida)、无足目(Apodida)。近年来随着分子生物学的发展, 从分子水平研究物种分类及系统发育成为必要且有效的手段。Miller等[9]的研究结果表明传统分类系统中的楯手目并非单系, 提出了新的分类系统, 将海参纲重新划分为7个目:枝手目、辛那参目(Synallactida)、芋参目、Persiculida(尚无中文名)、海参目(Holothuriida)、平足目、无足目, 停止使用楯手目和指手目。
线粒体DNA由于分子量小, 且广泛地分布于细胞中, 拷贝数量极大, 非常易于分离纯化, 可作为物种系统发育分析的有效分子标记。基于16S rRNA基因片段, 能有效区分海参的种类[10], 并对海参的亲缘关系和骨片演化进行分析[11-13]。本研究基于海参线粒体16S rDNA基因, 分析不同海参间的亲缘关系, 对基于形态学特征和分子水平的两套分类系统进行比较; 并应用扫描电子显微镜对其骨片进行观察, 探讨海参科内骨片的演化过程。
1 材料与方法 1.1 实验材料选用13种海参, 均为完整的海参个体, 用无水乙醇固定后带回实验室进行形态鉴定, 根据形态特征判定这13种海参分别属于海参科、刺参科和硬瓜参科, 基本信息见表 1。
|
|
表 1 13种海参的分类及样品信息 Tab.1 Basic information of 13 species of sea cucumbers in this study |
骨片制备的通用方法:从海参体壁切取1 cm2×1 mm的体壁组织, 每个样品重复取样3次, 用蒸馏水洗净, 吸水纸吸干水分, 置于1.5 mL离心管中, 滴加1 mL 10% NaClO溶液消化60 s左右, 挑出未消化的组织块, 待骨片沉淀完全, 吸去上层溶液, 滴加蒸馏水重复漂洗5次。充分漂洗后, 待骨片沉淀完全, 吸去上层溶液, 滴加无水乙醇漂洗2次。
吸取骨片悬液, 小心滴加在胶膜上, 烘干至酒精完全挥发, 喷金后通过扫描电子显微镜进行观测拍照。
1.3 DNA的提取、扩增、测序在无菌条件下, 切取海参样本体壁组织30 mg左右, 用超纯水洗净, 无菌纸吸干水分, 使用组织DNA提取试剂盒提取样品DNA, 试剂盒品牌为OMEGA, 所得DNA -20℃保存。
以基因组DNA模板, 参照无脊椎动物16S rDNA基因片段的通用引物[12]16Sar: 5'-CGCCTG TTTAACAAAAACAT-3', 16Sbr: 5'-CCGGTCTGA ACTCAGATCATG-3'进行PCR扩增, 引物经北京擎科新业生物技术有限公司合成。
PCR反应的总体积为20 μL, 含ddH2O 8 μL, Mix 10 μL, 引物各0.8 μL, 模板DNA 0.4 μL, 所用Mix为北京擎科新业生物技术有限公司青岛分公司提供,浓度为2×。PCR反应程序如下: 94℃预热5 min; 94℃变性30 s, 52℃退火45 s, 72℃延伸2 min, 经42个循环; 72℃延伸10 min。反应结束后, 制取1.5%琼脂糖凝胶, 用GelRed进行染色, 取3 μL产物进行凝胶电泳检测, 基于凝胶成像来检测PCR产物, 根据情况对PCR产物纯化。产物送至北京擎科新业生物技术有限公司进行双向测序。
1.4 数据处理双向测序后用DNASTAR和Clustal X软件进行拼接和比对, 应用DNAsp5软件计算单态位点、多态位点和插入缺失位点等, MEGA 5.0软件分析碱基组成。为分析实验所用的13种海参与其他海参之间的同源性和系统发生关系, 从GenBank上选取了海参科、刺参科、瓜参科、沙鸡子科、尻参科的11种海参的16S rDNA序列作为参照(表 2), 比较遗传距离, 并以杂色角孔海胆作外群, 采用邻接法(NJ)和最小进化法(ME)构建系统树, 通过MEGA 5.0、PAUP 4.0软件处理, 对所得系统树进行自展法检验(1000次重复)。
|
|
表 2 来自GenBank中的11种海参的基本信息 Tab.2 List of 11 species of sea cucumbers from GenBank |
13种海参体壁内骨片的主要样式包括桌形体(图 1 a-j)、假桌形体(图 1 k)、扣状体(图 2)、杆状体(图 3 a-c)、穿孔板(图 3 d-e)、花纹样体(见图 4 a-c)和颗粒体(图 4 d), 各海参骨片类型具体见表 3。

|
图 1 10种海参桌形体(a-j)及格皮氏海参假桌形体(k)扫描电镜图 a:明柄体参; b:红腹海参; c:棕环海参; d:玉足海参; e:虎纹海参; f:黑赤星海参; g:沙海参; h:黄疣海参; i1-i2:仿刺参; j:丛足硬瓜参; k:格皮氏海参. Fig.1 Type tables (a-j) of 10 sea cucumbers and pseudo table (k) of Pearsonothuria graeffei (SEM photographs) a: Labidodemas pertinax; b: Holothuria edulis; c: Holothuria fuscocinerea; d: Holothuria leucospilota; e: Holothuria pervicax; f: Holothuria cinerascens; g: Holothuria arenicola; h: Holothuria hilla; i1-i2: Apostichhopus japonicus; j: Sclerodactyla multipes; k: Pearsonothuria graeffei. |
|
|
表 3 13种海参骨片类型 Tab.3 Ossicles composition of 13 species of sea cucumber |
对13种海参骨片的观察发现, 同属的海参骨片有一定共性, 如海参属的7种海参均具有桌形体, 其中红腹海参(Holothuria edulis)、棕环海参(Holothuria fuscocinerea)、玉足海参(Holothuria leucospilota)、沙海参(Holothuria arenicola)、黄疣海参(Holothuria hilla)均具有扣状体, 而虎纹海参(Holothuria pervicax)和黑赤星海参(Holothuria cinerascens)则均具有杆状体。同时, 不同种海参的同类型骨片亦有所差异, 棕环海参、虎纹海参、仿刺参成年个体的桌形体塔部退化, 仅残留基部, 而其余具桌形体的海参桌形体塔部均存在; 棕环海参扣状体穿孔数较少, 红腹海参扣状体边缘有突起。不同属的海参之间骨片有较大差异, 如格皮氏海参(Pearsonothuria graeffei)所特有的假桌形体。以上结果进一步证明了骨片可作为海参种类鉴定的有效鉴别特征。
观察中还发现, 仿刺参幼体中桌形体塔部较高(图 1i2), 而成年个体的桌形体塔部则退化变低或消失(图 1i1);棕环海参、虎纹海参桌形体塔部退化, 多数只残留基部(图 1c和图 1e); 棕环海参扣状体简单, 穿孔数少(图 2b); 虎纹海参杆状体两侧有分支(图 3a)。子安辐肛参和蛇目白尼参花纹样体较为简单(图 4a, 图 4b), 而格皮氏海参花纹样体较为复杂(图 4c), 蛇目白尼参颗粒体为具有2~3个小穿孔的饼干型颗粒体(图 4d)。

|
图 2 5种海参扣状体扫描电镜图 a:红腹海参; b:棕环海参; c:玉足海参; d:沙海参; e:黄疣海参. Fig.2 Type buttons of 5 sea cucumbers (SEM photographs) a: Holothuria edulis; b: Holothuria fuscocinerea; c: Holothuria leucospilota; d: Holothuria arenicola; e: Holothuria hilla. |

|
图 3 3种海参杆状体(a-c)和3种海参穿孔板(d-f)扫描电镜图 a:虎纹海参; b:黑赤星海参; c:仿刺参; d:玉足海参; e:黑赤星海参; f:仿刺参. Fig.3 Rods (a-c) of 3 sea cucumbers and plates (d-f) of 3 sea cucumbers (SEM photographs) a: Holothuria pervicax; b: Holothuria cinerascens; c: Apostichhopus japonicus; d: Holothuria leucospilota; e: Holothuria cinerascens; f: Apostichhopus japonicus. |

|
图 4 3种海参花纹样体(a-c)和蛇目白尼参颗粒体(d)扫描电镜图 a:子安辐肛参; b:蛇目白尼参; c:格皮氏海参; d:蛇目白尼参. Fig.4 4 Type rosettes (a-c)of 3 sea cucumbers and oblate grain (d) of Bohadschia argus (SEM photographs) a: Actinopyga lecanora; b: Bohadschia argus; c: Pearsonothuria graeffei; d: Bohadschia argus. |
由于部分样品保存欠佳, 仅从8种海参中扩增出16S rDNA片段, 大小在465 bp左右, 其中玉足海参测定了3个样品, 存在2个变异位点, 定义了3个单倍型, 下文缩写为H.leu1、H.leu2和H.leu3;沙海参测定了2个样品, 存在3个变异位点, 定义了2个单倍型, 下文缩写为H.are1和H.are2;黄疣海参和丛足硬瓜参(Sclerodactyla multipes)测定了2个样品, 均只有一个单倍型; 其他4种海参测定了1个样品。序列递交到GenBank数据库, 序列号见表 4。8种海参16S rDNA部分序列的比对结果表明, 在比对的446个位点中, 单态位点253个, 多态位点163个, 其中简约信息位点105个, 插入缺失位点30个。
|
|
表 4 8种海参16S rDNA基因GenBank序列号、片段长度和碱基组成 Tab.4 Accession no., lengths and base contents of 16S rDNA gene fragments |
碱基组成分析(表 4)表明, 8种海参序列中, T碱基和A碱基含量均较高, A+T含量均高于C+G含量, 平均值为55.8%, 符合无脊椎动物线粒体DNA序列的特征。其中, 硬瓜参科的丛足硬瓜参A+T含量为62.3%, 远高于海参科的7种海参。
2.3 遗传距离为分析实验所用海参与其他海参之间的同源性和系统发生关系, 从GenBank上选取了海参科、刺参科、瓜参科、沙鸡子科、尻参科的11种海参的16S rDNA序列作为参照(表 2), 与本实验获得的8种海参的16S rDNA共同进行分析, 得到各序列间的遗传距离(表 5)。
|
|
表 5 基于16SrDNA序列片段的19种海参种间遗传距离 Tab.5 Genetic distances of 19 sea cucumber species based on sections of 16S rDNA |
遗传距离分析结果表明, 同一种海参之间的遗传距离最近, 为0.000~0.005, 这一结果说明, 16S rDNA的遗传稳定性相当高, 可有效区分不同种海参。不同种海参间遗传距离差别很大, 遗传距离在0.075~0.346;海参属内遗传距离为0.075~0.179, 海参科内不同属之间遗传距离为0.121~0.238;海参科内遗传距离为0.075~0.238, 海参科和刺参科之间的遗传距离为0.226~0.312。
2.4 分子系统发育树根据从GenBank上获取的11种海参的16S rDNA序列, 与本实验获得的8种海参的16S rDNA序列片段, 以杂色角孔海胆为外群, 建立NJ (图 5)、ME (图 6)系统发育树。NJ、ME系统发育树几乎完全一致, 沙海参和玉足海参各自不同的单倍型首先聚成一簇, 然后再与海参属的其余5种海参和柄体参属的1种海参聚成一簇。在科的水平上, 海参科的11种海参聚成一簇, 刺参科先与尻参科聚成一簇, 再与枝手目的沙鸡子科、硬瓜参科、瓜参科聚成一簇。
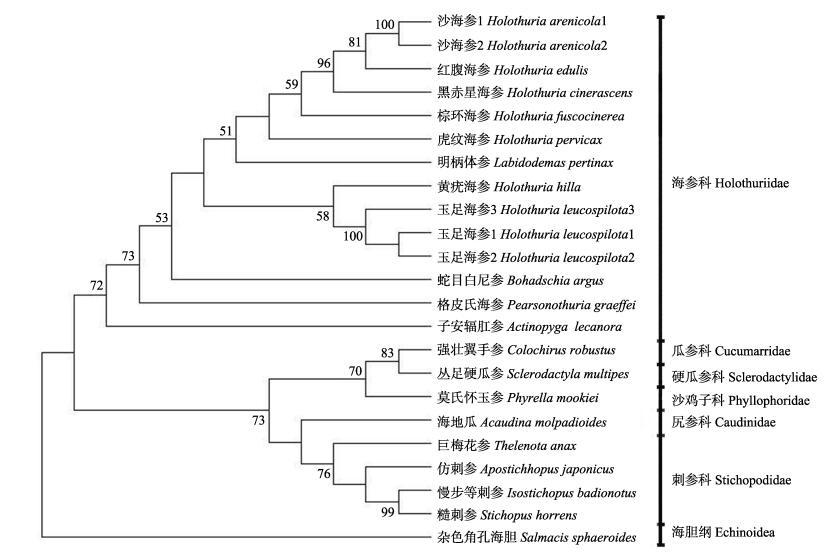
|
图 5 基于16S rDNA构建的19种海参Neighbor-Joining系统发育树 节点上的数字为1000次重复大于50%的自展值. Fig.5 Neighbor-Joining phylogenetic tree of 19 sea cucumbers reconstructed from 16S rDNA Numbers on the node represent percentage of 1000 bootstrap replications above 50%. |
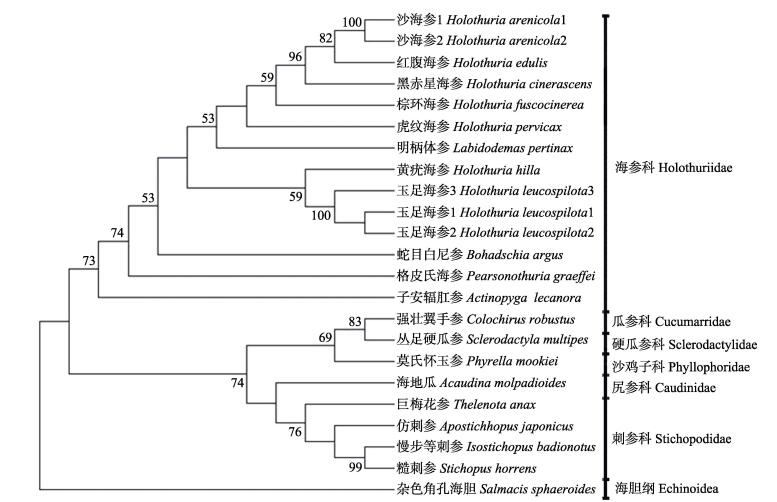
|
图 6 基于16S rDNA构建的19种海参Minimum-Evolution系统发育树 节点上的数字为1000次重复大于50%的自展值. Fig.6 Minimum-Evolution phylogenetic tree of 19 sea cucumbers reconstructed from 16S rDNA Numbers on the node represent percentage of 1000 bootstrap replications above 50%. |
本研究扩增了8种海参的16S rDNA的部分序列, 结果表明, 本研究扩增的线粒体16S rDNA序列在不同科属间具有明显的差异, 是研究科属间亲缘关系的适宜序列。
8种海参序列中, T碱基和A碱基含量均较高, A+T含量均高于C+G含量, 平均值为55.8%, 符合无脊椎动物线粒体DNA序列的特征。同种内个体序列完全相同或仅具极少变异位点, 远低于不同种之间的差异, 可作为区别不同种的有效手段。
从遗传距离看, 存在海参属内遗传距离大于海参科内不同属之间遗传距离, 海参科内遗传距离大于海参科和刺参科之间遗传距离的现象。海参属数目庞大, 约有150种, 而海参科又是海参纲内第二大科, 约有185种[1]。因此, 如此庞大的类群中不同种之间存在较大的差异属于正常现象。
3.2 基于16S rDNA基因片段的海参分子系统发育由16S rDNA基因片段构建的系统发育树可以看出, 同种海参的不同单倍型以极高的自展值聚在一起, 体现了良好的种内保守性。传统形态学分类中, 将玉足海参、虎纹海参和棕环海参同归为麦太参亚属(Mertensiothuria), 本实验结果却表明, 玉足海参与虎纹海参和棕环海参的亲缘关系较远, 反而与传统形态学分类中全疣参亚属(Thymiosycia)的黄疣海参亲缘关系较近。严俊贤[14]通过16S rDNA和18S rDNA分析了南海6种海参的系统发育, 亦发现玉足海参与棕环海参亲缘关系较远, 分子的分类结果与传统形态学亚属划分的不一致体现了形态学分类与分子系统分类的差异。
明柄体参(Labidodemas pertinax)与海参属的海参聚为一簇; 明柄体参与玉足海参、黄疣海参都是蠕虫状的海参, 与其他3个属的形态差别较大[9];同时, 明柄体参的骨片为桌形体和杆状体, 与海参属差别不大, 而其他3个属的海参以花纹样体为主, 骨片类型差距很大。因此, 海参科内柄体参属与海参属亲缘关系极近且骨片形态均相似, 与其他三个属明显不同, 可划分为同一个属。
刺参科先与尻参科聚成一簇, 再与枝手目的沙鸡子科、硬瓜参科、瓜参科聚成一簇, 相较刺参科, 海参科与尻参科和枝手目的沙鸡子科、硬瓜参科、瓜参科亲缘关系较近。Kerr等[15]通过形态特征对海参进行了研究, 其中, 刺参科和海参科仅在是否具有发达的背疣足方面有显著差异, 说明海参科和刺参科亲缘关系较近; 费来华等[12]通过分析10种海参16S rDNA序列多样性及亲缘关系认为, 属于枝手目的瓜参科与刺参科的亲缘关系较近; 陈丽梅等[16]通过研究4种海参16S rDNA表明, 刺参科先与枝手目的沙鸡子科、瓜参科聚类, 再与海参科聚类; Miller等[9]提出了新的分类系统, 认为刺参科属于辛那参目, 与芋参目、Persiculida(尚无中文名)和枝手目亲缘关系较近, 而与海参目海参科亲缘关系较远。海参科只具有1束生殖腺, 而刺参科、芋参目、枝手目的种类均具有2束生殖腺[6]。传统形态学分类中刺参科和海参科均属于楯手目, 而分子生物学研究表明刺参科和海参科遗传距离较远, 分属不同的目。传统分类学根据形态将具有楯形触手的海参科和刺参科归为一目, 与此相比较, 结合分子系统发育的新型分类系统更能反映海参的分类地位和亲缘关系。
3.3 基于16S rDNA基因片段的海参骨片演化分析本研究基于16S rDNA基因, 对海参科内骨片特征进行对比(图 7), 发现不同聚类间骨片类型逐渐不同。海参属与柄体参属聚为一簇, 均具有桌形体, 白尼参属(Bohadschia)、辐肛参属(Actinopyga)、皮氏海参属(Pearsonothuria)分支出去, 无桌形体, 出现花纹样体。扣状体和杆状体只存在于海参属和柄体参属中。其中棕环海参扣状体简单, 穿孔数少, 这或许是扣状体和颗粒体之间的过渡阶段(图 2 b)。海参属和柄体参属再与格皮氏海参和蛇目白尼参(Bohadschia argus)聚为一簇, 最后与子安辐肛参(Actinopyga lecanora)聚为一簇。其中, 格皮氏海参具有假桌形体(图 1 k), 与桌形体比较没有明显的底盘, 蛇目白尼参具有较扣状体更为简单的颗粒体(图 4 d),子安辐肛参仅具花纹样体。
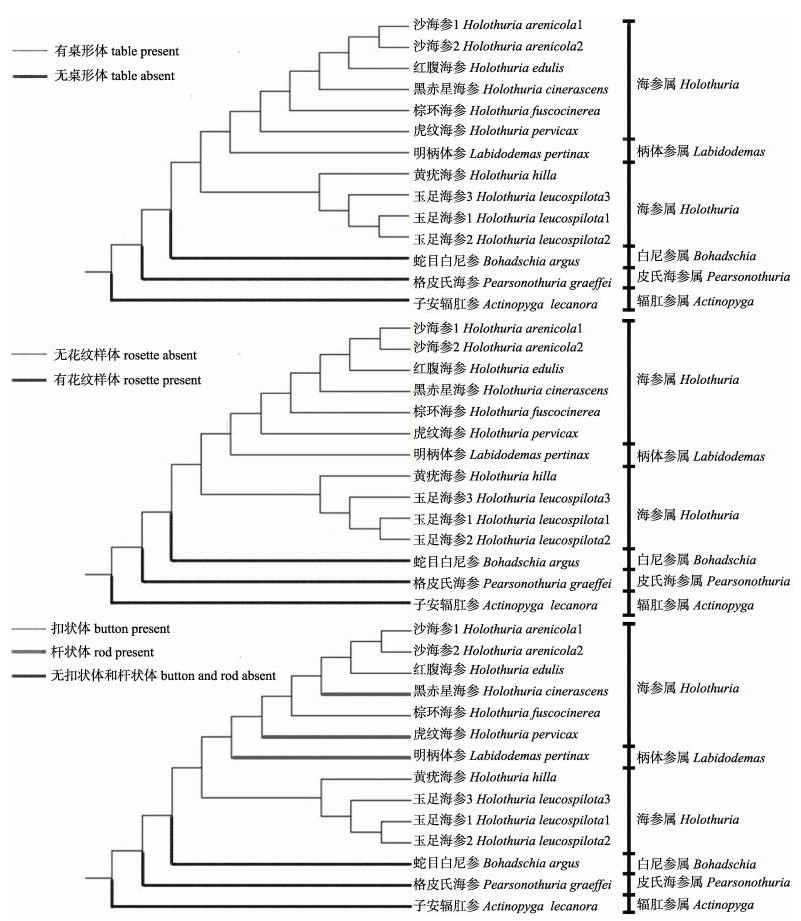
|
图 7 基于系统发育的海参科骨片特征对比 Fig.7 Ossicle contrast of family Holothuriidae based on phylogenetic |
观察发现, 仿刺参小个体的桌形体塔部较高, 而成年个体的桌形体塔部则退化变低或消失。廖玉麟[6]发现沙海参大个体较小个体桌形体塔部变低, 横梁变少, 扣状体穿孔数变少。Massin等[17]通过电镜研究了糙海参的骨片发育过程, 发现随着海参幼体的长大, 扣状体变短, 穿孔数变少。说明海参科内海参随着个体的长大, 骨片逐渐退化并变得简单, 经历了一个由繁到简的过程。因此, 尽管桌形体的演化仍存在争议, 它可能是起源于除平足目和无足目以外的其他海参, 并经历了多次独立的丢失和变化, 也有可能是在海参纲内经历了数次演化[9]。由此可以推测, 仅在海参科内, 海参骨片是由繁到简,从复杂的桌形体、扣状体, 向简单的花纹样体和颗粒体演化的。同时, 结合虎纹海参的杆状体和棕环海参的扣状体可以看出, 海参科内骨片的进化不是直接的, 而是逐渐地慢慢过渡。Kerr等[11]提出海参科种类至少经历了两轮从桌形体和扣状体到花纹样体的演化过程, 与本研究得出的结论一致。关于海参科内骨片的进化关系还有待进一步研究, 随着新技术的应用及研究中所涉及的种类数目的增多, 对海参骨片演化会有更清晰的认识。
| [1] |
Smiley S, Harrison F W, Chia F S. Microscopic Anatomy of Invertebrates:Echinodermata[M]. New York: Wiley-Liss Press, 1994: 401-471.
|
| [2] |
Liao Y L, Xiao N. Species composition and faunal characteristics of echinoderms in China seas[J]. Biodiversity Science, 2011, 19(6): 729-736. [廖玉麟, 肖宁. 中国海棘皮动物的种类组成及区系特点[J]. 生物多样性, 2011, 19(6): 729-736.] |
| [3] |
Cui G Y, Zhao L. Names and species identifying of edible sea cucumber in China[J]. Cuisine Journal of Yangzhou University, 2000, 17(3): 13-18. [崔桂友, 赵廉. 食用海参的名称与种类鉴别[J]. 扬州大学烹饪学报, 2000, 17(3): 13-18. DOI:10.3969/j.issn.1009-4717.2000.03.005] |
| [4] |
Wang L, Lu H X, Chen Q. Analysis and evaluation of the nutritional components of sea cucumber (Acaudina molpadioidea)[J]. Food and Fermentation Industries, 2014, 40(8): 215-218. [王磊, 陆海霞, 陈青. 东海海参(Acaudina molpadioidea)营养成分分析及评价[J]. 食品与发酵工业, 2014, 40(8): 215-218.] |
| [5] |
Guo Y Y, Ding Y, Xu F F, et al. Progress in research on main bioactive constituents of sea cucumber[J]. Food Science, 2014, 35(15): 335-344. [郭盈莹, 丁燕, 徐飞飞, 等. 海参中主要生物活性成分研究进展[J]. 食品科学, 2014, 35(15): 335-344. DOI:10.7506/spkx1002-6630-201415066] |
| [6] |
Liao Y L. Fauna Sinica:Phylum Echinodermata, Class Holothuroidea[M]. Beijing: Science Press, 1997. [廖玉麟. 中国动物志:棘皮动物门, 海参纲[M]. 北京: 科学出版社, 1997.]
|
| [7] |
Zhao S M. Shallow-Water Sea Cucumber of Taiwan[M]. Taichung: Museum of Natural Science, 1998. [赵世民. 台湾礁岩海岸的海参[M]. 台中: 自然科学博物馆, 1998.]
|
| [8] |
Li Y, Fei L H, Chen J X. The morphology of ossicles of 15 commercial holothurians[J]. Periodical of Ocean University of China, 2008, 38(2): 211-216. [李赟, 费来华, 陈家鑫. 十五种海参骨片的形态学研究[J]. 中国海洋大学学报(自然科学版), 2008, 38(2): 211-216.] |
| [9] |
Miller A K, Kerr A M, Paulay G, et al. Molecular phylogeny of extant Holothuroidea (Echinodermata)[J]. Molecular Phylogenetics and Evolution, 2017, 111: 110-131. |
| [10] |
Wen J, Hu C Q, Zhang L P, et al. PCR-RFLP identification of 16 commercial sea cucumber species on the basis of 16S rRNA gene[J]. Journal of Fishery Sciences of China, 2011, 18(2): 451-457. [文菁, 胡超群, 张吕平, 等. 16种商品海参16S rRNA的PCR-RFLP鉴定方法[J]. 中国水产科学, 2011, 18(2): 451-457.] |
| [11] |
Kerr A M, Janies D A, Clouse R M, et al. Molecular phylogeny of coral-reef sea cucumbers (Holothuriidae:Aspidochirotida) based on 16S mitochondrial ribosomal DNA sequence[J]. Marine Biotechnology, 2005, 7(1): 53-60. DOI:10.1007/s10126-004-0019-y |
| [12] |
Fei L H, Li Y, Chen J X. 16S rDNA sequence diversity and phylogenetic relationship of ten sea cucumbers[J]. Journal of Fishery Sciences of China, 2008, 15(5): 755-765. [费来华, 李赟, 陈家鑫. 10种海参16S rDNA序列多样性及其亲缘关系分析[J]. 中国水产科学, 2008, 15(5): 755-765. DOI:10.3321/j.issn:1005-8737.2008.05.006] |
| [13] |
Wen J, Hu C Q, Fan S G. Molecular phylogeny of 15 sea cucumbers and the inferences about the evolution of ossicles[J]. Marine Sciences, 2011, 35(5): 66-72. [文菁, 胡超群, 范嗣刚. 中国15种海参的分子系统发育和骨片演化的分析[J]. 海洋科学, 2011, 35(5): 66-72.] |
| [14] |
Yan J X. The phylogeny of six species sea cucumbers in South China Sea and studies on embryonic and larval development of sea cucumber Holothria scabra and Stichopus variegates[D]. Haikou: Hainan University, 2012. [严俊贤.六种南海海参的系统发育与糙海参和花刺参的胚胎与幼体发育研究[D].海口: 海南大学, 2012.]
|
| [15] |
Kerr A M, Kim J. Phylogeny of Holothuroidea (Echinodermata) inferred from morphology[J]. Zoological Journal of the Linnean Society, 2001, 133(1): 63-81. DOI:10.1111/j.1096-3642.2001.tb00623.x |
| [16] |
Chen L M, Li Q, Li Y. Sequence analysis of mitochondrial 16S rRNA and COI gene and molecular phylogeny of four species of sea cucumber[J]. Journal of Fishery Sciences of China, 2008, 15(6): 935-942. [陈丽梅, 李琪, 李赟. 4种海参16S rRNA和COI基因片段序列比较及系统学研究[J]. 中国水产科学, 2008, 15(6): 935-942. DOI:10.3321/j.issn:1005-8737.2008.06.006] |
| [17] |
Massin C, Mercier A, Hamel J F. Ossicle change in Holothuria scabra with a discussion of ossicle evolution within the Holothuriidae (Echinodermata)[J]. Acta Zoologica (Stockholm), 2000, 81(1): 77-91. |
 2019, Vol. 26
2019, Vol. 26

