2. 福建农林大学动物科学学院, 福建 福州 350002
2. College of Animal Sciences, Fujian Agriculture and Forestry University, Fuzhou 350002, China
拟穴青蟹(Scylla paramamosain)是中国南方沿海地区广泛养殖的最重要的海洋蟹类, 同时也是印度洋和太平洋地区许多国家重要的近岸渔业资源[1]。由于拟穴青蟹生长周期短、个体大、肉质肥美、营养价值高, 因此具有很高的经济价值。在中国, 拟穴青蟹已有100多年的养殖历史[2]。然而, 到目前为止, 拟穴青蟹养殖在很大程度上仍来源于从海里收集的幼蟹, 对自然种群造成了相当大的压力[1]。因此培育优良亲本, 建立规模化人工育苗, 引导养殖业者采用人工苗种进行养殖, 将对减少甚至完全避免对野生苗种的采捕, 保护青蟹自然资源具有重要意义[3]。
胚胎发育是甲壳动物个体发育过程中的重要环节, 其重要性不仅体现在胚胎是幼体变态发育的起点, 更主要的是胚胎发育的好坏是幼体体质的决定性因素之一。目前国内对拟穴青蟹胚胎发育的研究主要集中在形态观察和生理生化指标的测量, 如丁宏印等[4]、李少菁等[5]、Zeng[6]对拟穴青蟹胚胎发育过程中的主要生化组成、几种水解酶活力、不同的理化条件(温度、盐度、pH)对其影响等进行了研究, 而对于胚胎发育过程中相关基因的研究较少。因此, 建立稳定可靠的实时定量聚合酶链式反应(qRT-PCR)实验体系, 对于进一步开展拟穴青蟹参与胚胎发育的功能基因的研究尤为重要。
目前qRT-PCR已被广泛用于分析信使RNA(mRNA)的表达模式, 并比较不同生物样本中mRNA的相对水平, qRT-PCR技术的应用极大地促进了我们对各种生物基因表达的理解, 并成为准确测定mRNA表达水平的首选方法[7-8]。目前, 针对甲壳动物不同组织、不同发育阶段和不同实验处理的内参基因筛选已开展了大量研究, 其中包括中华绒螯蟹(Eriocheir sinensis)[9]、克氏原螯虾(Procambarus clarkii)[10]、斑节对虾(Peneaus monodon)[11]、鲑鱼虱(Lepeophtheirus salmonis)[12]和南美蓝对虾(P. stylirostris)[13]等。虽然以上实验对象都是甲壳纲的动物, 但内参基因筛选的结果不尽相同。此外, 此外, 即使对于同一物种如克氏原螯虾, 不同组织或不同卵巢发育阶段中, 最适内参基因也不相同[10]。因此, 对于不同物种、不同组织、不同发育时间和不同实验处理, 筛选适合研究对象的qRT-PCR内参基因至关重要。
本实验首次基于本实验室拟穴青蟹胚胎转录组中获得的内参基因及前人报道的常用内参基因, 选用geNorm、NormFinder和BestKeeper[14]3个内参基因评估软件, 分别对拟穴青蟹9个不同发育时期的胚胎及1个成熟卵巢组织进行内参基因表达稳定性的分析, 旨在为拟穴青蟹不同胚胎发育时期基因表达的研究提供最佳的内参基因。
1 材料与方法 1.1 实验对象本实验不同发育时期的胚胎取自抱卵拟穴青蟹, 所有雌蟹均为漳州诏安的野生群体。收取性腺饱满、已受精的雌蟹, 暂养于集美大学水产养殖基地, 每只蟹单独饲养在200 L的养殖缸中, 饲养期间投喂菲律宾蛤仔。暂养期间将温度逐渐上升到26℃左右, 保持水质清洁, 每天干露1 h左右, 直到青蟹抱卵。取样时小心地将抱卵蟹从背部抓起, 然后用干净的毛刷轻轻地把腹部的胚胎剥下。每次收集完胚胎后, 将青蟹放回原来的养殖缸, 继续暂养, 等待下次收集。不同发育阶段的胚胎根据下列时间点收集: 36 hpf (hours post fertilization) (囊胚期)、66 hpf (原肠期)、102 hpf (无节幼体期)、132 hpf (5对附肢期)、156 hpf (7对附肢期)、186 hpf (复眼色素期Ⅰ)、222 hpf (复眼色素期Ⅱ)、270 hpf (复眼色素期Ⅲ)、312 hpf (近孵化期)[15]。抱卵蟹样品数量n=3;同时还选取3只成熟雌性青蟹的卵巢作为对照。取样后用超纯水洗涤后立即置于液氮中, 后转移至–80℃超低温冰箱保存, 用于总RNA提取。
1.2 总RNA提取拟穴青蟹不同发育时期胚胎的RNA均使用TRIzol®Reagent(Vazyme, 中国)提取。使用NanoDrop®1000核酸分析仪(Spectrophotometer, 美国)检测RNA的浓度及纯度, 并通过1%的琼脂糖凝胶电泳检测其完整性, 检测合格后立即进行逆转录。
1.3 cDNA合成分别取1 μg各发育时期胚胎的总RNA, 去除基因组DNA后, 使用RevertAid First Strand cDNA Synthesis Kit(Thermo Scientific, 中国)进行逆转录。逆转录产物稀释100倍作为qRT-PCR模板, 冻存于-20℃冰箱。
1.4 引物设计根据本实验室拟穴青蟹胚胎转录组数据, 获得常见内参基因Rps6、Ak、28S、18S、β-actin和EF-1α的编码序列, 后采用Primer 3.0(http://bioinfo.ebc.ee/mprimer3/)对上述序列进行qRT-PCR引物设计。Tuba1a、Rpl13、UBQ和gapdh的引物序列来源于已经发表的拟穴青蟹不同组织内参筛选的文章[16](表 1)。
|
|
表 1 荧光定量PCR引物序列及扩增效率 Tab.1 Sequence and efficiency of the primers used in qRT-PCR |
本实验使用LightCycler 480(Roche, 美国)实时定量PCR仪完成。采用20 μL反应体系: SYBR Green Realtime PCR Master Mix 10 μL, 正反向引物各0.5 μL (10 μmoL/L), cDNA模板9 μL。反应条件为: 95℃变性5 min; 然后95℃变性10 s, 60℃退火10 s, 72℃延伸10 s, 循环45次。待扩增曲线完成后, 让定量PCR仪逐渐降温并持续读取荧光值, 进行熔解曲线分析, 以确保每对定量引物扩增得到的产物均为单一产物。本实验每个发育阶段有3个生物学重复, 每个样品做2次技术重复。
1.6 数据处理和分析Excel整理qRT-PCR实验得到的CT值, 利用geNorm、NormFinder和Bestkeeper[14]软件分析候选内参基因的稳定性, 并筛选出1~2个拟穴青蟹胚胎发育过程中相对表达稳定的内参基因。
2 结果与分析 2.1 基因表达水平及引物扩增效率10个候选内参基因熔解曲线均为单峰, 标准曲线相关系数R2大于0.98, 扩增效率范围在90%~110% (表 1), 说明引物扩增效率高, 无非特异性扩增, 引物特异性良好, 可用于qRT-PCR实验[17-18]。
2.2 内参基因Ct值分析采用平均Ct值和其标准差描述基因的表达水平。10个候选内参基因中, 28S、18S和EF-1α的Ct值较小, 相对表达量高; UBQ的Ct值较大, 相对表达量低。UBQ和28S的Ct值变化范围较大, EF-1α的Ct值变化范围相对较小(图 1)。所有候选基因中, EF-1α的Ct值差异最小, 各样本间标准差为0.730; β-actin的Ct值差异最大, 且远大于其他候选基因, 其标准差为4.363;除β-actin外, Ct值差异最大的为UBQ, 其标准差为1.931。因β-actin是以上候选基因中表达最不稳定的, 故后续软件分析中未将β-actin纳入, 仅分析9个候选内参基因。
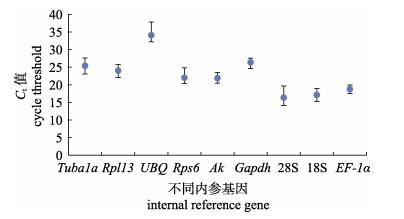
|
图 1 拟穴青蟹胚胎不同发育时期各候选内参基因的表达水平 Fig.1 The expression levels of the internal candidate genes in the different embryonic developmental stages of Scylla paramamosain |
本研究采用geNorm、NormFinder和Bestkeeper 3个软件对9个候选内参基因表达稳定性进行评价。
2.3.1 geNorm分析结果geNorm程序是2002年Vandesompele等[18]编写的用于qRT-PCR中筛选最适内参基因及确定最适内参基因数目的程序。软件使用前需将qRT-PCR得到的Ct值进行处理, 转变为相对定量数据。打开geNorm程序(使用Microsoft Office 2003), 导入处理好的数据, 程序会自动计算出各基因的M值, M值越小表示基因表达越稳定。geNorm程序还引入一个新的内参基因标准化因子配对变异V值, 并根据Vn/Vn+1值来确定最适内参基因的数目[19]。一般情况下默认V值为0.15 (该值可稍作调整), 如果存在Vn/Vn+1值< 0.15, 则最适内参基因的数量是n个。geNorm程序分析结果显示, 本研究中拟穴青蟹不同时期胚胎9个候选内参基因除UBQ和28S外, 其余7个内参基因M值均小于1.5, M值排序为UBQ > 28S > 18S > gapdh > Ak > Tuba1a > Rps6 > Rpl13=EF-1α (图 2)。此外, geNorm对不同发育时期9个候选内参基因的评估结果显示, 选取不同个数的候选内参基因V值差别不大, 因此需要2个内参基因才能够获得理想的目标基因定量结果(图 3)。
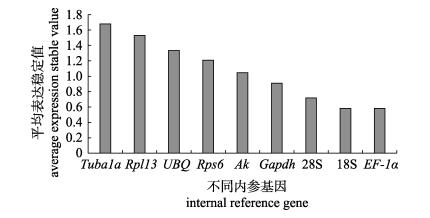
|
图 2 geNorm软件对拟穴青蟹胚胎不同发育时期内参基因表达稳定性排名 Fig.2 Ranking of the candidate internal reference genes calculated by geNorm in different embryonic developmental stages of Scylla paramamosain |
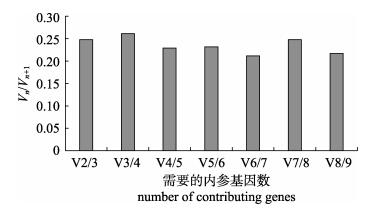
|
图 3 拟穴青蟹胚胎不同发育时期geNorm分析候选内参基因最适数目 Fig.3 Optimal number of the candidate internal reference genes calculated by geNorm in different development stages of Scylla paramamosain |
NormFinder程序是2004年Andersen等[20]编写的用于筛选qRT- PCR稳定内参基因的另一种程序。该程序计算原理类似geNorm程序, 且步骤更加简便, 只要计算出候选内参基因的相对表达量2-ΔCt, 然后将此数据导入NormFinder程序, 程序会自动计算出各候选内参基因表达稳定值并给出一个最佳基因。NormFinder分析结果表明, 9个内参基因的稳定性排序为gapdh > EF-1α > Tuba1a > Rpl13 > Ak > UBQ > Rps6 > 18S > 28S (表 2)。
|
|
表 2 NormFinder软件对9个候选内参基因表达情况的统计学分析 Tab.2 Statistic analysis of the 9 candidate internal reference genes based on their threshold cycle values using NormFinder software |
BestKeeper是Michael等[21]编写的用于提供内参基因和目标基因表达量分析的程序。直接将Ct值导入程序相应位置, 通过程序计算可以获得标准偏差(SD)和变异系数(CV)。其中标准偏差和变异系数越小, 内参基因稳定性越好; 标准偏差和变异系数越大, 稳定性越差。此外, 当SD>1时, 则说明该内参基因表达不稳定。BestKeeper分析结果表明, 9个内参基因标准偏差排序为28S > UBQ > Rps6 > Tuba1a > 18S > Rpl13 > Ak > EF-1α > gapdh; 变异系数排序为28S > 18S > Rps6 > UBQ > Ak > Tuba1a > EF-1α > Rpl13 > gapdh(表 3)。
|
|
表 3 BestKeeper软件对9个候选内参基因表达情况的统计学分析 Tab.3 Statistics analysis of the 9 candidate internal reference genes based on their threshold cycle values using BestKeeper software |
通过对9个候选内参基因在拟穴青蟹不同发育阶段的胚胎中的稳定性评估, 发现EF-1α, Rpl13和gapdh在不同内参分析程序中有较好的稳定性; 而两个相对表达量最高的基因18S和28S则表现出较差的稳定性。
3 讨论不同实验条件下, 基因表达量的分析是研究基因功能的重要组成部分。其中qRT-PCR因其快速性和精确性, 是目前研究基因表达水平最广泛使用的方法之一[22]。然而, 一般情况下, 不同实验条件下稳定表达的内参基因不同, 因此建议在进行qRT-PCR之前, 筛选适合实验条件下的内参基因, 而不是使用发表过的其他实验条件下的内参基因。
甲壳动物内参基因的筛选工作已有相关报道[9-13], 但相对于种类繁多和数目巨大的甲壳动物来说尚显不足。目前为止, 胚胎发育过程中的内参筛选主要集中在植物和鱼类, 针对甲壳动物胚胎发育过程内参基因的筛选尚未报道。使用筛选后的内参基因作为内参, 标准化目的基因qRT-PCR的表达量数据, 结果可能与未经筛选的内参基因所得到的结果有显著的差异, 在qRT- PCR实验前建议对实验所使用的内参基因进行筛选验证, 以防出现错误的实验结果[23]。
不同条件下甲壳动物内参基因的筛选已在鲑鱼虱、中华绒螯蟹、斑节对虾等物种中进行研究。Frost等[12]对鲑鱼虱4种候选内参基因在生活史8个不同阶段的表达稳定性进行评估, 最终认为Rps20与EF-1α是最佳内参基因组合; Huang等[9]对中华绒螯蟹10个候选内参基因在不同组织和不同发育阶段中的表达进行分析, 认为UBE和S27为最稳定内参基因, 并且以最不稳定的内参基因GST作为内参基因分析EcR在不同蜕皮阶段表达水平, 结果表明EcR (蜕皮激素受体)表达水平升高, 因此Huang等[9]认为应根据实验条件对内参基因进行评估和选择, 最好选择多个内参基因; Leelatanawit等[11]在不同群体的斑节对虾雌雄性腺中筛选内参基因, 发现EF-1α稳定性较好, 且在生殖相关基因表达水平验证中获得了期望的表达模式; Jiang等[10]对克氏原螯虾8个组织和卵巢6个不同发育时期的最适内参基因进行分析, 最终认为EIF和18S在不同组织中稳定表达, 适合作为内参基因, 而TBP和EIF则在卵巢不同发育阶段中稳定表达, 适合作为内参基因; Dhar等[13]认为gapdh在白斑病感染不同时间的南美蓝对虾肝胰腺中表达量稳定, 适合作为内参基因。以上研究表明, 在不同物种中即便是同为甲壳纲的物种内参基因的稳定性不尽相同[24]。因此, 在进行qRT-PCR前, 需要对内参基因加以筛选。
本研究使用了常用内参筛选程序geNorm, NormFinder和BestKeeper针对不同发育时期的拟穴青蟹胚胎, 从9个候选内参基因中筛选出合适的内参基因。结果表明不同分析软件所获得的最稳定表达的基因和最不稳定表达的基因是相对一致的, 但由于它们的统计算法有所不同, 部分分析结果存在微小差异。其中, geNorm程序得到EF-1α是最稳定的内参基因, gapdh处于中等稳定水平; NormFinder和BestKeeper中gapdh和EF-1α处于第一和第二名的稳定水平。因此, gapdh和EF-1α被认为是拟穴青蟹胚胎发育过程中基因表达量数据标准化的最合适内参基因。由于在geNorm程序中gapdh稳定性一般, 而EF-1α稳定性最好, 另外两个程序中gapdh和EF-1α稳定性差异极小。因此可以选用EF-1α作为后续qRT-PCR的内参基因, 或选用以上双内参作为后续实验的内参基因。
最新研究表明, 许多传统内参基因在不同条件下表达不够稳定[25-26], 本研究的结果也证实这一点。常用的内参基因β-actin, 18S和28S不适用于拟穴青蟹不同发育时期胚胎基因表达量数据的标准化。虽然β-actin在拟穴青蟹成体不同组织中表达稳定, 但是在拟穴青蟹胚胎发育过程中, 其表达在进行筛选的各内参基因中最不稳定, 并且导入内参程序时对其他内参基因稳定值造成很大影响, 因此不适合用于拟穴青蟹不同发育时期胚胎的qRT-PCR分析[27], 这一结果表明在同一物种不同时期的组织中最适内参可能不同[28-29]。综合3个内参筛选程序, 28S是不同胚胎发育时期中最不稳定表达的基因, 不适合作为内参基因。同时, 18S只具有中等稳定性, 也不建议用作内参基因。
| [1] |
Ye H H, Tao Y, Wang G Z, et al. Experimental nursery culture of the mud crab Scylla paramamosain (Estampador) in China[J]. Aquaculture International, 2011, 19(2): 313-321. |
| [2] |
Jiang Q L, Bao C C, Yang Y N, et al. Transcriptome profiling of claw muscle of the mud crab (Scylla paramamosain) at different fattening stages[J]. PLoS ONE, 2017, 12(11): e0188067. DOI:10.1371/journal.pone.0188067 |
| [3] |
Lin Q. Species composition of genus Scylla and genetic diversity of Scylla paramamosain (Estampador, 1949) population in China[D]. Xiamen: Xiamen University, 2008. [林琪.中国青蟹属种类组成和拟穴青蟹群体遗传多样性的研究[D].厦门: 厦门大学, 2008.] http://cdmd.cnki.com.cn/Article/CDMD-10384-2009077594.htm
|
| [4] |
Ding H Y, Yao H F, Li G L, et al. Effect of hatching water pH on embryonic development of Scylla serrata[J]. Journal of Aquaculture, 2010, 31(9): 8-11. [丁宏印, 姚海富, 李贵玲, 等. 孵化水体pH值对锯缘青蟹胚胎发育的影响[J]. 水产养殖, 2010, 31(9): 8-11.] |
| [5] |
Li S J, Wang G Z. Comparative studies on the hydrolytic enzyme activities for the mud crab, Scylla serrata (Forskål) during embryonic development[J]. Journal of Xiamen University (Natural Science), 1995, 34(6): 970-974. [李少菁, 王桂忠. 锯缘青蟹胚胎发育过程中几种水解酶活力的比较研究[J]. 厦门大学学报(自然科学版), 1995, 34(6): 970-974. DOI:10.3321/j.issn:0438-0479.1995.06.020] |
| [6] |
Zeng C S. Induced out-of-season spawning of the mud crab, Scylla paramamosain (Estampador) and effects of temperature on embryo development[J]. Aquaculture Research, 2007, 38(14): 1478-1485. DOI:10.1111/are.2007.38.issue-14 |
| [7] |
Forlenza M, Kaiser T, Savelkoul H F J, et al. The use of real-time quantitative PCR for the analysis of cytokine mRNA levels[M]. Totowa: Humana Press, 2012: 7-23.
|
| [8] |
Gadkar V J, Filion M. New developments in quantitative real-time polymerase chain reaction technology[J]. Current Issues in Molecular Biology, 2014, 16: 1-6. |
| [9] |
Huang S, Chen X W, Wang J, et al. Selection of appropriate reference genes for qPCR in the Chinese mitten crab, Eriocheir sinensis (Decapoda, Varunidae)[J]. Crustaceana, 2017, 90(3): 275-296. DOI:10.1163/15685403-00003651 |
| [10] |
Jiang H C, Qian Z J, Lu W, et al. Identification and characterization of reference genes for normalizing expression data from red swamp crawfish Procambarus clarkii[J]. International Journal of Molecular Sciences, 2015, 16(9): 21591-21605. DOI:10.3390/ijms160921591 |
| [11] |
Leelatanawit R, Klanchui A, Uawisetwathana U, et al. Validation of reference genes for real-time PCR of reproductive system in the black tiger shrimp[J]. PLoS ONE, 2012, 7(12): e52677. DOI:10.1371/journal.pone.0052677 |
| [12] |
Frost P, Nilsen F. Validation of reference genes for transcription profiling in the salmon louse, Lepeophtheirus salmonis, by quantitative real-time PCR[J]. Veterinary Parasitology, 2003, 118(1-2): 169-174. DOI:10.1016/j.vetpar.2003.09.020 |
| [13] |
Dhar A K, Bowers R M, Licon K S, et al. Validation of reference genes for quantitative measurement of immune gene expression in shrimp[J]. Molecular Immunology, 2009, 46(8-9): 1688-1695. DOI:10.1016/j.molimm.2009.02.020 |
| [14] |
Wu J Y, He B, Du Y J, et al. Analysis method of systematically evaluating stability of reference genes using geNorm, NormFinder and BestKeeper[J]. Modern Agricultural Science and Technology, 2017(5): 278-281. [吴建阳, 何冰, 杜玉洁, 等. 利用geNorm、NormFinder和BestKeeper软件进行内参基因稳定性分析的方法[J]. 现代农业科技, 2017(5): 278-281. DOI:10.3969/j.issn.1007-5739.2017.05.174] |
| [15] |
Chen J M. Foundationl studies on the embryonic development of the mud crab, Scylla serrata (Forskål)[D]. Xiamen: Xiamen University, 2005: 1-116. [陈锦民.锯缘青蟹Scylla serrata (Forskål)胚胎发育的基础研究[D].厦门: 厦门大学, 2005: 1-116.]
|
| [16] |
Fang Y B. Cloning and expression profile of two immune-related genes and studies on the applicability of eleven internal reference genes in mud crab, Scylla Paramamosain[D]. Shanghai: Shanghai Ocean University, 2013. [房娅博.拟穴青蟹免疫相关基因的克隆及内参基因的适用性分析[D].上海: 上海海洋大学, 2013.] http://cdmd.cnki.com.cn/Article/CDMD-10264-1014013573.htm
|
| [17] |
Olias P, Adam I, Meyer A, et al. Reference genes for quantitative gene expression studies in multiple avian species[J]. PLoS ONE, 2014, 9(6): e99678. DOI:10.1371/journal.pone.0099678 |
| [18] |
Vandesompele J, De Preter K, Pattyn F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biology, 2002, 3(7): research0034.1-0034.11.
|
| [19] |
Kidd M, Nadler B, Mane S, et al. GeneChip, geNorm, and gastrointestinal tumors:novel reference genes for real-time PCR[J]. Physiological Genomics, 2007, 30(3): 363-370. DOI:10.1152/physiolgenomics.00251.2006 |
| [20] |
Andersen C L, Jensen J L, Ørntoft T F. Normalization of real-time quantitative reverse transcription-PCR data:a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets[J]. Cancer Research, 2004, 64(15): 5245-5250. DOI:10.1158/0008-5472.CAN-04-0496 |
| [21] |
Pfaffl M W, Tichopad A, Prgomet C, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity:BestKeeper-Excel-based tool using pair-wise correlations[J]. Biotechnology Letters, 2004, 26(6): 509-515. DOI:10.1023/B:BILE.0000019559.84305.47 |
| [22] |
Valasek M A, Repa J J. The power of real-time PCR[J]. Advances in Physiology Education, 2005, 29(3): 151-159. |
| [23] |
Dheda K, Huggett J F, Chang J S, et al. The implications of using an inappropriate reference gene for real-time reverse transcription PCR data normalization[J]. Analytical Biochemistry, 2005, 344(1): 141-143. |
| [24] |
Zhang Y F, Zhao L J, Zeng Y L. Selection and application of reference genes for gene expression studies[J]. Plant Physiology Journal, 2014, 50(8): 1119-1125. [张玉芳, 赵丽娟, 曾幼玲. 基因表达研究中内参基因的选择与应用[J]. 植物生理学报, 2014, 50(8): 1119-1125.] |
| [25] |
Dheda K, Huggett J F, Chang J S, et al. The implications of using an inappropriate reference gene for real-time reverse transcription PCR data normalization[J]. Analytical Biochemistry, 2005, 344(1): 141-143. |
| [26] |
Kanakachari M, Solanke A U, Prabhakaran N, et al. Evaluation of suitable reference genes for normalization of qPCR gene expression studies in Brinjal (Solanum melongena L.) during fruit developmental stages[J]. Applied Biochemistry and Biotechnology, 2016, 178(3): 433-450. DOI:10.1007/s12010-015-1884-8 |
| [27] |
Huang J R, Huang H Y, Ye H H, et al. Cloning and analysis of β-actin gene from Scylla paramamosain[J]. Journal of Xiamen University (Natural Science), 2012, 51(2): 274-279. [黄静茹, 黄辉洋, 叶海辉, 等. 拟穴青蟹β-肌动蛋白基因的克隆与分析[J]. 厦门大学学报(自然科学版), 2012, 51(2): 274-279.] |
| [28] |
Liu H F, Gao L X, Hu Y H. Reference genes discovery and selection for quantitative real-time PCR in tree peony seed and petal tissue of different development stages[J]. Journal of Agricultural Biotechnology, 2015, 23(12): 1639-1648. |
| [29] |
Chen L, Zhao W, Zhan S Y, et al. The expression stability analysis of reference genes in the different tissues and skeletal muscle of different development periods in goat[J]. Acta Veterinaria et Zootechnica Sinica, 2014, 45(8): 1228-1236. [陈利, 赵薇, 占思远, 等. 山羊不同组织及不同发育时期骨骼肌内参基因的表达稳定性分析[J]. 畜牧兽医学报, 2014, 45(8): 1228-1236.] |
 2019, Vol. 26
2019, Vol. 26

