2. 中国水产科学研究院淡水渔业研究中心, 农业农村部长江下游渔业资源环境科学观测实验站, 江苏 无锡 214081
2. Scientific Observing and Experimental Station of Fishery Resources, Chinese Academy of Fishery Sciences, Wuxi 214081, China
洄游型刀鲚(Coilia nasus)又称长颌鲚, 是一种具有重要经济价值的小型洄游性鱼类, 多分布在沿海和附近海域的底层。每年春季, 成熟个体集群由海入江进行生殖洄游[1-2]。李文祥等[3]调查了3种生态型刀鲚鳃部、胃、肠和幽门盲囊中寄生蠕虫的种类和感染情况, 其中肠和幽门盲囊包括3种棘头虫:陈氏刺棘虫、鲇异吻钩棘头虫(Arhythmacanthus parasiluri)和长江傲刺棘头虫(Brentisentis yangtzensis), 陈氏刺棘虫在3种生态型的刀鲚中都有较高的感染率和平均丰度。同时陈氏刺棘虫在长江及其相通湖泊的淡水鱼体内被发现, 如短吻间银鱼(Hemisalanx brachyrostralis)、鱤(Elopichthys bambusa)、翘嘴鲌(Erythroculter ilishaeformis)等[4]。
复杂的肠道微生物群是免疫和代谢系统所必需的[5], 而肠道是寄生虫的理想栖息地, 它们与肠道微生物群存在相互作用[6]。寄生虫对宿主的损伤主要包括机械刺激及损伤、掠夺营养和毒素作用; 宿主对寄生虫的影响则主要通过免疫应答产生不同程度的抵抗[7]。迄今为止, 在水产动物方面针对寄生虫感染影响宿主肠道微生物群的研究相对较少, 以往的研究主要针对于寄生虫对宿主的致病作用; 而在哺乳动物方面, 相关的研究结果大部分都表明寄生虫感染会破坏宿主胃肠道微生物群。例如山羊捻转血矛线虫(Haemonchus contortus)感染不影响皱胃的微生物多样性, 但会导致真胃中微生物菌群组成的显著改变, 例如增加了普雷沃氏菌属以及巴斯德氏菌属的丰度, 而丁酸弧菌属在相对丰度上也存在显著差异[8]。还有研究证明寄生虫感染可以调节宿主免疫系统从而治疗宿主炎症, 例如Ramanan等[9]的最新研究证明, 消化道蠕虫感染可以通过启动免疫应答调节共生菌和致病细菌在肠道内的平衡从而治疗炎症性肠病; Broadhurst等[10]发现用寄生虫感染发生慢性腹泻的猕猴, 可以通过减少对肠道细菌的免疫应答来改善肠道炎症的症状。
本实验于刀鲚洄游汛期内在南通江段选取6尾对照组和6尾实验组, 对V3~V4区进行16S rDNA扩增子测序, 首次研究陈氏刺棘虫对洄游型刀鲚肠道微生物群的影响。本实验的研究结果为洄游型刀鲚感染寄生虫后的微生物群变化提供理论依据, 有助于了解洄游型刀鲚、陈氏刺棘虫和肠道微生物群落三者之间的关系, 研究洄游型刀鲚肠道微环境稳态。
1 材料和方法 1.1 样品采集刀鲚样本在2017年7月长江南通江段(120°54'20.88"E, 31°54'20.52"N)采集, 共解剖刀鲚297尾, 从中选取6尾感染陈氏刺棘虫刀鲚作为实验组(平均感染刺棘虫数量为20.5±3.5), 6尾未感染寄生虫刀鲚作为对照组(表 1)。在无菌条件下进行处理, 在低温环境下用无菌剪刀和镊子剪取刀鲚肠道, 挤出内容物, 用无菌水冲洗肠道, 一并放入2 mL无菌冻存管, 置于液氮中保存。
|
|
表 1 对照组和感染陈氏刺棘虫组刀鲚的生物学测量数据 Tab.1 Biologic measurement data of the uninfected and infected Coilia nasus groups |
刀鲚肠道细菌总DNA使用OMEGA公司E.Z.N.A. Stool DNA Kit试剂盒提取, 提取DNA之后分类保存。
实验采用16S rDNA V3~V4区通用引物进行扩增, 所用引物序列为343F前段引物(5'- TACGGRAGGCAGCAG-3ʹ)和798R后段引物(5'-AGGGTATCTAATCCT-3ʹ)。PCR的反应体系为含15 mmol/L MgCl2的10×Buffer 10.0 μL, 2.5 mmol/L dNTP 2.0 μL, 10 μmol/L引物各5 μL, 5 U/μL Taq酶1.0 μL, DNA模板4.0 μL, 灭菌去离子水73 μL, 总体积100 μL。PCR反应条件: 94℃预变性5 min; 94℃变性30 s, 56℃退火30 s, 72℃延伸50 s, 连续20个循环; 最后72℃延伸10 min。PCR产物通过1%琼脂糖凝胶电泳检测。PCR扩增产物经纯化和定量后, 在每个样品接头加上不同的Barcode序列, 用Illumina MiSeq高通量测序技术序列测定和分析。
1.3 数据分析首先对原始双端测序数据进行去杂并拼接, 随后进行质控, 去除低质量的碱基序列、模糊碱基序列以及Barcode, 获得Clean reads, 用UCHIME[11]去除嵌合体, 进行下游分析, 并对所有的优质序列以97%相似度进行操作分类单元(Operational Taxonomic Unit, OTU)划分, 基于OTU结果计算样品的Alpha多样性。随后从各OTU中挑选丰度最大的序列作为代表序列, 与Silva数据库(V123)进行比对,在不同分类学水平上进行物种注释和物种丰度统计; 通过Excel分析并绘制门和科分类水平的相对丰度图表; 使用R语言制作绘制Venn图, 并对Weighted的UniFrac距离矩阵进行NMDS分析, 描述群落样本的结构分布; 使用线性判别分析(LDA)效应大小(LEfSe)算法比较对照组和感染组中刀鲚肠道微生物组的组成(相对丰度)差异。
2 结果与分析 2.1 共有OTU分析根据两组12个样品Venn图显示, 共有OTU数为32个(图 1), 其所对应的微生物群为邻单胞菌属(Plesiomonas)、消化链球菌属(Peptostreptococcus)、弧菌属(Vibrio)、发光杆菌属(Photobacterium)、Epulopiscium(未分类的梭菌)、梭状芽孢杆菌(Clostridium prazmowski)、厌氧芽孢杆菌属(Clostridium sensu stricto)、Defluviitaleaceae、Lachnoclostridium、理研菌科RC9肠道菌群(Rikenellaceae_RC9_gut_group)、弓形菌属(Arcobacter)、拟普雷沃菌属(Alloprevotella)、Psychrilyobacter、Candidatus hepatoplasma、拟杆菌属(Bacteroides)、螺旋菌属(Spirochaeta)、支原体细菌(Mycoplasmataceae bacterium)、幽门螺杆菌属(Helicobacter)、Sunxiuqinia。
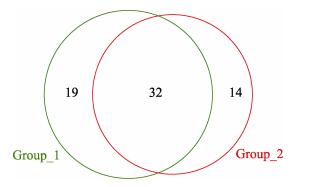
|
图 1 对照组和感染组共有OTU分布图 Group_1表示对照组, Group_2表示感染组. Fig.1 OTU distribution in uninfected and infected group Group_1: uninfected groups, Group_2: infected groups. |
使用Qiime软件对各样品均一化处理后的数据进行多样性分析, 计算Observed Species和3种Alpha多样性指数, 包括Chao1指数(Chao1 richness estimator)(图 2A)、香农指数(Shannon Wiener index)和辛普森多样性指数(Simpson index)(图 2B), 结果表明对照组和感染组之间的肠道微生物Alpha多样性差异并不显著。如Shannon指数(对照组3.0±1.72, 感染组3.05±0.61)没有显著差异(P > 0.05);另外, 对照组和感染组系统发育多样性(PD)(对照组为13.34±7.95, 感染组为17.03±3.57)也没有显著差异(P > 0.05)。
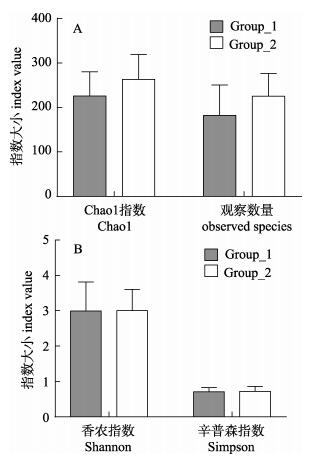
|
图 2 对照组和感染陈氏刺棘虫组刀鲚肠道的微生物多样性指数
A. Chao1指数和观察数量; B.香农指数和辛普森指数. Group_1表示对照组, Group_2表示感染组. Fig.2 Diversity index of intestinal microbiome between uninfected and infected Coilia nasus A. Chao1 index and observed species; B. Shannon Wiener index and Simpson index. Group_1: uninfected group, Group_2: infected group. |
Beta多样性分析基于weight UniFrac的NMDS (图 3), 图中可以看出12个样品距离相近, 两组样本间的距离也相近, 此结果表明对照组和感染组刀鲚肠道微生物群落结构组成较为相似。
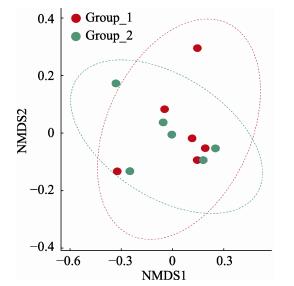
|
图 3 单个肠道样本基于Weight UniFrac的NMDS图 Group_1表示对照组, Group_2表示感染组. Fig.3 NMDS plots of the microbiomes of individual intestinal samples base on Weight UniFrac Group_1: uninfected group; Group_2: infected group. |
对照组刀鲚肠道微生物群共鉴定出22个门类, 感染组鉴定出18个门类, 其中Latescibacteria, Parcubacteria, 浮霉菌门(Planctomycetes)和WCHB1_60只在对照组中被发现, 而疣微菌门(Verrucomicrobia)只在感染组中检测到。数据结果(图 4)显示, 对照组和感染组的刀鲚肠道微生物群结构较为相似, 两组鱼肠道微生物中丰度较高(相对丰度≥0.5%)的4类菌门分别为变形杆菌门(Proteobacteria)、厚壁菌门(Firmicutes)、软壁菌门(Tenericutes)和拟杆菌门(Bacteroidetes), 相对丰度总量均高于92%。其中, 变形菌门和厚壁菌门含量远高于其他菌门, 为两组刀鲚肠道中的优势菌, 变形菌门、软壁菌门和拟杆菌门在对照组中的含量高于感染组, 而感染组厚壁菌门的含量高于对照组, 另外梭杆菌门在感染组中的比例(6.26%)远高于对照组(0.85%), 但两组间没有显著性差异(P > 0.05)。
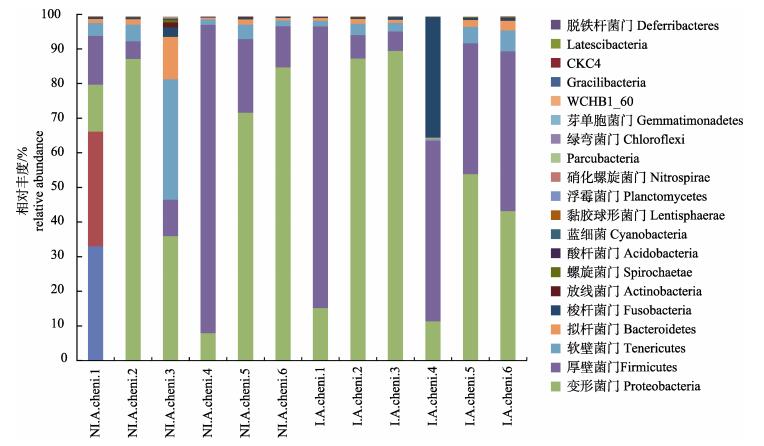
|
图 4 刀鲚肠道门分类水平的微生物组成 NI.A.cheni1-NI.A.cheni6为Group_1对照组, I.A.cheni1-I.A.cheni6为Group_2感染组. Fig.4 Phylum-level gut microbiota composition of Coilia nasus NI.A.cheni: uninfected controls Group_1; I.A.cheni: infected group Group_2. |
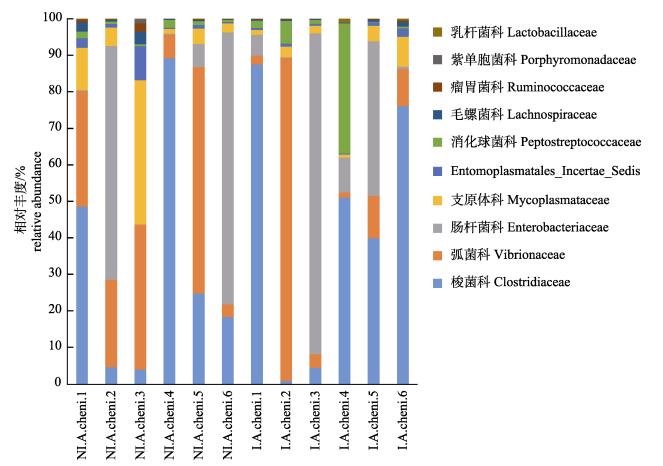
筛选出每个样品中相对丰度较高的细菌, 统计各样品在科水平上的菌群结构(图 5), 在已鉴定出的130个科中, 相对丰度较高的共有7个(≥0.5%), 其中弧菌科(Vibrionaceae)、支原体科(Mycoplasmataceae)、Entomoplasmatales_Incertae_ Sedis和毛螺菌科(Lachnospiraceae)在对照组中含量低于感染组, 而梭菌科(Clostridiaceae)、肠杆菌科(Enterobacteriaceae)和消化球菌科(Peptostreptococcaceae)在感染组中含量高于对照组, 但差异性分析结果显示这7个科没有显著性差异(P > 0.05)。在丰度较低的科中, 链球菌科(Streptococcaceae)和乳杆菌科(Lactobacillaceae)在感染组中的丰富度高于对照组。
|
图 5 刀鲚肠道科分类水平对照组和感染组微生物群的差异 NI.A.cheni1-NI.A.cheni6为Group_1对照组, I.A.cheni1-I.A.cheni6为Group_2感染组. Fig.5 Differences in bacterial communities at family level of the Coilia nasus gut in the two groups NI.A.cheni: uninfected controls Group_1; I.A.cheni: infected group Group_2. |
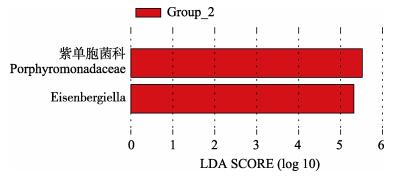
基于多重假设检验校正的Wilcoxon非参数t检验, 有且仅有2种OTU的相对丰度存在显著差异(P < 0.05)。LDA Score显示对照组和感染组之间的丰度具有显著差异(LDA Score≥2.0)。紫单胞菌科(Porphyromonadaceae)和Eisenbergiella在感染组中的相对丰度显著高于对照组(图 6)。
|
图 6 两组刀鲚之间LDA得分≥2.0的2个具有显著不同丰度的分类群 Group_2表示感染组. Fig.6 Two significantly discriminative taxa between the uninfected control and infected Coilia nasus with an absolute Linear Discriminant Analysis LDA score ≥2.0 Group_2: infected group. |
以往研究鱼类肠道微生物的分子生物学方法主要有变性梯度凝胶电泳(PCR-DGGE)[12]、末端片段长度多态性等指纹图谱技术(T-RFLP)[13]等, 但这些传统方法具有一定的局限性。高通量测序技术可以对肠道微生物进行高灵敏度的精确定量, 获得覆盖整个微生物群落的信息, 根据16S rDNA的V3~V4可变区测序结果, 能够对微生物群落有更加全面的认识。本研究首次使用16S rDNA高通量测序研究长江洄游刀鲚肠道微生物群, 共检测出22个门、37个纲、85个目和130个科的细菌种类。其中绝对优势菌门为变形菌门, 次级优势菌为厚壁菌门, 与草鱼(Ctenopharyngodon idellus)、鲫(Carassius auratus)[14]和大西洋鲑(Salmo salar)[15]等肠道菌群分析结果一致。另外刀鲚肠道内还检测到软壁菌门、拟杆菌门、放线菌门和绿弯菌门, 与聂志娟等[16]利用PCR-DGGE指纹图谱技术测定的结果相似, 同时本实验还鉴定出脱铁杆菌门、纤维杆菌门、螺旋菌门、酸杆菌门、黏胶球形菌门、浮霉菌门、硝化螺旋菌门、芽单胞菌门等丰度低于0.1%的微生物群。
3.2 刀鲚肠道微生物菌群组间差异关于胃肠道寄生虫感染对鱼类肠道中微生物群影响的相关报道很少, 本研究首次研究了陈氏刺棘虫对刀鲚肠道微生物群落结构组成的影响。结果表明感染陈氏刺棘虫的刀鲚肠道微生物群落多样性与未感染组比较没有显著差异(P > 0.05, 图 1)。
门分类水平下, 变形菌门和厚壁菌门均为对照组和感染组的优势菌群, 陈氏刺棘虫的感染并不影响其优势地位。疣微菌门只在感染组刀鲚肠道中检测到, 而软壁菌门的比例有下降趋势, 这与Nagalingam等[17]的研究结果相一致, 该研究用含有5%葡聚糖硫酸钠(DSS)的饮用水处理野生型小鼠, 诱导结肠炎后检测胃肠组织微生物群, 发现疣微菌门仅存在于DSS处理小鼠中, 并且软壁菌门比例也有所减少, 说明疣微菌门可能与肠炎的发生有关, 这也解释了感染组中疣微菌门的出现。
在科水平对两组刀鲚肠道微生物丰富度进行比较分析, 发现感染组肠杆菌科、消化球菌科和乳杆菌科丰富度均有所上升。肠杆菌科是一类革兰氏阴性杆菌, 部分为条件致病菌。Rausch等[18]在线虫感染后第14天的小鼠的结肠中也检测到肠杆菌科丰富度具有升高趋势。Ahn[19]研究也发现结直肠癌患者较正常人类肠道中消化球菌科含量显著增加, 而毛螺菌科含量显著较少。另外Walk等[20]用线虫(Heligmosomoides polygyrus)感染小鼠后发现, 乳杆菌科含量均呈上升趋势, 这与本实验中的结果很相似, 而Reynolds[21]的研究结果也显示乳杆菌科中一些菌属的丰度与小鼠肠道寄生线虫(H. polygyrus)的感染呈正相关。这说明肠杆菌科、消化球菌科和乳杆菌科丰富度上升与刺棘虫有一定相关性。
紫单胞菌科(Porphyromonadaceae)和Eisenbergiella在刺棘虫感染组中的相对丰度显著提高。紫单胞菌科(Porphyromonadaceae)属于拟杆菌门拟杆菌目, 紫单胞菌科的许多种属是人类和动物胃、肠道和口腔的原生微生物群的一部分, 但该家族中的一些物种通常与各种人和动物的感染有关[22]。Eisenbergiella是厚壁菌门毛螺菌科一种拟议的属, 其中一个种是Eisenbergiella tayi, 它是一种厌氧菌, 丁酸是其主要发酵终产物[23], 也是结肠细胞的主要能量来源, 并且参与维持结肠黏膜健康[24], 感染组刀鲚肠道中Eisenbergiella相对丰度的显著升高可能是宿主自发产生的一种应激反应, 即通过增加有益微生物群来消除寄生虫感染的危害。
3.3 刀鲚肠道内环境稳态本实验的研究结果表明, 陈氏刺棘虫感染组和对照组刀鲚肠道微生物群落结构和多样性水平未表现出显著差异。一些相关的研究也体现了相似的结果, 其中Cooper等[25]的研究比较了居住在厄瓜多尔的人类粪便样本中的微生物群落, 16S rDNA基因测序结果显示未感染的实验者或携带鞭虫(Trichuris trichiura)的实验者的微生物群落组成没有明显差异; 而对于鞭虫和蛔虫(Ascaris lumbricoides)共同感染的受试者, 厚壁菌门中梭菌属的丰度下降并且总体微生物多样性降低。同样, Cantacessi等[26]将钩虫人为感染至健康的人类受试者体内, 8周后取粪便检测, 结果并没有观察到寄生虫对微生物群落结构的明显影响, 但受感染个体的微生物丰富度有增加的趋势。Wu等[27]观察到感染了猪鞭毛虫(Trichuris suis)21 d的猪近端结肠微生物群的组成表现出显著变化, 如琥珀酸弧菌属(Succinivibrio)和瘤胃球菌属(Ruminococcus)等13%属的相对丰度显著降低。但有研究表明在接种感染猪鞭虫后7~9周之间宿主有一些自身治愈反应, 例如改变了小肠中的脂肪酸吸收[28]。
宿主、肠道微生物群和寄生虫之间的多维联系组成了一个复杂的生态系统, 其中一种组分的改变会引起其余组分的应答反应[29]。微生物群落会因肠道受到寄生虫活动的刺激而发生变化, 因此刀鲚在感染陈氏刺棘虫后, 刀鲚的肠道微生物群落随之发生相应改变; 另一方面, 肠道寄生虫与微生物群落相互作用, 改变宿主和肠道微生物群之间的平衡, 刀鲚通过调节肠道微生物群结构来抵御寄生虫带来的不良影响, 例如刀鲚肠道自身可能通过改变产生丁酸的细菌丰度来调节肠道丁酸的合成。鱼类肠道微生物群落的建立是一个长期进化的过程, 很多研究也表明, 肠道微生物群与宿主的免疫和代谢系统有着密切而复杂的关系[30], 在病原体侵入时鱼类肠道共生菌通过调节宿主的免疫系统维持内部的动态平衡[31], 而本研究结果未体现出寄生虫感染后刀鲚肠道微生物的显著变化也可能和肠道微生物的定植能力有关。同时, 肠道微生物群可影响寄生虫的感染与活力[30], 这需待后续实验进行探究。
4 结论两组刀鲚肠道微生物群结构没有显著差异, 但在某些微生物的丰富度上有微小变化。例如感染组中疣微菌门、肠杆菌科和乳杆菌科含量发生了变化, 并且紫单胞菌科和Eisenbergiella相对丰度显著增加。本研究结果表明, 洄游型刀鲚在陈氏刺棘虫感染强度不高时也具有保持其肠道微生物群稳定性的能力, 这体现洄游型刀鲚在其生长过程中保持内环境稳态的能力和对生态环境的适应性。洄游型刀鲚、陈氏刺棘虫和肠道微生物群之间的相互作用机制, 需要进一步从功能互作上进行验证, 为刀鲚洄游过程中的摄食、繁殖等行为的研究提供参考。由于受到实验样本数量的限制, 以及感染强度、感染周期、个体体质差异等因素对实验结果产生的影响, 后续将进一步通过实验对本研究结果进行验证。
| [1] |
Liu Y L, Wu Z Q, Hu M L. Advances on tapertail anchovy Coilia ectenes in China[J]. Fisheries Science, 2008, 27(4): 205-209. [刘引兰, 吴志强, 胡茂林. 我国刀鲚研究进展[J]. 水产科学, 2008, 27(4): 205-209. DOI:10.3969/j.issn.1003-1111.2008.04.012] |
| [2] |
Cooperative Organization for Investigation of Coilia ectenes Resources in the Yangtze River. Resource and utilization of Coilia ectenes in the Yangtze River[J]. Freshwater Fisheries, 1976(8): 24-26. [长江刀鲚资源调查协作组. 长江刀鱼资源及其利用[J]. 淡水渔业, 1976(8): 24-26.] |
| [3] |
Li W X, Wang G T. Helminth communities in Coilia nasus from anadromous, freshwater and landlocked stocks[J]. Chinese Journal of Zoology, 2014, 49(2): 233-243. [李文祥, 王桂堂. 洄游型、淡水型和陆封型刀鲚的寄生蠕虫群落结构[J]. 动物学杂志, 2014, 49(2): 233-243.] |
| [4] |
Yu Y, Wu H S. Studies on the fauna of Acanthocephala of fishes from middle reaches of the Changjiang (Yangtze) River[J]. Acta Hydrobiologica Sinica, 1989, 13(1): 38-50. [余仪, 伍惠生. 长江中游鱼类寄生棘头虫区系的研究[J]. 水生生物学报, 1989, 13(1): 38-50. DOI:10.3321/j.issn:1000-3207.1989.01.002] |
| [5] |
Macpherson A J, Harris N L. Interactions between commensal intestinal bacteria and the immune system[J]. Nature Reviews Immunology, 2004, 4: 478-485. DOI:10.1038/nri1373 |
| [6] |
Liu L L. The interaction between Trichinella spiralis and intestinal microbes[D]. Changchun: Jilin University, 2012. [刘丽丽.旋毛虫与肠道微生物间相互作用的研究[D].长春: 吉林大学, 2011.] http://cdmd.cnki.com.cn/Article/CDMD-10183-1011102184.htm
|
| [7] |
Ren Y X, Sun M Z, Qin Y H, et al. Relationship between parasite and host[J]. Chinese Journal of Microecology, 2014, 24(1): 1055-1056. [任一鑫, 孙明忠, 秦元华, 等. 寄生虫与宿主的关系[J]. 中国微生态学杂志, 2014, 24(1): 1055-1056.] |
| [8] |
Li R W, Li W, Sun J, et al. The effect of helminth infection on the microbial composition and structure of the caprine abomasal microbiome[J]. Scientific Reports, 2016, 6: 20606. DOI:10.1038/srep20606 |
| [9] |
Ramanan D, Bowcutt R, Lee S C, et al. Helminth infection promotes colonization resistance via type 2 immunity[J]. Science, 2016, 352(6285): 608-612. DOI:10.1126/science.aaf3229 |
| [10] |
Broadhurst M J, Ardeshir A, Kanwar B, et al. Therapeutic helminth infection of macaques with idiopathic chronic diarrhea alters the inflammatory signature and mucosal microbiota of the colon[J]. PLoS Pathogens, 2012, 8(11): e1003000. DOI:10.1371/journal.ppat.1003000 |
| [11] |
Edgar R C, Haas B J, Clemente J C, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2194-2200. DOI:10.1093/bioinformatics/btr381 |
| [12] |
Muyzer G. Denaturing gradient gel electrophoresis (DGGE) in microbial ecology: Electrophoresis-Principles and Basics[D]. 2004, 3.4.4: 743-769.
|
| [13] |
Osborn A M, Moore E R B, Timmis K N. An evaluation of terminal-restriction fragment length polymorphism (T-RFLP) analysis for the study of microbial community structure and dynamics[J]. Environmental Microbiology, 2010, 2(1): 39-50. |
| [14] |
Li T T, Li A H. Comparison of the community structure of three different kinds of intestinal microbes in fish by high-throughput sequencing[C]//Proceedings of the Member Conference and Academic Annual Meeting of Hubei and Wuhan Microbiology Society, 2013. [李彤彤, 李爱华.应用高通量测序技术比较三种不同鱼肠道微生物的群落结构[C]//湖北省暨武汉微生物学会会员代表大会暨学术年会, 2013.] http://cpfd.cnki.com.cn/Article/CPFDTOTAL-HDHY201311018177.htm
|
| [15] |
Dehler C E, Secombes C J, Sam M. Seawater transfer alters the intestinal microbiota profiles of atlantic salmon (Salmo salar L.)[J]. Scientific Reports, 2017, 7: Article No. 13877. DOI:10.1038/s41598-017-13249-8 |
| [16] |
Nie Z J, Xu G C, Du F K, et al. PCR-DGGE fingerprinting and diversity analysis of the predominant bacterial community in Coilia nasus[J]. Acta Hydrobiologica Sinica, 2015, 39(5): 1019-1026. [聂志娟, 徐钢春, 杜富宽, 等. 长江刀鲚体内菌群PCR-DGGE指纹图谱及多样性比较分析[J]. 水生生物学报, 2015, 39(5): 1019-1026.] |
| [17] |
Nagalingam N A, Kao J Y, Young V B. Microbial ecology of the murine gut associated with the development of dextran sodium sulfate-induced colitis[J]. Inflammatory Bowel Diseases, 2015, 17(4): 917-926. |
| [18] |
Rausch S, Held J, Fischer A, et al. Small intestinal nematode infection of mice is associated with increased enterobacterial loads alongside the intestinal tract[J]. PLoS ONE, 2013, 8(9): e74026. DOI:10.1371/journal.pone.0074026 |
| [19] |
Ahn J, Sinha R, Pei Z H, et al. Human gut microbiome and risk for colorectal cancer[J]. Journal of the National Cancer Institute, 2013, 105(24): 1907-1911. DOI:10.1093/jnci/djt300 |
| [20] |
Walk S T, Blum A M, Ewing S A, et al. Alteration of the murine gut microbiota during infection with the parasitic helminth Heligmosomoides polygyrus[J]. Inflammatory Bowel Diseases, 2010, 16(11): 1841-1849. DOI:10.1002/ibd.21299 |
| [21] |
Reynolds L A. Immune response and the intestinal microbiota in control of susceptibility to Heligmosomoides polygyrus[OL].[2013-06-29]. https://www.era.lib.ed.ac.uk/handle/1842/7960.
|
| [22] |
Sakamoto M. The Family Porphyromonadaceae[M]. Heidelberg: Springer, 2014: 811-824.
|
| [23] |
Jousimies-Somer H R, Summanen P, Citron D M, et al. Wadsworth-KTL Anaerobic Bacteriology Manual[M]. Belmont: Star Publisher, 2002.
|
| [24] |
Amir I, Bouvet P, Legeay C, et al. Eisenbergiella tayi gen. nov., sp. nov., isolated from human blood[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(3): 907-914. |
| [25] |
Cooper P, Walker A W, Reyes J, et al. Patent human infections with the whipworm, Trichuris trichiura, are not associated with alterations in the faecal microbiota[J]. PLoS ONE, 2013, 8(10): e76573. DOI:10.1371/journal.pone.0076573 |
| [26] |
Cantacessi C, Giacomin P, Croese J, et al. Impact of experimental hookworm infection on the human gut microbiota[J]. Journal of Infectious Diseases, 2014, 210(9): 1431-1434. DOI:10.1093/infdis/jiu256 |
| [27] |
Wu S, Li R W, Li W, et al. Worm burden-dependent disruption of the porcine colon microbiota by Trichuris suis infection[J]. PLoS ONE, 2012, 7(4): e35470. DOI:10.1371/journal.pone.0035470 |
| [28] |
Kringel H, Roepstorff A. Trichuris suis population dynamics following a primary experimental infection[J]. Veterinary Parasitology, 2006, 139(1): 132-139. |
| [29] |
Yang G L, Xing X. Advances in the study on the interaction between helminths and intestinal flora[J]. Chinese Journal of Microecology, 2017, 29(9): 1087-1089. [杨桂连, 邢鑫. 蠕虫感染与肠道菌群相互作用的研究进展[J]. 中国微生态学杂志, 2017, 29(9): 1087-1089.] |
| [30] |
Zaiss M M, Harris N L. The interaction of commensal intestinal bacteria with helminth parasites[J]. Parasite Immunology, 2015, 38(1): 5-11. |
| [31] |
Sun J F, Jia Y L. Advances in intestinal microbiota and immunomodulation in fish[J]. Fisheries Science & Technology Information, 2016, 43(2): 96-100. [孙敬锋, 贾艳丽. 鱼类肠道菌群与免疫调节研究进展[J]. 水产科技情报, 2016, 43(2): 96-100.] |
 2019, Vol. 26
2019, Vol. 26

