2. 宁波大学海洋学院, 浙江 宁波 315211
2. School of Marine Sciences, Ningbo University, Ningbo 315211, China
大弹涂鱼(Boleophthalmus pectinirostris)属虾虎鱼目(Gobiiformes)、虾虎鱼科(Gobiidae)、弹涂鱼属(Boleophthalmus), 是一种暖水、广温和广盐性的滩涂养殖经济鱼类, 具有广阔市场前景[1]。之前的研究表明, 作为一种近岸穴居鱼类, 大弹涂鱼虽然栖息在环境复杂的潮间带泥滩, 但却具有很强的抗病能力[2]。因此, 其能适应不同环境刺激的免疫系统具有非常重要的研究价值。
主要组织相容性复合体(major histocompatibility complex, MHC)是染色体上的一组编码免疫球蛋白受体的基因遗传区域, 这个区域由许多紧密连锁并高度多态的基因群构成[3-4]。根据MHC基因编码的产物结构与功能的不同, MHC基因可分为I类和Ⅱ类, 它们的主要功能是参与适应性免疫, 通过特异性识别内源性和外源性抗原并提呈给T细胞, 从而发挥免疫作用[5-7]。目前已在多种鱼类上分离和鉴定到了MHC Ⅰ类基因, 如斑马鱼(Danio rerio)[8]、中华鲟(Acipenser sinensis)[9]、虹鳟(Oncorhynchus mykiss)[10]和大西洋鲑(Salmo salar)[11]等。随着研究的不断深入, 近年来已确定MHC Ⅰ类基因几乎能在所有现存的有颌脊椎动物中找到, 即使在最古老的类群中也能找到这类基因[12], 这表明在物种进化的过程中, MHC Ⅰ类基因一直发挥着不可替代的作用。目前, 关于大弹涂鱼MHC Ⅰ类基因的研究还未有报道。
盐度是能够影响水生动物生存的重要环境因子, 尤其是在鱼类的呼吸代谢和生长发育等方面具有显著影响[13-15]。例如在低盐度环境下处理银鲳(Pampus argenteus), 其肾中谷胱甘肽S-转移酶活性显著上升[16]; 研究发现水体盐度变化可以影响虹鳟糖及氨基酸等营养物质在肠内的运输, 并在脂肪代谢过程中对脂肪酶活性起重要的调节作用[17]。目前, 关于盐度胁迫对鱼类免疫基因的影响则鲜有报道。为研究高盐胁迫下病毒感染对大弹涂鱼免疫基因的影响, 本研究旨在分析大弹涂鱼MHC Ⅰα基因的序列特征, 评估大弹涂鱼与其他已鉴定鱼类MHC Ⅰα基因的进化关系, 分析其mRNA在不同组织中的表达以及在高盐刺激下注射poly(I:C) (polyinosinic : polycytidylic acid, 是一种免疫兴奋剂, 它以钠盐的形式被用来模拟病毒感染)后的表达情况, 为进一步研究大弹涂鱼的免疫机制奠定理论基础。
1 材料与方法 1.1 实验材料与试剂用于本实验的大弹涂鱼购于浙江省宁波慈溪市山下村的滩涂围养户, 体重(21±0.5) g, 于宁波大学高等技术研究院鱼类健康与进化研究室控温暂养一周(室温20℃, 水体盐度为15, 用海水晶配制), 每天换水一次。
病毒感染模拟物poly(I:C)购于西格玛奥德里奇贸易有限公司(上海, 中国), 用于RNA提取的TaKaRa RNAiso Plus试剂购于大连宝生物工程有限公司(大连, 中国), 分子量标记物GeneRulerTM 1kb DNA Marker购于生工生物工程股份有限公司(上海, 中国), 用于反转录和荧光定量分析的TransScript® All-in-one First-Strand cDNA Synthesis SuperMix for qPCR试剂盒购于全式金生物技术有限公司(北京, 中国)。
1.2 实验方法 1.2.1 实验设计在暂养一周后, 将180尾体质健康、活力旺盛的大弹涂鱼, 随机分为两组, 对照组和实验组(水体盐度均为30), 每组设3个重复, 在6个50 L的水族箱内进行实验, 24 h泵气保证氧气充足。根据预实验结果, 将大弹涂鱼用MS 222 (TMS, tricaine methanesulfonate)麻醉后, 实验组腹腔注射0.2 mL 0.15 μg/μL poly(I:C), 对照组腹腔注射等体积的0.9% NaCl生理盐水。在注射后0 h、6 h、12 h、24 h、36 h、48 h、72 h时间点将大弹涂鱼组织(肝、脾、肠、头肾、皮肤、鳃、脑和肌肉)取出, 迅速放入液氮中, 然后转移到–80℃冰箱保存。
1.2.2 RNA提取与序列扩增采用RNAiso Plus试剂, 根据制造商要求, 提取大弹涂鱼各组织总RNA, 并采用TransGen试剂盒进行反转, 得到可以用于实时荧光定量的cDNA模板。根据实验室已获得的大弹涂鱼cDNA文库, 筛选出MHC Ⅰα基因并验证。在NCBI (https://www.ncbi.nlm.nih.gov/ncbisearch)公共数据库中检索获得近缘物种的相关序列, 在MHC 1α基因编码序列两端的保守区, 利用软件Primer Premier 6.0设计特异性引物, 所有引物均由生工生物工程股份有限公司(上海, 中国)合成并验证(表 1)。PCR反应体系为(10 μL): 0.4 μL正向引物、0.4 μL反向引物、1 μL cDNA模板(0.1 μg/μL)、5 μL的Taq酶和3.2 μL无菌水, 反应程序如下: 95℃ 5 min; 95℃ 15 s, 60℃ 15 s, 72℃ 1 min, 循环30次; 72℃ 5 min。
|
|
表 1 本研究中用到的引物 Tab.1 The primers used for this study |
为了研究大弹涂鱼MHC Ⅰα基因在健康鱼体和病毒感染后的表达情况, 进行了荧光定量实验(real-time quantitative PCR), 将MHC Ⅰα基因和β-actin基因的cDNA模板以5为单位进行6个梯度的稀释, 用于制作目的基因和内参基因的标准曲线。实时荧光定量PCR反应体系为20 μL, 其中包括0.4 μL正向引物、0.4 μL反向引物、0.4 μL cDNA模板(0.1 μg/μL)、10 μL TransStart TipGreen qPCR SuperMix、8.8 μL无菌水。热循环系统反应条件为: 95℃ 5 min, 95℃ 15 s, 40个循环; 60℃ 25 s, 72℃ 25 s, 3次重复实验。
1.3 数据分析该实验所得数据用平均值±标准差x±SD表示, 利用SPSS 22.0统计软件进行单因素方差分析(one-way ANOVA)以及独立样本t检验来检测数据均值差异的显著性, P < 0.05表示具有显著性差异, P < 0.01表示极显著差异。利用OriginPro 8图表软件进行柱状图绘制。
1.4 生物信息学分析其他物种序列由在线程序NCBI获得; 序列同源性比对和相似性搜索用在线程序BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行; 采用DNAMAN软件预测氨基酸序列以及开放阅读框; 利用在线程序PHDsec (https://prabi.ibcp.fr/htm/site/web/home)和SOPMA (https://prabi.ibcp.fr/htm/site/web/home)预测氨基酸二级结构; 采用在线程序SignalP 4.1 (http://www.cbs.dtu.dk/services/SignalP/)预测信号肽序列; 利用在线程序SMART (http://smart.embl-heidelberg.de/)预测蛋白质结构; 利用DNAStar软件预测蛋白质的分子量和等电点; 采用MEGA5.0软件构建系统发生树[18]。
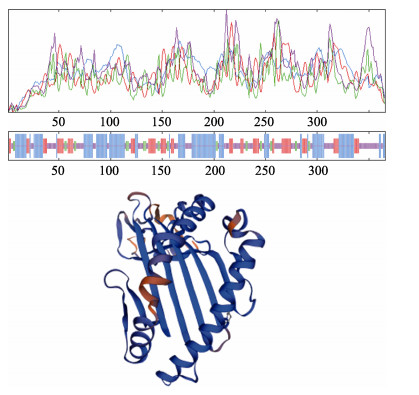
2 结果与分析 2.1 MHC Ⅰα基因序列特征大弹涂鱼MHC Ⅰα基因cDNA序列共有1445个核苷酸, 包括一个由1101个核苷酸组成的完整开放阅读框(ORF), 编码一个由366个氨基酸组成的蛋白(图 1), 包括5个结构域:前导肽、α1结构域、α2结构域、α3结构域和跨膜区。其分子质量约为41.1 kD, 等电点为4.83。其二级结构经PHDsec进行预测, 结果表明, α-螺旋(α-helix)占37.16%, β-转角(β-turn)占7.65%, 延伸链(extended strand)占21.31%, 无规则卷曲(random coil)占33.88%, 具有1个由29个氨基酸组成的前导肽(leader peptide); 3个蛋白激酶C-磷酸化位点(S/T-X-R/K); 1个酪蛋白激酶Ⅱ磷酸化位点(S/T-X-X-D); 1个N-糖基化位点(N-X-S/T); 和1个由23个氨基酸构成的跨膜区(transmembrane region)(图 2)。

|
图 1 大弹涂鱼MHC Ⅰα基因cDNA序列和推导的氨基酸预测 起始密码子用圆圈表示, 终止密码子用星号(*)表示, N连接糖基化位点用方框表示, 蛋白激酶C磷酸化位点以直下划线表示, 酪蛋白激酶Ⅱ磷酸化位点用双直线表示, 前导肽显示为阴影, 跨膜区域用虚线表示. Fig.1 cDNA and deduced amino acid sequence of MHC Ⅰα gene of Boleophthalmus pectinirostris The start codon is circled and the stop codon is indicated by an asterisk (*); N-linked glycosylation sites are represented with a box; protein kinase C phosphorylation sites are underlined; casein kinase Ⅱ phosphorylation sites is underlined with a double straight line; leader peptide is shown in shadow; the transmembrane region is underlined with a dashed line. |
|
图 2 大弹涂鱼MHC Ⅰα蛋白二级结构预测和三级结构模式图 蓝色线条表示α-螺旋(α-helix), 绿色线条表示β-转角(β-turn), 红色线条表示延伸链(extended strand), 无规则卷曲(random coil)则用紫色线条表示. Fig.2 The predicted protein secondary and tertiary structure ideographs of MHC Ⅰα gene of Boleophthalmus pectinirostris The blue line represents α-helix; the green line represents β-turn; the red line represents extended strand; and the random coil is represented by purple line. |
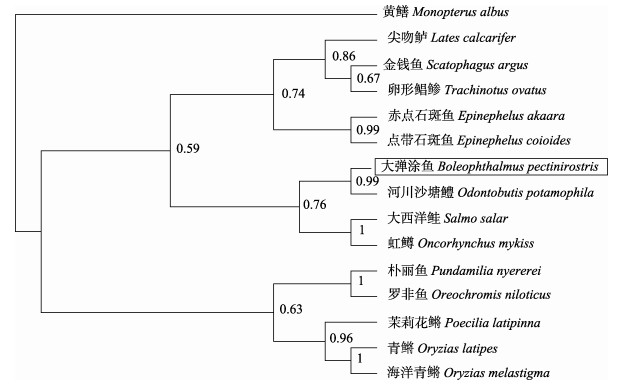
为了研究大弹涂鱼和其他已鉴定鱼类MHC Ⅰα基因的进化关系, 采用MEGA5.0软件中的邻近法(NJ法, 可靠性由1000次自展分枝检验)构建系统发生树(图 3), 所用到的物种及其MHC Ⅰα基因登录号见表 2。如图 3所示, 不同的物种聚成几枝, 大弹涂鱼与河川沙塘鳢(Odontobutis potamophila)最先聚成独立分枝, 然后与鲑科鱼[包括虹鳟(Oncorhynchus mykiss)和大西洋鲑(Salmo salar)]形成第一枝, 再与剩下的鲈形目鱼类聚在一起, 最后与由丽鱼科[尼罗罗非鱼(Oreochromis niloticus)和朴丽鱼(Pundamilia nyererei)]和怪颌鳉科[茉莉花鳉(Poecilia latipinna)和青鳉(Oryzias latipes)]组成的另一枝聚在一起。
|
|
表 2 本研究中所涉及的物种名称及登录号 Tab.2 The species and Chinese names and accession numbers of MHC Ⅰα sequences used in this study |
|
图 3 大弹涂鱼MHC Ⅰα基因和其他鱼类的系统发生树 节点上的数字表示后验概率.大弹涂鱼用方框标出. Fig.3 Phylogenetic tree of MHC Ⅰα of Boleophthalmus pectinirostris and other fish The numbers on the nodes represent the posterior probability. The great blue-spotted mudskipper is indicated by box. |
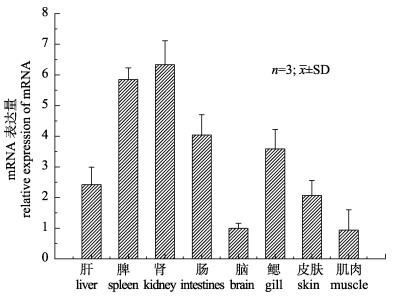
以健康、未进行注射感染实验的大弹涂鱼组织(肝、脾、肾、鳃、脑、肠、肌肉和皮肤)为材料, 以反转录合成的第一链cDNA为模版, 以MHC ⅠαQF1和MHC ⅠαQR1为引物, 应用qPCR技术检测MHC Ⅰα基因在大弹涂鱼健康组织中的表达。结果表明(图 4), MHC Iα基因mRNA在不同组织中均有表达, 其中肾和脾组织中表达量最高, 在鳃和肠组织中表达次之。
|
图 4 大弹涂鱼MHC Ⅰα基因在健康组织中的表达情况 各组织的表达情况通过RT-PCR检测, β-actin用作内参, 为了便于比较, 将大脑中的表达水平设为1. Fig.4 Tissue distribution of MHC Ⅰα gene of Boleophthalmus pectinirostris under normal physiological condition Expression levels of MHC Ⅰα in various tissues were measured by RT-PCR. β-actin was used as the internal control to calibrate the cDNA templates. For the convenience of comparison, the expression level in brain was set as 1. |
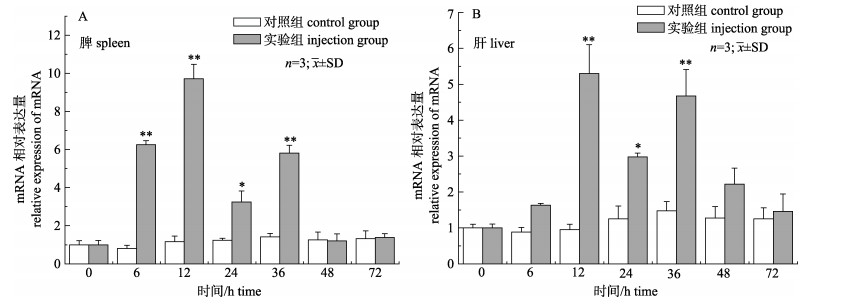
RT-PCR分析腹腔注射poly(I:C)后的大弹涂鱼MHC Ⅰα基因mRNA表达变化, 结果显示(图 5), 在肝组织中, 对照组MHC Ⅰα基因mRNA表达量在所有时间点内均没有显著变化(P > 0.05);而实验组的表达量具有显著变化, 在6 h时, MHC Iα基因mRNA表达量开始上升(1.63倍); 在12 h时表达量显著上升, 达到最大值(P < 0.01, 为5.30倍); 在24 h时, 表达量有所下降, 但仍然显著高于对照组的表达量(P < 0.05, 为2.98倍); 在36 h时, 表达量又一次升高, 仍然显著高于对照组(P < 0.01, 为4.68倍); 以后, 表达量逐渐下降到48 h时的2.22倍和72 h的1.46倍。在脾组织中, 对照组MHC Ⅰα基因mRNA表达量在所有时间点内同样没有显著变化(P > 0.05);而实验组的表达量具有显著变化, 但峰值的出现时间早于肝组织, 即在6 h时, 实验组MHC Ⅰα基因mRNA表达量显著上升(P < 0.01, 为6.25倍); 在12 h时, 实验组的表达量更是显著上升达到最大值(P < 0.01), 为9.71倍在24 h时, 表达量也有下降, 但仍然高于对照组(P < 0.05, 为3.25倍); 在36 h时, 表达量又一次升高, 且显著高于对照组(P < 0.01, 为5.81倍); 其表达量在48 h和72 h逐渐恢复到正常水平(分别为1.21倍和1.38倍)。
|
图 5 大弹涂鱼肝和脾中MHC Ⅰα基因在poly(I:C)刺激下的表达 β-actin用作内参. *表示P < 0.05, **表示P < 0.01. Fig.5 The hepatic and splenic responses of Boleophthalmus pectinirostris MHC Ⅰα under poly(I:C) challenge β-actin was used as the internal control. *means P < 0.05; ** means P < 0.01. |
MHC I分子在脊椎动物免疫系统中起重要作用, 它主要识别细胞内抗原, 结合细胞内源肽并呈递到CD8+ T细胞, 从而引发适应性免疫[6, 12]。迄今为止, 许多研究报道了不同鱼类的MHC Ⅰα基因的鉴定和表达[8-9, 11], 然而大弹涂鱼MHC Ⅰα基因的表征仍不清楚。本研究首次分析了大弹涂鱼MHC Ⅰα基因的氨基酸序列, 发现只有1个N-糖基化位点, 一些鱼类被预测有一个单一的糖基化位点, 如牙鲆(Paralichthys olivaceus)[19]、卵形鲳鯵(Trachinotus ovatus)[20]、舌齿鲈(Dicentrarchus labrax)[21], 而刺鱼(Gasterosteus aculeatus)有3个糖基化位点[22], 条纹斑竹鲨(Chiloscyllium plagiosum)则无糖基化位点[23]。蛋白质二级结构分析表明, 大弹涂鱼MHC Ⅰα分子的α螺旋、β折叠结构规则, 形态固定, 处于蛋白质内部, 而β转角和无规则卷曲处于蛋白质的表面, 结构突出, 有利于与抗体结合, 因此可能作为抗原结合部位。
在现存物种的漫长进化过程中, MHC基因家族大约出现在5亿年前的低等脊椎动物中, 并在那时开始分化[4]。本研究构建了系统发生树分析大弹涂鱼和其他已鉴定鱼类MHC Ⅰα基因的进化关系, 由进化树可以看出, 大弹涂鱼与河川沙塘鳢(Odontobutis potamophila)的MHC Ⅰα基因分子进化距离最近, 亲缘关系最密切, 这与两者同属虾虎鱼亚目(Gobioidei)的传统分类学相符。
MHC基因的组织特异性表达已在许多硬骨鱼中报道, 并广泛分布于许多鱼类的各种组织中[8-11], 本研究评估了大弹涂鱼MHC Iα基因mRNA在不同组织中的表达情况, 结果表明, MHC Ⅰα基因mRNA在肾和脾组织中表达量最高, 鳃和肠组织中表达次之, 在肝、脑、皮肤和肌肉中表达量较低。这个结果与虹鳟(Oncorhynchus mykiss)中MHC Ⅰα基因表达结果相似, 在肠、肾、胸腺和脾表达量较高, 脑、肝中表达量较低[10], 在牙鲆(Paralichthys olivaceus)中, MHC Ⅰα基因在鳃、脑、肾、肠、脾中的表达量也较高, 在肌肉、卵巢和皮肤中表达较低[19]。众所周知, 脾和肾是鱼类的主要免疫器官[12, 20], 而肠道和鳃由于经常暴露在外界环境中, 可以接触到许多病原体[24], MHC Ⅰα基因在这些组织中的高表达, 可能表明这些组织都参与了大弹涂鱼的免疫应答。
鱼类养殖环境受到许多自然因素和人为因素的影响, 自然养殖水域的盐度经常会发生变化, 而盐度的变化会导致鱼类代谢加速、耗氧率升高、能量需求增加, 严重时会造成生理机能的失常[13]。有学者认为, 在盐度胁迫条件下, 鱼类会通过提升免疫功能来做出相应的防御机制, 例如, IgM (immunoglobulin M)是由B淋巴细胞产生的一类能特异识别并中和相应抗原的、具有免疫作用的球蛋白, 将尼罗罗非鱼(Oreochromis niloticus)从淡水转移至24‰盐度养殖一段时间后, IgM含量显著上升[25]; 补体系统是先天免疫的重要组分, 研究发现分别在盐度6、12和38的水体下养殖金头鲷(Sparus aurata ) 3个月, 盐度38组的补体旁路活性最高, 并与盐度6组的形成显著差异[26]。poly(I:C)是dsRNA的人工模拟物, 本研究以在高盐度水体下腹腔注射poly(I:C)的形式模拟高盐胁迫后大弹涂鱼被病毒感染后的情况, 结果表明, 大弹涂鱼腹腔注射poly(I:C)后, 肝和脾组织中MHC Ⅰα基因mRNA表达显著上升, 在12 h时, mRNA表达量达到最大值, 且脾组织的峰值出现时间早于并大于肝组织, 这与鮸(Miichthys miiuy)、大黄鱼(Pseudosciana crocea)等硬骨鱼类在病毒感染后的表达相一致[27-28]。这可能证明在高盐胁迫下, MHC Ⅰα基因仍然参与了大弹涂鱼关于病毒感染的免疫应答, 且脾组织的免疫应答速度比肝组织更迅猛, 这也为研究高盐水体环境下病毒感染对大弹涂鱼免疫应答机制的影响提供理论借鉴。
4 结论本研究首次分析了大弹涂鱼MHC Ⅰα基因的序列特征和与其他已鉴定鱼类MHC Ⅰα基因的进化关系, 初步评估了大弹涂鱼MHC Ⅰα基因mRNA在不同组织中的表达情况以及在高盐胁迫下注射poly(I:C)后对其表达的影响, 为进一步了解环境刺激对虾虎鱼类和其他硬骨鱼类免疫应答机制的影响奠定基础。
| [1] |
Chen S X, Hong W S, Zhang Q Y, et al. Rate of oxygen consumption and tolerance of hypoxia and desiccation in mudskipper (Boleophthalmus pectinirostris) and Chinese black sleeper (Bostrichthys sinensis) embryos[J]. Acta Oceanologica Sinica, 2006, 25(4): 91-98. |
| [2] |
Chen S X, Hong W S, Su Y Q, et al. Microhabitat selection in the early juvenile mudskipper Boleophthalmus pectinirostris (L.)[J]. Journal of Fish Biology, 2008, 72(3): 585-593. DOI:10.1111/jfb.2008.72.issue-3 |
| [3] |
Bernatchez L, Landry C. MHC studies in nonmodel vertebrates: what have we learned about natural selection in 15 years?[J]. Journal of Evolutionary Biology, 2003, 16(3): 363-377. DOI:10.1046/j.1420-9101.2003.00531.x |
| [4] |
Rodgers J R, Cook R G. MHC class Ⅰb molecules bridge innate and acquired immunity[J]. Nature Reviews Immunology, 2005, 5(6): 459-471. DOI:10.1038/nri1635 |
| [5] |
Hughes A L, Yeager M. Natural selection at major histocompatibility complex loci of vertebrates[J]. Annual Review of Genetics, 1998, 32(1): 415-435. DOI:10.1146/annurev.genet.32.1.415 |
| [6] |
K nig R, Huang L Y, Germain R N. MHC class Ⅱ interaction with CD4 mediated by a region analogous to the MHC class Ⅰ binding site for CD8[J]. Nature, 1992, 356(6372): 796-798. DOI:10.1038/356796a0 |
| [7] |
Ohta Y, Okamura K, McKinney E C, et al. Primitive synteny of vertebrate major histocompatibility complex class Ⅰ and class Ⅱ genes[J]. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(9): 4712-4717. DOI:10.1073/pnas.97.9.4712 |
| [8] |
Bingulac-Popovic J, Figueroa F, Sato A, et al. Mapping of mhc class Ⅰ and class Ⅱ regions to different linkage groups in the zebrafish, Danio rerio[J]. Immunogenetics, 1997, 46(2): 129-134. DOI:10.1007/s002510050251 |
| [9] |
Wang D Q, Zhong L, Wei Q W, et al. Evolution of MHC class Ⅰ genes in two ancient fish, paddlefish (Polyodon spathula) and Chinese sturgeon (Acipenser sinensis)[J]. FEBS Letters, 2010, 584(15): 3331-3339. DOI:10.1016/j.febslet.2010.05.065 |
| [10] |
Hansen J D, La Patra S. Induction of the rainbow trout MHC class Ⅰ pathway during acute IHNV infection[J]. Immunogenetics, 2002, 54(9): 654-661. DOI:10.1007/s00251-002-0509-x |
| [11] |
Grimholt U, Olsaker I, de Vries Lindstr m C, et al. A study of variability in the MHC class Ⅱ β1 and class Ⅰ α2 domain exons of Atlantic salmon, Salmo salar L.[J]. Animal Genetics, 1994, 25(3): 147-153. |
| [12] |
Terado T, Okamura K, Ohta Y, et al. Molecular cloning of C4 gene and identification of the class Ⅲ complement region in the shark MHC[J]. The Journal of Immunology, 2003, 171(5): 2461-2466. DOI:10.4049/jimmunol.171.5.2461 |
| [13] |
Livingstone D R. Contaminant-stimulated reactive oxygen species production and oxidative damage in aquatic organisms[J]. Marine Pollution Bulletin, 2001, 42(8): 656-666. DOI:10.1016/S0025-326X(01)00060-1 |
| [14] |
Brown J A, Moore W M, Quabius E S. Physiological effects of saline waters on zander[J]. Journal of Fish Biology, 2001, 59(6): 1544-1555. DOI:10.1111/jfb.2001.59.issue-6 |
| [15] |
Nolan D T, Op't Veld R L J M, Balm P H M, et al. Ambient salinity modulates the response of the tilapia, Oreochromis mossambicus (Peters), to net confinement[J]. Aquaculture, 1999, 177(1-4): 297-309. DOI:10.1016/S0044-8486(99)00093-9 |
| [16] |
Yin F, Peng S M, Sun P, et al. Effects of low salinity on antioxidant enzymes activities in kidney and muscle of juvenile silver pomfret Pampus argenteus[J]. Acta Ecologica Sinica, 2011, 31(1): 55-60. DOI:10.1016/j.chnaes.2010.11.009 |
| [17] |
Rubino J G, Zimmer A M, Wood C M. Intestinal ammonia transport in freshwater and seawater acclimated rainbow trout (Oncorhynchus mykiss): evidence for a Na+ coupled uptake mechanism[J]. Comparative Biochemistry and Physiology Part A: Molecular & Integrative Physiology, 2015, 183: 45-56. |
| [18] |
Tamura K, Peterson D, Peterson N, et al. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10): 2731-2739. DOI:10.1093/molbev/msr121 |
| [19] |
Srisapoome P, Ohira T, Hirono I, et al. Cloning, characterization and expression of cDNA containing major histocompatibility complex class Ⅰ, Ⅱα and Ⅱβ genes of Japanese flounder Paralichthys olivaceus[J]. Fisheries Science, 2004, 70(2): 264-276. DOI:10.1111/fis.2004.70.issue-2 |
| [20] |
Zhang X H, Dai Z X, Zhang G H, et al. Molecular characterization, balancing selection, and genomic organization of the tree shrew (Tupaia belangeri) MHC class Ⅰ gene[J]. Gene, 2013, 522(2): 147-155. DOI:10.1016/j.gene.2013.03.113 |
| [21] |
Pinto R D, Randelli E, Buonocore F, et al. Molecular cloning and characterization of sea bass (Dicentrarchus labrax, L.) MHC class Ⅰ heavy chain and β2-microglobulin[J]. Developmental & Comparative Immunology, 2013, 39(3): 234-254. |
| [22] |
Scharsack J P, Kalbe M, Schaschl H. Characterization of antisera raised against stickleback (Gasterosteus aculeatus) MHC class Ⅰ and class Ⅱ molecules[J]. Fish & Shellfish Immunology, 2007, 23(5): 991-1002. |
| [23] |
Shen T, Lei M L, Wang J R, et al. Molecular cloning, organization, expression and 3D structural analysis of the MHC class Ⅰa gene in the whitespotted bamboo shark (Chiloscyllium plagiosum)[J]. Veterinary Immunology and Immunopathology, 2014, 157(1-2): 111-118. DOI:10.1016/j.vetimm.2013.10.012 |
| [24] |
Pang J C, Gao F Y, Lu M X, et al. Major histocompatibility complex class ⅡA and ⅡB genes of Nile tilapia Oreochromis niloticus: Genomic structure, molecular polymorphism and expression patterns[J]. Fish & Shellfish Immunology, 2013, 34(2): 486-496. |
| [25] |
Dominguez M, Takemura A, Tsuchiya M, et al. Impact of different environmental factors on the circulating immunoglobulin levels in the Nile tilapia, Oreochromis niloticus[J]. Aquaculture, 2004, 241(1-4): 491-500. DOI:10.1016/j.aquaculture.2004.06.027 |
| [26] |
Cuesta A, Laiz-Carrión R, del Río M P M, et al. Salinity influences the humoral immune parameters of gilthead seabream (Sparus aurata L.)[J]. Fish & Shellfish Immunology, 2005, 18(3): 255-261. |
| [27] |
Liu G Z, Zheng W B, Chen X H. Molecular cloning of proteasome activator PA28-β subunit of large yellow croaker (Pseudosciana crocea) and its coordinated up-regulation with MHC class Ⅰ α-chain and β2-microglobulin in poly I:C-treated fish[J]. Molecular Immunology, 2007, 44(6): 1190-1197. DOI:10.1016/j.molimm.2006.06.024 |
| [28] |
Xu T J, Sun Y N, Cheng Y Z, et al. Genomic sequences comparison and differential expression of miiuy croaker MHC class Ⅰ gene, after infection by Vibrio anguillarum[J]. Developmental & Comparative Immunology, 2011, 35(4): 483-489. |
 2019, Vol. 26
2019, Vol. 26

