翘嘴鳜(Siniperca chuatsi)是中国特有的重要经济鱼类, 分布于中国除青藏高原外的各大水系, 属于鲈形目(Perciformes), 鮨科(Serranidae), 鳜属。翘嘴鳜具有生长速度快、肉质好、个体大的特点, 是重要的养殖品种[1]。与此同时, 与翘嘴鳜生长、应激相关基因的克隆和表达也受到关注[2-3]。Wang等[4]从GH (growth hormone)、IGF (Insulin-like growth factor)基因上筛选出6个SNP (single nucleotide polymorphisms)位点, 并验证其与翘嘴鳜生长具有显著的相关性。Zhu等[5]通过饥饿再投喂实验对翘嘴鳜肌肉进行了microRNA分析, 并且筛选出4个在急性肌肉萎缩刺激条件下对肌肉中基因转录、翻译有重要作用的miRNAs位点。了解不同生长性状翘嘴鳜的分子表达模式差异, 可以帮助揭示翘嘴鳜生长调控网络, 筛选分子育种标记, 改良翘嘴鳜品质。
转录组测序可以帮助分析特定组织的基因表达情况, 寻找生物调控网络和机理。通过转录组测序可获得大量基因表达信息, 用于比较不同状态下基因表达量的差异, 获取与目的性状相关的基因表达情况, 从而了解调控目的性状的基因网络。随着测序技术的逐渐成熟, 转录组测序被应用于免疫、性别、生长、应激等多种表型相关的关键基因调控网络研究和重要性状分子标记鉴定, 水产动物中转录组测序研究也越来越多[6]。Wang等[7]用转录组测序技术筛选了抗肠道败血病候选基因和疾病相关表达差异显著的单核苷酸多态性位点; Sun等[8]采用Illumina测序技术筛选斑点叉尾鮰(Ietalurus punetaus)性别相关基因, 并通过进一步的分析发现许多涉及性腺发生、精子发生、性别分化的基因; Jeukens等[9]利用RNA-seq技术研究了短小型和正常体型白鲑(Salmonidae)的适应性转录组变化, 发现白鲑短小型高表达基因主要与免疫、DNA复制以及能量代谢有关, 而正常体型则与蛋白质合成有关。
鱼类的生长伴随鱼类整个生活周期, 受到基因和环境等多方面因素的共同影响, 由于其调控机制非常复杂, 受到了研究者广泛的关注。而转录组测序可以在没有研究物种基因信息的情况下进行分析, 对其功能基因组进行深入研究。本研究首次使用转录组测序对体重差异翘嘴鳜进行差异基因分析, 为翘嘴鳜生长相关基因的研究提供了完整的基因表达背景。
本研究根据全同胞家系翘嘴鳜出现体型显著差异现象, 通过对体型差异翘嘴鳜个体进行肌肉转录组测序, 对差异表达基因进行注释和通路富集分析, 研究同一家系在相同养殖条件下, 极大个体(Overweight)和极小个体(Underweight)翘嘴鳜肌肉基因表达模式差异, 了解大、小个体间差异基因表达特点, 为深入开发翘嘴鳜生长相关分子标记提供科学依据。
1 材料与方法 1.1 实验材料本研究中翘嘴鳜亲鱼来自于佛山市南海区百容水产良种股份有限公司, 经人工授精繁殖后, 获得翘嘴鳜全同胞家系。每天饱食投喂, 养殖水温在26~28℃, pH变化范围在6.5~7.0, 溶氧保持在6 mg/L以上。养殖3个月后, 对所有个体进行称重、测量, 从中选择体型极大个体和极小个体各5尾, 禁食24 h, 使用MS-222将实验鱼麻醉后快速对肌肉组织进行取样。
1.2 RNA提取方法将样本在液氮中充分研磨, 按照E.Z.N.A.® Total RNA Kit Ⅱ试剂盒操作规范进行RNA提取, 使用DNase I对提取到的总RNA消化1 h, 去除DNA残留。分别取1 μL RNA样品用核酸蛋白测定仪和Agilent2100对RNA样品的纯度、质量和浓度进行检测, 同时取800 ng RNA于1%琼脂糖凝胶进行电泳检测RNA样品完整性。
1.3 RNA逆转录及建库分别将翘嘴鳜极大个体和极小个体各5尾的肌肉组织RNA等量混合, 得到两个RNA pools, 用Oligo(dT)磁珠富集纯化mRNA, 以片段mRNA为模板, 逆转录酶和六碱基随机引物合成cDNA第一链, 再用RNaseH和DNA聚合酶合成第二链。使用T4 DNA聚合酶、Klenow DNA聚合酶和T4多聚核苷酸激酶修复DNA末端, T4 DNA连接酶连接测序接头, 通过纯化选择200 bp+25 bp的cDNA片段扩增得到DNA文库。采用双端测序(paired-end)技术, 将构建好的两个测序文库用Illumina HiSeqTM2000进行测序, 获得900 bp左右的双端测序的片段(paired-end reads)。
1.4 测序数据分析测序得到的原始图像数据经碱基识别(base calling)转化为序列数据称之为原始数据(raw data)或原始片段(raw reads), 经过对raw reads序列进行筛选, 去除含adaptor、N比例大于5%及低质量reads(质量值Q≤20的碱基数占整个read的50%以上), 得到可供后续分析的过滤后片段(clean reads)。使用短reads组装软件Trinity做转录组从头组装, 使用序列聚类软件TGICL做进一步序列拼接和去冗余处理, 得到不含N的非重复序列基因(Unigene)。用SOAPaligner/soap2将clean reads比对到参考基因组和参考基因序列中进行基因注释。
1.5 基因功能注释及分类将Unigene序列与蛋白数据库Nr (nonredundant protein)、Swiss-Prot、KEGG (Kyoto Encyclopedia of Genes and Genomes)和COG (Clusters of Orthologous Groups)做Blastx比对(evalue < 0.00001), 取比对结果最好的蛋白确定Unigene的序列方向。根据Nr注释信息, 通过Blast2GO软件对得到的Unigene进行GO(Gene Ontology)富集。用WEGO软件对所有Unigene做GO分类统计, 将Unigene描述为分子功能、细胞组分、参与的生物过程3个功能分类, 认识该物种的基因功能分布特征。
1.6 差异基因表达分析使用RPKM法(Reads Per Kb per Million reads)计算基因表达量。RPKM法可以消除基因长度和测序量差异对计算基因表达的影响, 计算得到的基因表达量可直接用于比较不同样品间的基因表达差异。对差异检验的P值作多重假设检验校正, 通过控制FDR(False Discovery Rate)来决定P值的域值。FDR值越小, 差异倍数越大, 表明表达差异越显著。差异表达基因评判标准为FDR≤0.001且倍数差异不低于2倍的基因。分别用WEGO软件对差异表达基因进行GO功能分类统计和用KEGG数据库对差异表达基因进行Pathway注释。
1.7 差异基因荧光定量PCR(Real-time PCR)验证提取肌肉中RNA进行逆转录, 以肌肉cDNA为模板, 对转录组结果中6个差异表达显著的部分基因进行荧光定量PCR验证, 包括生长轴的生长激素受体(GHR2)和胰岛素样生长因子受体(IGFR1)、肌肉纤维组成蛋白(Mhc)、涉及蛋白和折叠和运输的热休克蛋白Hsp90(heat shock 90)、涉及蛋白质生成的4ebp、肌肉纤维调节蛋白(MyoD)。以18S、β-actin为内参基因, 通过RT-PCR(realtime fluores-cence quantitative PCR)检测不同基因的表达水平。
2 结果与分析 2.1 体重差异翘嘴鳜的选择本实验在相同养殖条件下, 从同一家系的60尾健康翘嘴鳜个体中挑选极大个体(Overweight)、极小个体(Underweight)各5尾, 极大个体组平均体重(60.2±5.7) g; 极小个体组平均体重(13.2±1.4) g, 极大个体组平均体重可达极小个体组4倍, 体长、体高也分别达到极小个体组两倍, 具有明显的体型差异(表 1)。
|
|
表 1 翘嘴鳜极大个体和极小个体生长性状 Tab.1 Growth traits of overweight and underweight Siniperca chuatsi |
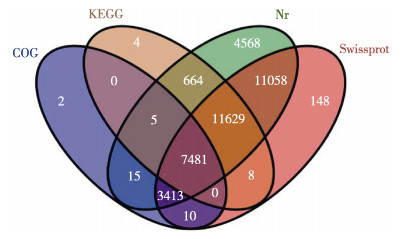
经Illumina测序后, 两个翘嘴鳜转录组样本共获得95581082条raw reads, 其中极大个体组48963598条, 极小个体组46617484条; 经过筛选去除低质量序列后, 共得到91264026条clean reads, 平均长度约为100 bp, 占总reads数的95.48%;测序结果中GC含量为51.31%, Q20百分比为98.57%, 具有良好的测序质量, 两个转录组本间无显著数据量差异, 可用于分析两个转录本表达模式差异。经Trinity拼接组装后, 两个转录本共得到73353条Unigene, 平均长度为703 bp(表 2)。在去除短序列和重复序列之后, 经过Nr、Swiss-Prot、KEGG和COG4个数据库共定义39005条Unigene。Nr、COG、Swissprot及KEGG分别定义了38833条、10926条、33749条和19791条Unigene, 确定Unigene序列方向后, 通过定义最多Unigene数量的Nr数据库进行注释(图 1)。
|
|
表 2 翘嘴鳜极大个体和极小个体转录组测序数据产出及拼接结果汇总 Tab.2 The brief information of deep sequencing and assembly for overweight and underweight Siniperca chuatsi |
|
图 1 Nr、COG、KEGG、Swissprot数据库比对结果 Fig.1 Detection of homologous genes in public databases (Nr, COG, KEGG, Swissprot) |
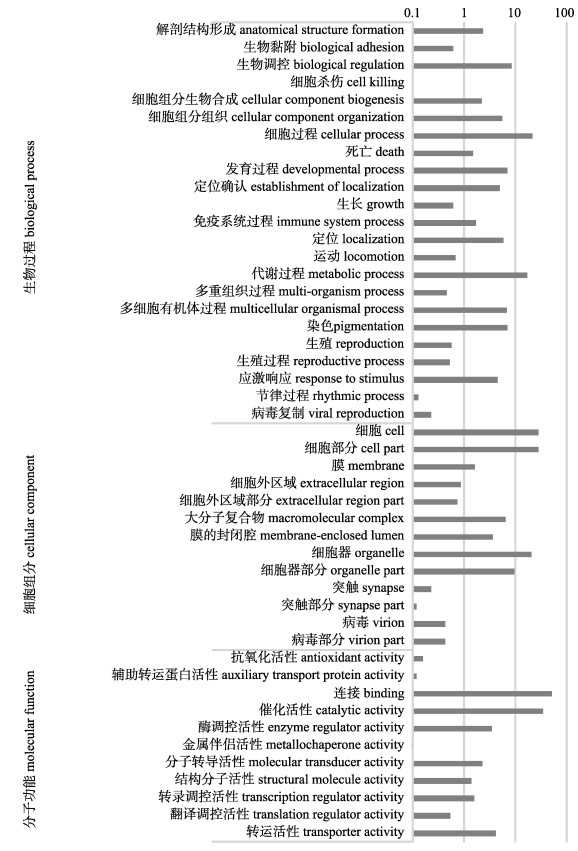
计算RPKM的大小衡量基因表达水平的高低, 经过RPKM标准化计算处理后, 共获得8942个差异表达基因, 其中7858个Unigene在极小个体中表达量较高。GO富集将5150个差异表达基因富集到3个分集, 分别是生物过程1530条、细胞组分1737条和分子功能1883条。
在生物过程分集中, 细胞(cell)和细胞成分(cell part)是主要的功能分组; 分子功能分集中, 连接(binding)是主要功能分组; 细胞代谢过程(cellular process)是生物过程分集中的主要功能分组(图 2)。
|
图 2 翘嘴鳜极大个体和极小个体转录组测序差异表达基因GO数据库比对结果 横轴表示GO注释的基因数目以10为底数的对数. Fig.2 The GO analysis of different expression genes between overweight and underweight Siniperca chuatsi The lateral axis indicates the number of genes annotated by GO after log transformation. |
通过翘嘴鳜转录组数据分析得到了大量的差异表达基因, 其中大部分基因在极小个体组表达量更高, 如GHR2 (growth hormone receptor 2)、IGFR1 (type 1 insulin-like growth factor receptor)等(表 3)。通过KEGG分析, 以P < 0.05判定为有显著意义, 共获得25个通路。富集差异表达基因最多的通路是内质网蛋白加工(protein processing in endoplasmic reticulum)、泛素调节的蛋白质降解(ubiquitin mediated proteolysis)以及RNA转运(RNA transport), 分别是119条(3.77%)、101条(3.2%)和91条(2.88%)差异表达基因(表 4)。
|
|
表 3 翘嘴鳜极大个体和极小个体差异表达基因 Tab.3 The different expression genes between underweight and overweight Siniperca chuatsi |
|
|
表 4 翘嘴鳜极大个体和极小个体差异表达基因通路分析 Tab.4 The pathway analysis of different expression genes between underweight and overweight Siniperca chuatsi |
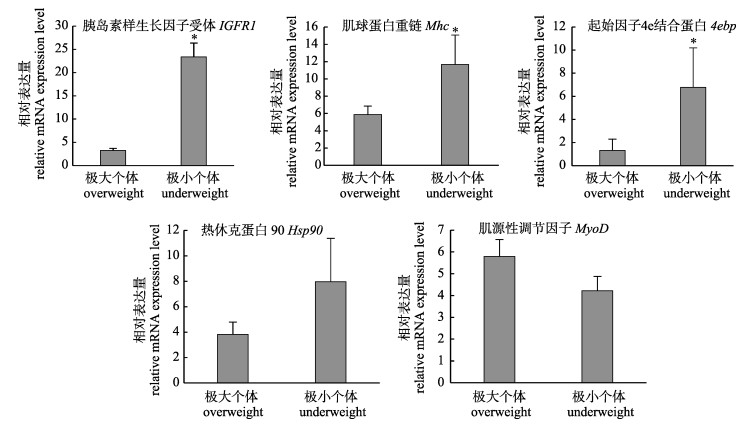
RT-PCR扩增产物结果显示, 熔解曲线单一; 实验有限循环值在15~30之间, 且内参基因18S的Ct比其他待测基因Ct值小, β-actin在不同个体间表达量稳定。通过RT-PCR, 基因表达差异趋势与转录组结果相同(图 3)。
|
图 3 翘嘴鳜极大个体和极小个体差异表达基因荧光定量PCR验证 y轴表示相对表达量, 误差线表示标准差, *表示显著差异(P < 0.05). Fig.3 The RT-PCR analysis of gene expression in overweight and underweight Siniperca chuatsi The y-axis indicates relative expression level. Error bars mean standard deviation. * indicate the differences between the two groups are significant (P < 0.05). |
鱼类的生长伴随于鱼类整个生活周期, 与基因、环境等多方面因素有关, 在体内受到生长与生殖调控轴相互作用的影响[10]。过去的研究中发现, 许多鱼类中都存在不同家系个体具有显著生长差异的特性, 甚至来自同一家系的个体也会出现明显生长差异的现象, 具有显著生长差异的个体成为研究生长差异调控机制的重要对象[11-13]。Kocmarek等[13]对同一品系虹鳟在不同产卵期的大个体和小个体肌肉和肝脏分别进行mRNA微阵列分析, 评估不同季节(晚秋vs春初)和体型(大vs小)间转录组水平差异。王博等[14]通过对生长存在显著差异的翘嘴鳜个体进行肌肉microRNA转录组测序, 发现miR-122、miR-192、miR-451、let-7j-5p共4个表达显著差异的miRNA, 差异表达miRNAs参与生长、发育、代谢等重要途径。与其他鱼类类似, 本研究发现, 来自同一家系的翘嘴鳜在相同养殖条件下生长至3月龄, 出现明显的生长差异现象。通过挑选出极大个体和极小个体各5尾, 极大个体组体重可达极小个体组4倍以上, 体长、提高均可达其2倍, 具有明显的体形差异。
在活鱼体内, 鱼体体重的30%~80%为骨骼肌重量, 是鱼类经济价值的主要体现。鱼类出生后肌肉的生长主要通过纤维细胞的增殖(hyperplasia)和肥大(hypertrophy)达到肌纤维数量、直径和长度的增加, 最终达到生长的目的, 而这一过程受到一系列基因在转录水平上的综合调控[15]。生长因子、调控蛋白、转录因子都被证实参与了鱼类肌肉形成的调控过程[16]。本研究通过对极大个体和极小个体肌肉组织进行转录组测序, 共获得8942个差异表达基因, 其中大部分差异表达基因在极小个体中表达量增加。极小个体组包含更多的表达量上调的基因说明极小个体组具有更高的基因表达活性。相似的结果在其他研究中也观察到。Kocmarek等[13]对不同大小的虹鳟微阵列分析结果显示, 无论在春季或秋季, 小鱼组肝脏和肌肉中包含更多的表达上调的基因, 表达量上调基因涉及蛋白转录、翻译以及细胞凋亡, 造成了小鱼肌肉中较高的细胞周转率, 从而导致体型差异。在具有不同生长率的珍珠贝中, 通过分别对大小个体进行转录组分析发现, 小个体组具有更多表达量上调的基因, 包括与贝壳形成、能量代谢、肌肉生成等有关的基因, 具有小个体追赶生长的趋势[17]。
本实验中发现大量在极大个体和极小个体间表达量差异显著的基因, GH-IGF通路受体基因GHR2、IGFR1, GH-IGF通路下游基因4ebp (initiation factor 4E-binding protein 1)、转录延长因子, 肌肉纤维组成基因Mhc (myosin heavy chain)、Mlc (myosin light chain 1)、Troponin等在极小个体的翘嘴鳜中表达量升高, 但MyoD1在极大个体和极小个体间表达水平并无显著差异。通过对差异表达基因进行KEGG富集通路分析结果显示, 大量的基因富集在与蛋白生成、消化有关的通路, 与差异表达基因分析趋势一致。GH-IGF是硬骨鱼中重要的生长调控通路, GH促进肝脏分泌IGFs, 与IGFR结合, 通过PI3K/AKT和ERK/MAPK两条通路调控生长, 前者促进蛋白生成, 后者促进细胞增生及增殖[18]。PI3K通路磷酸化, 调节下游分子4ebp, 进而参与调节转录延长因子、转录起始因子以调控生长。通过在牙鲆饥饿再投喂实验中发现, 硬骨鱼PI3K通路和ERK/MAPK通路在肌肉生长中具有重要作用[19]。然而GHR、IGFR表达量升高与生长率并非正相关, 4月龄的虹鳟用GH处理后出现生长抑制, 而其GHR1和GHR2表达量则升高; 虹鳟、翘嘴鳜等鱼类在经过饥饿后, GHR2、IGFR1表达量升高。与本研究结果一致, 在受到饥饿或生长抑制时, 虽然生长率与GHR2、IGFR1基因的表达成反比趋势, 可能参与了不利条件下肌肉能量储存代谢通路的激活[20], 同时有观点认为GHR2可能促进了tGHR (truncted GH receptor)的生成, 而tGHR诱导了某些分解反应, 从而限制了GH信号通路[21], 小个体翘嘴鳜中, GHR2表达量升高符合GH信号通路被抑制的趋势。Mhc和Mlc是两种重要的组成肌肉纤维的蛋白链, 肌钙蛋白(Troponin)是间质细胞决定分化为肌细胞后, 所表达的肌肉特异性蛋白, 参与调节肌肉收缩和舒张, Troponin表达量增加利于增强肌肉收缩, 进而通过促进肌纤维的合成[22]。在本研究中, Troponin在小个体中具有更高的表达量。与凡纳滨对虾研究结果一致, 李喜莲通过对不同个体凡纳滨对虾肌肉中表达基因进行抑制性消减杂交发现, Troponin在小个体中的表达量明显高于大个体[23]。
肌肉中大量组成成分是蛋白质, 鱼类的生长与蛋白合成及消化紧密相关。KEGG聚类结果显示, 富集差异表达基因最多的通路是内质网蛋白加工(protein processing in endoplasmic reticulum)、泛素调节的蛋白质降解(ubiquitin mediated proteolysis)以及RNA转运(RNA transport), 分别是119条(3.77%)、101条(3.2%)和91条(2.88%)差异表达基因(表 4)。RNA转运是细胞基因表达的基础, 而内质网蛋白质加工则帮助蛋白质的正确折叠, 并对错误折叠的蛋白质进行降解, 是蛋白生成的基础。与差异表达基因趋势一致, 在小个体翘嘴鳜中大部分基因表达量更高, 具有更活跃的表达模式。泛素调节的蛋白质降解通路是蛋白质降解的重要途径, 通过与酶结合后产生级联反应达到蛋白质降解的目的[24]。本研究中小个体翘嘴鳜蛋白质降解通路表达量增加, 蛋白质积累减少, 具有更低的生长率。其他鱼类研究显示, 智利羽鼬鳚(Cenypterus chilensrs)在应激条件下, 泛素调节的蛋白质降解具有更高的基因表达, 也证实了在应激条件下, 鱼类的低生长率与蛋白质合成代谢有关[25]。
4 结论综上所述, 来自同一家系翘嘴鳜个体在相同养殖条件下仍然产生明显的体型差异, 通过对极大个体、极小个体肌肉组织进行转录组测序, 比较基因表达差异发现, GHR2、IGFR1、4ebp、Mhc、Mlc、Troponin等多个基因在极小个体中表达量增加, 而大量差异表达基因富集在蛋白生成和消化相关通路, 可能参与了翘嘴鳜生长调控。本实验通过转录组测序获得了大量翘嘴鳜转录本信息, 研究结果为翘嘴鳜更深入的基因调控网络研究提供丰富的基因资源。
| [1] |
Li M F. Research progress on biology of mandarin fish[J]. Modern Fisheries Information, 2010, 25(7): 16-21. [李明锋. 鳜鱼生物学研究进展[J]. 现代渔业信息, 2010, 25(7): 16-21. DOI:10.3969/j.issn.1004-8340.2010.07.005] |
| [2] |
Wang P F, Zeng S, Xu P, et al. Two HSP90 genes in mandarin fish Siniperca chuatsi:Identification, characterization and their specific expression profiles during embryogenesis and under stresses[J]. Fish Physiology and Biochemistry, 2016, 42(4): 1123-1136. DOI:10.1007/s10695-016-0202-x |
| [3] |
Lu X, Gu Y M, Hou X C, et al. Molecular characterization, tissue distribution, and expression regulation from fasting and re-feeding of two growth hormone receptors in mandarin fish Siniperca chuatsi[J]. Fisheries Science, 2016, 82(1): 155-169. DOI:10.1007/s12562-015-0941-0 |
| [4] |
Wang H F, Sun J J, Lu X, et al. Identification of insulin-like growth factor I gene polymorphisms using high-resolution melting and its effect on growth traits in sinipercid species[J]. Fisheries Science, 2013, 79(3): 439-446. DOI:10.1007/s12562-013-0607-8 |
| [5] |
Zhu X, Li Y L, Chen D X, et al. Selection of reference genes for MicroRNA quantitative expression analysis in Chinese perch, Siniperca chuatsi[J]. International Journal of Molecular Sciences, 2015, 16(12): 8310-8323. DOI:10.3390/ijms16048310 |
| [6] |
Wang Z, Gerstein M, Snyder M. RNA-Seq:a revolutionary tool for transcriptomics[J]. Nature Reviews Genetics, 2009, 10(1): 57-63. DOI:10.1038/nrg2484 |
| [7] |
Wang R J, Sun L Y, Bao L S, et al. Bulk segregant RNA-seq reveals expression and positional candidate genes and allele-specific expression for disease resistance against enteric septicemia of catfish[J]. BMC Genomics, 2013, 14: 929. DOI:10.1186/1471-2164-14-929 |
| [8] |
Sun F Y, Liu S K, Gao X Y, et al. Male-biased genes in catfish as revealed by RNA-Seq analysis of the testis transcriptome[J]. PLoS ONE, 2013, 8(7): e68452. DOI:10.1371/journal.pone.0068452 |
| [9] |
Jeukens J, Renaut S, St-Cyr J, et al. The transcriptomics of sympatric dwarf and normal lake whitefish (Coregonus clupeaformis spp., Salmonidae) divergence as revealed by next-generation sequencing[J]. Molecular Ecology, 2010, 19(24): 5389-5403. DOI:10.1111/j.1365-294X.2010.04934.x |
| [10] |
Dutta H. Growth in fishes[J]. Gerontology, 1994, 40(2-4): 97-112. DOI:10.1159/000213581 |
| [11] |
Liu P P, Chen X H, Zhong L Q, et al. Comparison of growth performance of different families of yellow catfish (Pelteobagrus fulvidraco)[J]. Journal of Nanjing Normal University (Natural Science Edition), 2013, 36(1): 90-93. [刘朋朋, 陈校辉, 钟立强, 等. 黄颡鱼不同家系生长性能的比较[J]. 南京师大学报(自然科学版), 2013, 36(1): 90-93. DOI:10.3969/j.issn.1001-4616.2013.01.016] |
| [12] |
Wang X A, Ma A J, Lei J L, et al. Comparison of the growing performance of different families of turbot (Scophthalmus maximus L.)[J]. Marine Sciences, 2011, 35(4): 1-8. [王新安, 马爱军, 雷霁霖, 等. 大菱鲆不同家系生长性能的比较[J]. 海洋科学, 2011, 35(4): 1-8.] |
| [13] |
Kocmarek A L, Ferguson M M, Danzmann R G. Differential gene expression in small and large rainbow trout derived from two seasonal spawning groups[J]. BMC Genomics, 2014, 15: 57. DOI:10.1186/1471-2164-15-57 |
| [14] |
Wang B, Tian Y Y, Sun C F, et al. Analysis of microRNA transcriptome and identification of growth related miRNA of Siniperca chuatsi[J]. Genomics and Applied Biology, 2017, 36(2): 603-613. [王博, 田园园, 孙成飞, 等. 翘嘴鳜microRNA转录组分析及生长相关miRNA鉴定[J]. 基因组学与应用生物学, 2017, 36(2): 603-613.] |
| [15] |
Fuentes E N, Valdés J A, Molina A, et al. Regulation of skeletal muscle growth in fish by the growth hormone-Insulin-like growth factor system[J]. General and Comparative Endocrinology, 2013, 192: 136-148. DOI:10.1016/j.ygcen.2013.06.009 |
| [16] |
Patterson S E, Mook L B, Devoto S H. Growth in the larval zebrafish pectoral fin and trunk musculature[J]. Developmental Dynamics, 2008, 237(2): 307-315. |
| [17] |
Shi Y, He M X. Differential gene expression identified by RNA-Seq and qPCR in two sizes of pearl oyster (Pinctada fucata)[J]. Gene, 2014, 538(2): 313-322. DOI:10.1016/j.gene.2014.01.031 |
| [18] |
Dai X Y, Zhang W, Zhuo Z J, et al. Neuroendocrine regulation of somatic growth in fishes[J]. Science China Life Sciences, 2015, 58(2): 137-147. |
| [19] |
Fuentes E N, Zuloaga R, Valdes J A, et al. Skeletal muscle plasticity induced by seasonal acclimatization involves IGF1 signaling:implications in ribosomal biogenesis and protein synthesis[J]. Comparative Biochemistry and Physiology Part B:Biochemistry & Molecular Biology, 2014, 176: 48-57. |
| [20] |
Pierce A L, Fox B K, Davis L K, et al. Prolactin receptor, growth hormone receptor, and putative somatolactin receptor in Mozambique tilapia:Tissue specific expression and differential regulation by salinity and fasting[J]. General and Comparative Endocrinology, 2007, 154(1-3): 31-40. DOI:10.1016/j.ygcen.2007.06.023 |
| [21] |
Edens A, Talamantes F. Alternative processing of growth hormone receptor transcripts[J]. Endocrine Reviews, 1998, 19(5): 559-582. |
| [22] |
Sun Y. Transcriptomic studies on the growth superiorities in a grouper hybrid (Epinephelus fuscogutatus ♀)×(Epinephelus lanceolatus ♂)[D]. Guangzhou: Sun Yat-sen University, 2016. [孙颖.棕点石斑鱼(♀)×鞍带石斑鱼(♂)杂交F1代生长优势的转录组学研究[D].广州: 中山大学, 2016.] http://cdmd.cnki.com.cn/Article/CDMD-10558-1016791189.htm
|
| [23] |
Li X L. Screening, cloning, identifying and function analyzing of genes involved in body weight regular in abdominal muscles of white shrimp[D]. Yangling: Northwest A & F University, 2011. [李喜莲.凡纳滨对虾肌肉组织中体重调控相关基因的筛选、克隆鉴定及功能研究[D].杨凌: 西北农林科技大学, 2011.] http://cdmd.cnki.com.cn/Article/CDMD-10712-1011402976.htm
|
| [24] |
Rechsteiner M C. Ubiquitin-mediated proteolysis: An ideal pathway for systems biology analysis[C]//Proceedings of the Conference on the Advances in Systems Biology. Boston: Springer, 2004: 49-59. https://link.springer.com/chapter/10.1007%2F978-1-4419-8861-4_5
|
| [25] |
Aedo J E, Maldonado J, Aballai V, et al. mRNA-seq reveals skeletal muscle atrophy in response to handling stress in a marine teleost, the red cusk-eel (Genypterus chilensis)[J]. BMC Genomics, 2015, 16: 1024. DOI:10.1186/s12864-015-2232-7 |
 2020, Vol. 27
2020, Vol. 27

