小黄黝鱼(Micropercops swinhonis)隶属硬骨鱼纲(Osteichthyes), 鲈形目(Perciformes), 沙塘鳢科(Eleotridae), 小黄黝鱼属, 分布于长江及其以北各大河流中, 喜成群生活于河流中下层及水草丛内, 以浮游动植物及藻类为食, 是中国特有小型淡水底栖鱼类[1]。在河南鱼类资源调查队对河南省境内鱼类资源野外调查过程中, 发现多条河流中鱼类多样性较低, 生境片段化与过度捕捞现象普遍存在, 部分河流为季节性河流且水利设施的建设导致部分河流下游水资源匮乏, 鱼类资源几乎消失, 而在水资源充沛的河段, 人口分布密集, 水资源污染状况较严重且存在违法捕捞现象, 野生小黄黝鱼资源面临威胁。
目前关于小黄黝鱼的研究主要集中在繁殖生物学、生态学及分类学[2-6]等方面, 2017年郑新等[7]曾对中国小黄黝鱼种群遗传结构和分化开展研究, 但在河南省境内样点设置仅黄河下游(濮阳)一处, 未涉及河南省其他水域, 不能全面反映河南省小黄黝鱼遗传多样性现状。在已完成的河南省鱼类资源调查1630个样点中仅少数样点获得小黄黝鱼样品, 且其中多个样点样本数量仅1~3尾, 与资料中记载的河南省常见种存在差距。河南省涵盖长江、黄河、淮河、海河四水系, 整体上西部山区东部平原的地势结构、气候环境均会对鱼类的种质资源产生影响。因此, 本研究选取具有代表性的10个采样点, 开展小黄黝鱼种群遗传多样性研究, 对全面了解河南省境内各水系小黄黝鱼遗传多样性现状及加强河南省野生鱼类资源保护工作具有重要意义。
线粒体DNA因其结构简单、遵循母系遗传且几乎无重组现象的特点常被用作分子标记, 其中, COI因其基因进化相对保守、足够的变异位点、序列长度合适等优势被普遍应用于群体遗传多样性研究[8-12]。本研究以线粒体COI基因为分子标记, 对采自河南省长江、黄河、淮河、海河四水系的小黄黝鱼样品进行遗传多样性研究, 以期全面了解河南省小黄黝鱼种质资源状况, 并为保护与合理利用小黄黝鱼种质资源提供科学依据。
1 材料与方法 1.1 实验材料选取河南省鱼类资源调查队2012年以来采集的10个地理群体小黄黝鱼样品作为分析对象, 样本使用地笼、刺网、撒网等工具采集, 根据形态学鉴定后, 取背部肌肉组织或鳍条放入无水乙醇中备用, 采样点分别涉及河南省长江水系、黄河水系、淮河水系及海河水系。研究样本信息如表 1所示。
|
|
表 1 小黄黝鱼样本信息 Tab.1 Sampling information of Micropercops swinhonis |
采用酚-氯仿法抽提基因组DNA[13]。利用1%琼脂糖凝胶电泳和紫外分光光度计检测DNA质量与含量, 将检测合格的基因组DNA放于–20℃保存, 用于后续试验。
线粒体COI基因扩增引物参考Hubert等[14], COI-F: TCTCAACCAACCATAAAGACATTGG, COI-R: TATACTTCTGGGTGCCCAAAGAATCA, 在商业公司合成引物。30 μL PCR反应体系包含: 15 μL MixTaq酶(Solarbio, 北京)、12 μL无菌双蒸水、1 μL DNA模板、上下游引物各1 μL(10 μmol/L)。PCR反应程序: 94℃预变性5 min; 94℃变性30 s, 55℃退火30 s, 72℃延伸1 min, 30次循环; 72℃终延伸10 min, 4℃保存。PCR产物经1%琼脂糖凝胶电泳检测合格后, 送至天一辉远生物科技有限公司进行双端测序, 测序引物同PCR引物。
1.3 数据分析将测序得到的峰图文件利用Lasergene version7.0中的SeqMan[15]进行组装拼接, 并进行人工校正, 将组装好的数据用Bioedit[16]进行多重序列比对获得一致序列用于后续分析。用DnaSP 5.0软件[17]计算单倍型多样性(haplotype diversity, Hd)和核苷酸多样性(nucleotide diversity, π)等遗传多样性参数, 并进行错配分布(mismatch distribution)分析; 用Arlequin 3.5[18]软件包中的AMOVA方法分析群体间遗传结构和遗传变异; 同时, 利用SAMOVA 2.0(Spatial Analysis of Molecular Variation)[19]软件进行单倍型的地理遗传结构检测, 尝试定义的分组数K为2~9, 每组1000次重复, 观察所得结果FCT的变化规律, FCT处在峰值时对应的K即是最佳分组数; 采用群体遗传分化指数(FST)评价种群间的遗传分化程度; 采用Fu’s FS 检验和Tajima’s D进行中性检验, 结合错配分布结果推断群体历史是否发生过近期扩张; 用MEGA 6.0[20]计算群体间的遗传距离, 并以中华沙塘鳢(Odontobutis sinensis)作为外类群, 构建最大似然树(maximum-likelihood, ML); 利用FigTree v1.4.2对系统发育树进行编辑, 利用NETWORK 5.0软件构建单倍型网络图分析单倍型间进化关系; 运用BEAST v 1.7.4[21]软件, 探讨种群历史动态, 由Tracer v1.5[22]生成贝叶斯天际线点图(Bayesian Skyline Plot, BSP)。
2 结果与分析 2.1 序列特征及遗传多样性对10个不同地理种群77尾小黄黝鱼样品COI基因进行测序, 获得一致序列长度为640 bp, 变异位点21个, 占总碱基数的3.28%;简约信息位点8个, 占总碱基数1.25%; G(18.5%)的含量明显低于T (31.9%)、C(25.6%)和A(24.0%)的含量; 共检测到24个单倍型, 其中, Hap_2 (PH、BSH、HZH、TRQ、YH、QH、WH)与Hap_1 (PH、TRQ、HXC、WH)为4水系共享; Hap_3 (PH、BSH、HXC)与Hap_5 (BSH、HXC)为长江与淮河水系共享; Hap_7 (MJH、HZH、TRQ、XLSSK、HXC、WH)为黄河与淮河水系共享; Hap_8 (MJH、QH)为黄河与海河水系共享, 其余为水系特有单倍型(表 2)。
|
|
表 2 河南省四水系各群体的遗传多样性信息 Tab.2 Genetic diversity of Micropercops swinhonis populations from four river systems in Henan Province |
遗传多样性分析结果显示(表 2), 所有样本平均核苷酸多样性π=0.00361, 单倍型多样性Hd = 0.89088。各群体的核苷酸多样性在0.00078~0.00433之间变化, 单倍型多样性在0.60000~ 0.93333之间变化, 小黄黝鱼种群显示较高单倍型多样性与低核苷酸多样性的特点。
2.2 小黄黝鱼群体的遗传结构各群体间遗传分化指数FST变化范围为‒0.11139~0.62264, 其中HZH(‒0.11139~0.38889)、TRQ(‒0.11139~0.28749)、WH(‒0.09213~0.33159)与其他群体间(除PH外)存在显著性差异(P < 0.05); MEGA 6.0分析结果显示, 群体间遗传距离在0.001~0.003之间(表 3)。
|
|
表 3 河南省四水系小黄黝鱼群体间遗传分化指数(FST, 对角下方)和遗传距离(对角上方) Tab.3 Pairwise FST (below diagonal) and genetic distance (above diagonal) among Micropercops swinhonis populations from four river systems in Henan Province |
将小黄黝鱼群体按照水系划分为4个组群(长江水系、黄河水系、淮河水系、海河水系)进行AMOVA分析, 结果表明, 主要遗传变异来自种群内, 占78.42%, 组间遗传变异占16.00%(表 4)。
|
|
表 4 河南省四水系小黄黝鱼群体AMOVA分析 Tab.4 Analysis of molecular variance (AMOVA) among Micropercops swinhonis populations from four river systems in Henan Province |
SAMOVA分析结果表明, 主要遗传变异来自种群内(表 5), 并且随着分组数K的增加, 组间遗传差异总体上呈现增长趋势, 并且在K为9时出现最大值(图 1)。
|
|
表 5 不同分组(K)时小黄黝鱼群体的SAMOVA分析结果 Tab.5 The SAMOVA results of Micropercops swinhonis populations under different group numbers (K) |
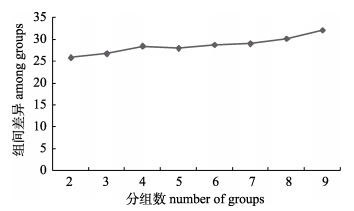
|
图 1 小黄黝鱼种群结构的SAMOVA结果 Fig.1 SAMOVA results of Micropercops swinhonis population structure |
利用MEGA 6.0, 以中华沙塘鳢作为外类群, 根据小黄黝鱼24个单倍型, 基于K2P双参模型[23], Bootstrap设置为1000次[24], 其余参数按默认值设置, 利用最大似然法构建ML系统发育树(图 2), 结果显示, 群体间交叉嵌套现象普遍, 没有形成明显的谱系。
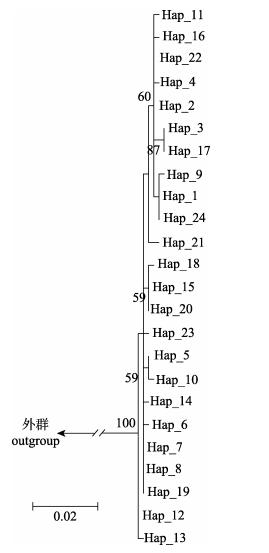
|
图 2 小黄黝鱼10个种群COI单倍型的ML系统树 Fig.2 ML phylogenetic tree of Micropercops swinhonis based on COI haplotypes from ten populations |
利用NETWORK 5.0构建单倍型网络图(图 3), 显示了与ML树相似的结果, 小黄黝鱼不同水系的单倍型散在分布于整个网络上, 没有明显的地理结构。
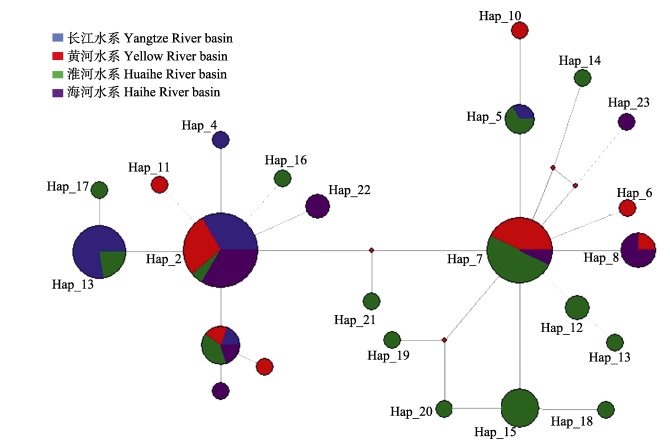
|
图 3 基于NETWORK 5.0软件构建的小黄黝鱼群体单倍型网络图 Fig.3 Network of haplotypes of Micropercops swinhonis using NETWORK 5.0 |
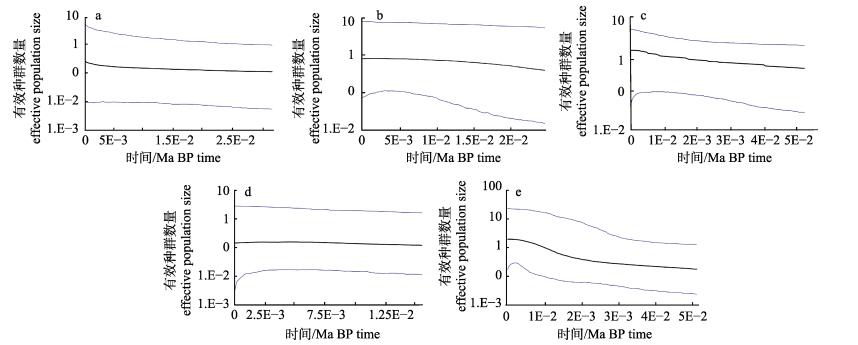
按照不同水系进行种群错配分布(mismatch distribution)分析。结果显示, 长江、黄河、淮河、海河4个水系中的小黄黝鱼均呈现多峰分布(图 4a, 4b, 4c, 4d), 而将所有群体作为一个整体进行分析时呈单峰分布(图 4e); BSP结果显示, 长江水系(图 5a)、黄河水系(图 5b)、淮河水系(图 5c)以及海河水系(图 5d)都呈现出逐步上升的趋势, 将这些水系的小黄黝鱼群体作为一个整体来分析结果显示, 河南省小黄黝鱼群体同样呈现出整体逐步增长的趋势, 并且在0.02 Ma BP左右增长速度开始加快(图 5e)。
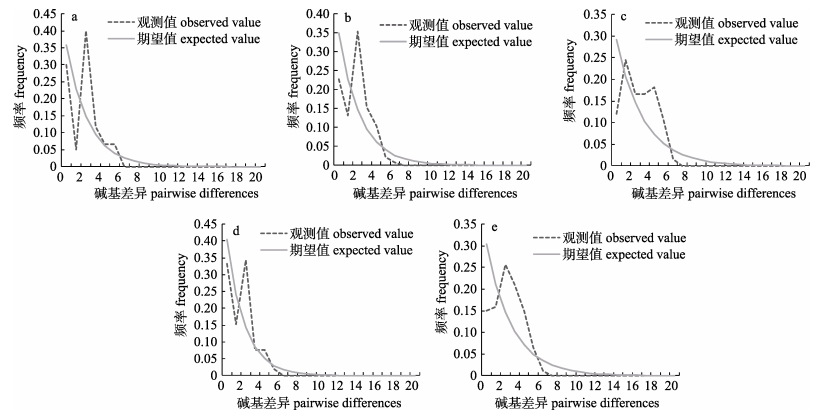
|
图 4 河南省四水系中小黄黝鱼群体错配分布分析 a.长江水系; b.黄河水系; c.淮河水系; d.海河水系; e.整体. Fig.4 Mismatch distribution analysis of Micropercops swinhonis sampled from four river systems in Henan Province a. Yangtze River; b. Yellow River; c. Huaihe River; d. Haihe River; e. total. |
|
图 5 河南省四水系中小黄黝鱼群体贝叶斯天际线点图 a.长江水系; b.黄河水系; c.淮河水系; d.海河水系; e.整体. Fig.5 BSP (Bayesian skyline plots) of Micropercops swinhonis sampled from four river systems in Henan province a. Yangtze River; b. Yellow River; c. Huaihe River; d Haihe River; e. total. |
Tajima’s D与Fu’s FS分析结果, 各群体均没有出现显著负值, 但总体水平上Fu’s FS中性检验结果为显著负值。
3 讨论 3.1 小黄黝鱼遗传多样性分析核苷酸多样性(π)由于累积时间远大于单倍型多样性(Hd), 更能反映群体的遗传多样性, 当π为0.0015~0.0047时, 显示出较低的遗传多样性[25]。本研究中, 小黄黝鱼群体表现出较低的核苷酸多样性, 其中黄河水系的MJH群体核苷酸多样性(π=0.00078)最低, 而淮河水系HXC、YH群体以及长江水系PH群体、海河水系WH群体核苷酸多样性都较MJH群体高, 说明长江、淮河、海河三水系的小黄黝鱼遗传多样性高于黄河水系, 在鱼类资源野外调查工作中笔者发现, 黄河水系较少发现小黄黝鱼群体且捕捞的个体数量较少。黄河水系小黄黝鱼种质资源出现衰退现象, 与野外调查工作实际相吻合。本研究中调查区域内小黄黝鱼群体具有高Hd (0.89088)低π (0.00361)的遗传多样性模式, 与郑新等[7]对小黄黝鱼群体的研究结果一致。相似的遗传多样性特征还在河南省其他鱼类群体中发现, 程清清等[26]对河南省黄河及卫河(海河水系)中的䱗(Hemiculter leucisculus)和棒花鱼(Abbottina rivularis)进行研究, 结果显示两种鱼都同样表现出高Hd低π的模式; 方冬冬等[27]对淮河源区䱗研究时也出现相同的遗传多样性模式, 并且他们均提出种群近期可能经历了瓶颈效应后的小群体急剧扩张, 这可能是导致高Hd低π模式的原因, 另外人为干扰及水库大坝建设也可能导致遗传多样性低。因此, 笔者推测小黄黝鱼群体出现高Hd低π的遗传多样性模式可能是由于: (1)水资源污染、水利设施建设等使鱼类生境退化、种群扩散与种群间基因交流受阻, 遗传漂变现象增加[28-29]; (2)小黄黝鱼群体历史动态显示在近期经历了种群扩张事件, 单倍型数量增加而未积累明显的核苷酸差异; (3)原生观赏鱼市场的兴起可能导致对小黄黝鱼捕捞量的增加, 加速种群数量急剧减少。有研究表明, 环境适应能力与遗传多样性成正比[9], 目前河南省小黄黝鱼遗传多样性较低表明其环境适应能力较弱, 因此, 为防止小黄黝鱼遗传多样性遭到破坏, 应当对其采取科学合理的保护措施, 尽可能地减少人类干扰活动, 提供更优质的生存环境。
3.2 小黄黝鱼种群遗传结构分析FST作为衡量群体间遗传分化程度的重要指标, 超过0.25时表示遗传分化很大[30], 小黄黝鱼种群间仅37.78%的FST > 0.25, 其中58.82%来源于长江水系(PH、BSH)。另外, 各种群均存在特有单倍型, 表明种群间存在一定程度遗传分化, 但总体而言小黄黝鱼种群间分化程度较低。长江水系(PH、BSH)与其他水系小黄黝鱼种群间存在更多的遗传分化, 刘慧芬等[31]对河南省四水系宽鳍鱲(Zacco platypus)的研究结果也显示, 长江水系中宽鳍鱲群体同其他水系群体间存在较大分化, 并指出基因交流存在阻碍是主要原因。因此, 笔者推测长江水系小黄黝鱼群体具有更显著的群体间遗传分化可能是因为底栖鱼类移动能力弱, 加之PH、BSH与其他水系间种群由于大别山产生天然的隔离, 导致缺乏基因交流的机会。
AMOVA结果显示出主要的遗传变异来源于种群内, 但组间变异量占16.00%, FST值显示出黄河与淮河, 黄河与海河, 两两之间FST值比较显示出了分化且数据具有显著性差异, 但分化水平较低, 结合单倍型网络和系统发育树, 结果反映调查区域内种群没有形成显著的水系及地理分布格局, 但一些水系的种群间表现出较弱的遗传分化。SAMOVA结果也表明主要的遗传变异来源于种群内, 随着分组数量的增加而呈现上升趋势而不存在单峰结构, 同样表明小黄黝鱼群体不具有显著的地理结构。李强[32]对长江与淮河流域中沙塘鳢属鱼类的研究发现, 不同地理区域也出现了交叉聚类的现象, 并表示基因交流可能是导致该现象的原因。我们推测河南省不同地理种群间的小黄黝鱼存在基因交流, 据文献记载, 黄河、长江、淮河等历史上均有洪泛灾害、水系变迁的情况[33-35], 而且引黄济卫、南水北调以及上下游省份的跨流域调水工程[36-37]也会造成水系间沟通。除此之外, 原生鱼作为观赏鱼的兴起, 引种等过程也可能导致种群地理隔离被打破[7], 这些情况都可能使各水系小黄黝鱼种群间存在基因交流, 从而导致分化关系模糊。
3.3 小黄黝鱼种群历史动态分析Fu’s FS中性检验结果为显著负值, 且整体的错配分布图具有明显单峰, 表明河南省小黄黝鱼群体近期发生过群体扩张事件。从河南地区小黄黝鱼的群体历史动态来看, 无论是按照单个水系分析还是作为整体进行分析, 结果均呈逐步增长趋势。由于自然气候、河流地质变迁、人类活动等因素均对鱼类群体历史动态产生影响[38-39], 小黄黝鱼的有效种群数量增长趋势在不同水系、各个历史时期均呈现出不同的增长趋势, 此外, 调查区域属于华北区, 是典型的季风区, 0.080~ 0.117 Ma BP期间总的气候类型为暖湿气候[40], 适宜小黄黝鱼生存、繁殖, 可能是引起小黄黝鱼有效种群数量总体呈现增长趋势加快的原因。
| [1] |
Wu H L, Zhong H S. Faunica Sinica, Osteichthyes, Perciformes (V) Gobioidei[M]. Beijing: Science Press, 2008: 141-145. [伍汉霖, 钟汉生. 中国动物志硬骨鱼纲鲈形目(五)虾虎鱼亚目[M]. 北京: 科学出版社, 2008: 141-145.]
|
| [2] |
Kim I S, Kim B J. Population ecology of the goby, Micropercops swinhonis in the Puan-gun, Ch'ollabuk-do, Korea[J]. Korean Journal of Limnology, 1997, 30: 47-54. |
| [3] |
Kim I S. Breeding habits and egg development of the goby, Micropercops swinhonis[J]. The Korean Journal of Ecology, 1996, 19(5): 477-486. |
| [4] |
Iwata A, Sakai H, Shibukawa K, et al. Developmental characteristics of a freshwater goby, Micropercops swinhonis, from Korea[J]. Zoological Science, 2001, 18(1): 91-97. DOI:10.2108/zsj.18.91 |
| [5] |
Nichol J T. The freshwater fishes of China[J]. Nature History of Central Asia, 1943, 9: 258-259. |
| [6] |
Yi B L. Notes on species of genus Hypseleotris (Eleotridae) with respect to its sex dimorphism[J]. Journal of Huazhong Agricultural College, 1982, 1(3): 72-77. [易伯鲁. 黄黝鱼属的种类及其两性异形[J]. 华中农学院学报, 1982, 1(3): 72-77.] |
| [7] |
Zheng X, Li C H. Population structure and genetic divergence of Micropercops swinhonis[J]. Journal of Shanghai Ocean University, 2017, 26(3): 358-365. [郑新, 李晨虹. 中国小黄黝鱼种群遗传结构和分化研究[J]. 上海海洋大学学报, 2017, 26(3): 358-365.] |
| [8] |
Amor M D, Norman M D, Cameron H E, et al. Allopatric speciation within a cryptic species complex of Australasian octopuses[J]. PLoS ONE, 2014, 9(6): e98982. DOI:10.1371/journal.pone.0098982 |
| [9] |
Hu J, Hou X Y, Yin S W, et al. Genetic diversity and divergence of cheilinus undulatus of different geographic populations of the South China Sea revealed by COI and Cyt b gene analyses[J]. Acta Hydrobiologica Sinica, 2014, 38(6): 1008-1016. [胡静, 侯新远, 尹绍武, 等. 基于mtDNA COI和Cyt b基因序列对南中国海不同海域波纹唇鱼群体遗传多样性的研究[J]. 水生生物学报, 2014, 38(6): 1008-1016.] |
| [10] |
Shan Y J, Lu C Y, Li C, et al. Study of DNA barcoding and genetic variation based on the mitochondrial COI gene sequences in five carp varieties[J]. Journal of Fishery Sciences of China, 2013, 30(5): 931-938. [单云晶, 鲁翠云, 李超, 等. 基于线粒体COI基因序列的5种鲤养殖品种遗传多样性研究[J]. 中国水产科学, 2013, 30(5): 931-938.] |
| [11] |
Zhang Y, Zhou C J, Gu Q H, et al. Molecular phylogenetic analysis of gobies in the north of Henan based on COI and D-Loop sequences[J]. Journal of Henan Normal University (Natural Science Edition), 2016, 44(5): 94-100. [张毅, 周传江, 顾钱洪, 等. 基于COI和D-Loop序列豫北虾虎鱼分子系统分析[J]. 河南师范大学学报(自然科学版), 2016, 44(5): 94-100.] |
| [12] |
Liang H W, Meng Y, Luo X Z, et al. Genetic diversity of six Monopterus albus populations based on COI gene sequences[J]. Journal of Fishery Sciences of China, 2018, 25(4): 837-846. [梁宏伟, 孟彦, 罗相忠, 等. 基于线粒体COI基因的6个黄鳝群体遗传多样性[J]. 中国水产科学, 2018, 25(4): 837-846.] |
| [13] |
Sambrook J, Fritsch E F, Maniatis T. Molecular Cloning: A Laboratory Manual[M]. New York: Cold Spring Harbor Laboratory Press, 1989: 1626.
|
| [14] |
Hubert N, Hanner R, Holm E, et al. Identifying Canadian freshwater fishes through DNA barcodes[J]. PLoS ONE, 2008, 3(6): e2490. DOI:10.1371/journal.pone.0002490 |
| [15] |
Swindell S R, T N Plasterer. Seqman. Contig assembly[J]. Methods in Molecular Biology, 1997, 70(1): 75-89. |
| [16] |
Tippmann H F. Analysis for free: Comparing programs for sequence analysis[J]. Briefings in Bioinformatics, 2004, 5(1): 82-87. DOI:10.1093/bib/5.1.82 |
| [17] |
Rozas J, Rozas R. DnaSP, DNA sequence polymorphism: an interactive program for estimating population genetics parameters from DNA sequence data[J]. Bioinformatics, 1995, 11(6): 621-625. DOI:10.1093/bioinformatics/11.6.621 |
| [18] |
Excoffier L, Laval G, Schneider S. Arlequin (version 3.0): An integrated software package for population genetics data analysis[J]. Evolutionary Bioinformatics, 2005, 1: 47-50. |
| [19] |
Dupanloup I, Schneider S, Excoffier L. A simulated annealing approach to define the genetic structure of populations[J]. Molecular Ecology, 2002, 11(12): 2571-2581. DOI:10.1046/j.1365-294X.2002.01650.x |
| [20] |
Tamura K, Stecher G, Peterson D, et al. MEGA6: Molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [21] |
Drummond A J, Suchard M A, Xie D, et al. Bayesian phylogenetics with BEAUti and the BEAST 1.7[J]. Molecular Biology and Evolution, 2012, 29(8): 1969-1973. DOI:10.1093/molbev/mss075 |
| [22] |
Rambaut A, Drummond A J. Tracer v1.4[J]. Encyclopedia of Atmospheric Sciences, 2007, 141(3567): 2297-2305. |
| [23] |
Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. Journal of Molecular Evolution, 1980, 16(2): 111-120. DOI:10.1007/BF01731581 |
| [24] |
Felsenstein J. Confidence limits on phylogenies: An approach using the bootstrap[J]. Evolution, 1985, 39(4): 783-791. DOI:10.1111/j.1558-5646.1985.tb00420.x |
| [25] |
Lan H, Shi L M. The origin and genetic differentiation of native breeds of pigs in southwest China: An approach from mitochondrial DNA polymorphism[J]. Biochemical Genetics, 1993, 31(1-2): 51-60. DOI:10.1007/BF02399819 |
| [26] |
Cheng Q Q, Gu Q H, Fang D D, et al. Comparison of population genetic structure of two co-distributed cyprinid species[J]. Journal of Fisheries of China, 2017, 41(8): 1169-1182. [程清清, 顾钱洪, 方冬冬, 等. 同域分布两种鲤科鱼类种群遗传结构的比较[J]. 水产学报, 2017, 41(8): 1169-1182.] |
| [27] |
Fang D D, Gu Q H, Zhou C J, et al. Genetic diversity of hemiculter leucisculus in fountainhead area of Huaihe River[J]. Fisheries Science, 2018, 37(5): 665-673. [方冬冬, 顾钱洪, 周传江, 等. 淮河源区䱗群体遗传多样性研究[J]. 水产科学, 2018, 37(5): 665-673.] |
| [28] |
González-Astorga J, Núñez-Farfán J. Effect of habitat fragmentation on the genetic structure of the narrow endemic Brongniartia vazquezii[J]. Evolutionary Ecology Research, 2001, 3(7): 861-872. |
| [29] |
Harrison R. Molecular changes at speciation[J]. Annual Review of Ecology and Systematics, 1991, 22(1): 281-308. DOI:10.1146/annurev.es.22.110191.001433 |
| [30] |
Freeland J R. Molecular Ecology[M]. Chichester: John Wiley & Sons, 2005: 112-116.
|
| [31] |
Liu H F, Zhang C, Wang J, et al. Genetic diversity of wild Zacco platypus populations among four river systems in Henan Province analyzed by mitochondrial COI gene[J]. Journal of Fishery Sciences of China, 2018, 25(2): 269-277. [刘慧芬, 张超, 王静, 等. 河南境内四水系宽鳍鱲野生群体的遗传多样性[J]. 中国水产科学, 2018, 25(2): 269-277.] |
| [32] |
Li Q. Multilocus genetic diversity and population structure for the Odontobutis distributed in Yangtze river basin and Huai River[D]. Shanghai: Shanghai Ocean University, 2015. [李强.基于多基因位点的长江流域及淮河流域沙塘鳢属鱼类遗传多样性与种群遗传结构分析[D].上海: 上海海洋大学, 2015.] http://cdmd.cnki.com.cn/Article/CDMD-10264-1015974879.htm
|
| [33] |
Li C. Study on overground river of Ergu River in Northern Song Dynasty[J]. Historical Geography, 2013, 12(2): 57-70. [李丞. 北宋二股河地上河问题研究[J]. 历史地理, 2013, 12(2): 57-70.] |
| [34] |
Ye Q C. Study on the erosion environmental development orientation in the middle Yellow River[J]. Yellow River, 1996(11): 11-15. [叶青超. 黄河下游地上河发展趋势研究[J]. 人民黄河, 1996(11): 11-15.] |
| [35] |
Zhang Y Y, Li Z L, Liu X J. Evolution of interconnected river and lake networks in the Huai River Basin over the last millennium[J]. South-to-North Water Transfers and Water Science & Technology, 2016, 14(4): 77-83. [张永勇, 李宗礼, 刘晓洁. 近千年淮河流域河湖水系连通演变特征[J]. 南水北调与水利科技, 2016, 14(4): 77-83.] |
| [36] |
Jin M. Research on the urbanization response of interconnected river system network in Zhengzhou City[D]. Zhengzhou: Zhengzhou University, 2014. [靳梦.郑州市水系连通的城市化响应研究[D].郑州: 郑州大学, 2014.] http://cdmd.cnki.com.cn/article/cdmd-10459-1014392861.htm
|
| [37] |
Yu L. Research on the river system form structure and connectivity of Huaihe river basin[D]. Zhengzhou: Zhengzhou University, 2017. [于璐.淮河流域水系形态结构及连通性研究[D].郑州: 郑州大学, 2017.] http://cdmd.cnki.com.cn/Article/CDMD-10459-1017150510.htm
|
| [38] |
Gao Y, Wang S Y, Guo J. Quaternary palaeoenvironmental oscillations drove the evolution of the Eurasian Carassius auratus complex (Cypriniformes, Cyprinidae)[J]. Journal of Biogeography, 2012, 39(12): 2264-2278. DOI:10.1111/j.1365-2699.2012.02755.x |
| [39] |
Flanagan S P, Rose E, Jones A G. Population genomics reveals multiple drivers of population differentiation in a sex-role-reversed pipefish[J]. Molecular Ecology, 2016, 25(20): 5043-5072. DOI:10.1111/mec.13794 |
| [40] |
Shen Y P, Kang J C. A 150 ka climatic record of loess from grain-size data in Linxia, Gansu[J]. Arid Land Geography, 1992, 15(3): 44-49. [沈永平, 康建成. 十五万年来临夏黄土粒度变化的气候记录[J]. 干旱区地理, 1992, 15(3): 44-49.] |
 2020, Vol. 27
2020, Vol. 27

