石磺是一种栖息于潮间带滩涂区域, 以土壤中藻类为食的肺螺类生物, 隶属于软体动物门(Mollusca)、石磺科(Onchidiidae)[1]。中国目前主要有4种石磺, 其中瘤背石磺(Onchidium reevesii)广泛分布于从江苏到广东东南部的沿海地区[2]。自然状态下瘤背石磺退潮后出来觅食, 涨潮前必须及时进入洞穴中或者爬至芦苇枝、红树或者潮上带湿地躲避海水, 表现出能准确感知当地海域涨潮的能力。石磺能够感知潮汐及时归穴的行为早在100多年前就已经引起了学术界的关注[3]。涨潮引起潮水的交换, 带来丰富的浮游植物和底栖藻类, 为一些滤食性的双壳贝类提供了充足的食物来源, 如太平洋牡蛎、长牡蛎等[4]。同样,潮水的交换会引起潮间带底栖藻类的更替,为瘤背石磺提供了食物来源。瘤背石磺及时感知当地半日潮的(12.4 h)潮汐规律, 形成潮汐记忆能力后归穴的行为机制一直未见报道。
海面波浪会产生5~10 Hz的环境噪声, 海流会产生10~200 Hz的声音; 低频声音对海洋动物的行为、生理以及早期发育、变态及生长都有一定的影响[5]。Kaifu等[6]研究了不同频率声音刺激对章鱼(Octopus ocellatus)在水下呼吸活动的作用, 发现声音频率在50~150 Hz范围时, 章鱼延长了在水中的呼吸活动。声音刺激后, 章鱼延长在水中屏蔽呼吸的时间以探测猎物。Buscaino等[7]研究单个和由25只组成的群体欧洲棘龙虾(Palinurus elephas)分别在有无捕食者章鱼的条件下的信号发生情况, 经过声学分析后发现, 在有捕食者时龙虾发出刺耳的声音, 且群体龙虾发生的信号更强, 这种信号可能与个体之间的交流有关。Cao等[8]研究了人类噪音对缢蛏(Sinonovacula constricta)下潜行为的影响机理, 揭示其可能是经体表感应震动感知噪音污染, 从而改变代谢和肌肉运动, 促使下潜行为的显著变化。栖息于潮间带的无脊椎动物对环境变化非常敏感, 其生活习性很大程度上受到潮汐变化的影响, 其中最重要的摄食和躲避敌害行为与潮汐节律有密切的联系。潮汐的能量主要集中在低频部分, 潮汐预报精度关键在低频声音部分(5~100 Hz)[9]。据刘杰等[10]在瘤背石磺产卵与天文潮汐的关系研究中发现, 在7—9月, 瘤背石磺在每个月的小潮集中大量产卵, 在大潮来临之前结束产卵, 证明瘤背石磺的产卵与潮汐相关。瘤背石磺对当地潮汐的节律性具有明显感知,这是否与潮汐来临时产生的低频声音有关, 它是否通过长期感知潮汐产生的低频声音, 形成了对潮汐记忆的功能。
钙调素(calmodulin, CaM)蛋白是由148~152个氨基酸组成的小分子多功能钙感应蛋白, 广泛分布于动植物细胞中, 参与细胞的多种生理功能调节[11]。类钙调素蛋白(calmodulin-like, CaM-like)与钙调素一样, 属于EF-hand超家族蛋白成员之一, 具有多种生物学功能, 如参与核苷酸代谢[12]、细胞增殖分化[13]、糖原代谢[14]、神经元之间的信息传递[15]以及基因表达[16]。最近的研究也表明CaM-like基因参与钙离子的释放和运输以及细胞分化[17-18]。Ca2+是细胞内信息传递的第二信使, CaM是Ca2+的主要受体, CaM在Ca2+信号传导中具有非常重要的作用[19]。有研究报道, CaM在细胞内无自主活性, 当机体受到环境胁迫、病毒入侵和机械损伤等外界因素的刺激时, Ca2+内流与CaM结合形成Ca2+/CaM复合物后被激活, 从而激活下游基因CaMKII, 引起相应的生理行为变化[20-21]。CaMKII是Ser/Thr激酶家族成员之一, 在生物体内广泛存在, 对多种蛋白质的生理功能的磷酸化有重要的作用[22]。在钙离子–钙调蛋白依赖性蛋白激酶Ⅱ(Ca2+-CaM-CaMKII)的神经系统突触信号传递中, CaMKII发挥着重要的作用, CaMKII的缺失会影响信号的传递, 使空间学习和记忆能力受损, 而CaMKII的激活参与LTP的过程, 这说明CaMKII是空间学习和记忆能力的基础[23]。
本实验在瘤背石磺神经转录组的基础上利用RACE技术首次克隆出CaM-like基因的cDNA全长, 通过生物信息分析软件分析其序列; 利用qRT-PCR技术检测CaM-like基因在瘤背石磺背部皮肤、腹足、神经节、肠、肝胰腺、口器和蛋白腺的分布情况; 通过荧光定量PCR检测CaM-like基因在不同声波频率刺激下不同时间点的表达量和CaMKII基因在不同声波频率连续刺激12.4 h时的表达量变化, 进一步阐明CaM-like基因和CaMKII基因在瘤背石磺神经节部位中感知模拟潮汐声波频率的功能。本研究将为今后探究海洋无脊椎动物对感知潮汐节律的深入研究提供依据。
1 材料与方法 1.1 实验材料瘤背石磺采集于上海市崇明岛(121.40°E, 31.62°N)半日潮低潮时, 暂养于实验室内的培养箱, 每天定时投喂玉米粉, 添加水分保持土壤湿润, 定期清除死亡个体和清理粪便。实验室的温度控制在(25±0.5) ℃, 每天12 h光照和12 h黑暗, 暂养15 d后, 实验个体体重(3.58±0.11) g, 体长(7.60± 0.12) cm。
1.2 实验方法 1.2.1 总RNA的提取和cDNA克隆选取体长和体重相近的成年健康瘤背石磺5只分别剪取神经节部位, 采用Trizol (TaKaRa, 大连)试剂提取总RNA, 取质量好的RNA为模板, 采用PrimerScriptTM RT试剂盒(TaKaRa, 大连)反转录为cDNA, 于‒20 ℃保存。
1.2.2 CaM-like基因cDNA全长的克隆从本实验室所测的神经转录组数据库中筛选出一条与神经信号传递有关的部分序列, 进行NCBI-BLAST比对, 结果显示与其他无脊椎动物的类钙调素蛋白基因具有较高的同源性, 命名其为瘤背石磺类钙调素蛋白基因(Or. CaM-like gene)。利用Premier 5.0软件设计引物CaM-like-F1、CaM-like-R1 (表 1)对该部分片段进行验证。PCR反应体系为50 μL: 2×Taq Master Mix 25 μL (TaKaRa, 上海), CaMKIV-F1 1.5 μL, CaMKIV-R1 1.5 μL, ddH2O 20 μL, cDNA 2 μL。PCR反应程序: 94 ℃预变性3 min; 94 ℃ 30 s, 60.2 ℃ 30 s, 72 ℃ 60 s, 38个循环; 72 ℃延伸10 min。扩增产物经1%琼脂糖凝胶电泳检测后, 采用DNA割胶回收试剂盒(TIANGEN, 北京)回收目的条带, 连接到pGEM-T vector (Promega, 美国), 于16 ℃过夜连接14 h, 并转化到大肠杆菌(DH5α, 天根), 挑取阳性克隆菌落培养, 菌液PCR引物M13 (表 1), 通过菌液PCR后送生工生物工程(上海)有限公司测序。
|
|
表 1 实验所用的引物序列 Tab.1 The sequences of primers used in this study |
Or. CaM-like gene cDNA全长5′端和3′端的克隆采用RACE (rapid-amplification of cDNA ends)技术, 按照SMARTER®RACE5′/3′Kit User Manual (TaKaRa, Japan)进行, 克隆过程中所用的引物如表 1所示。采用割胶回收试剂盒回收目的条带, 连接到pGEM-T vector, 通过LB平板蓝白斑筛选阳性克隆, 挑取白斑菌落, 经过菌液PCR反应检测后, 挑取目的菌液送生工生物工程(上海)有限公司测序。测序成功的序列利用BioEdit软件进行拼接, 获得CaM-like基因的cDNA全长。
1.2.3 序列信息分析利用Home-ORFinder-NCBI (https://www.ncbi.nlm.nih.gov/orffinder/)查找开放阅读框(ORF); 使用在线网站(http://web.expasy.org/protparam/)预测编码蛋白的理化性质; 利用Signal 4.1 (http://www.cbs.dtu.dk/services/SignalP/)分析信号肽; 使用SWISS-MODEL (http://swissmododel.expasy.org/)在线分析蛋白质的三维结构; 从NCBI基因库中获取其他物种的CaM-like基因编码的氨基酸序列, 使用Clustal X软件进行氨基酸序列多重比对, 利用MEGA 6.0软件采用邻位法(Neighbor-Joining)构建系统进化树, 1000次bootstraps, 其他参数均使用默认值。
1.2.4 实时荧光定量PCR选取在实验室内暂养的9只体重和体长相似的健康瘤背石磺, 分别取背部皮肤、腹足、神经节、肠、肝胰腺、口器、蛋白腺等共7个组织, 将3只个体做成一个混样, 共3个实验样品组, 采用Trizol试剂提取总RNA, 并将其反转成cDNA, ‒20 ℃保存。根据CaM-like基因的ORF框内的基因序列利用在线网(https://www.genscript.com/tools/real-time-pcr-tagman-primer-tool)设计qRT-PCR引物为CaM-like-RT-F和CaM-like-RT-R (表 1)。以管家基因18S rRNA为内参基因, 使用SYBR premix Ex TaqTM Ⅱ试剂盒(Vazyme Biotech, 南京)在ABI Q6仪器上进行qRT-PCR。反应体系为20 μL: Premix Ex TaqTM(2×) 10 μL、引物(10 mol/L)各0.8 μL、ddH2O 6.4 μL、cDNA模板2 μL。采用相对定量算法[24]计算基因的相对表达量。
1.2.5 不同频率的声波刺激下CaM-like基因在神经节的相对表达量选取210个健康的、体重和体长相似的瘤背石磺平均分成6个实验组, 放置在定制塑料培养箱(150 cm×80 cm×60 cm), 每个塑料箱由隔音玻璃隔开, 塑料箱内铺一层约10 cm厚的潮间带滩涂的土壤, 放置在实验室内, 保持土壤湿润, 控制温度在(25±0.5) ℃, 光周期为12 h光照:12 h黑暗。利用AWA1651型信号发生器, 连接到SA-JZ系列强力电动式激振器(无锡世敖科技有限公司, 江苏)发出正弦波声波, 增益强度7~14 Upk, 以声波频率分别为0 Hz、5 Hz、25 Hz、50 Hz、75 Hz、100 Hz连续刺激瘤背石磺8 d, 期间定时定点投喂玉米粉, 在第8天时分别在不同频率声波连续刺激下0 h、6.2 h、12.4 h、24 h取样, 其中0 h对照组为在室内暂养的石磺, 没有声波刺激的空白组。每个时间点取9个个体, 剪取神经节部位保存于液氮中, 采用Trizol试剂分别提取RNA, 并将其反转成cDNA, –20 ℃保存。实时荧光定量PCR检测CaM-like基因在不同声波频率刺激下, 不同时间点的表达量, 实验步骤同1.2.4。
1.2.6 不同声波频率刺激下12.4 h CaMKII基因在神经节的相对表达量取步骤1.2.5中不同波频率连续刺激12.4 h时的瘤背石磺神经节部位的mRNA, 进行实时荧光定量PCR, 检测CaMKII基因在不同声波频率刺激下的表达量[24]。根据瘤背石磺神经转录组CaMKII基因的ORF框内的基因序列, 利用在线网站(https://www.genscript.com/tools/real-time-pcr-tagman-primer-tool)设计qRT-PCR引物CaMKII-RT-F和CaMKII-RT-R(表 1), 实验步骤同1.2.4。
1.3 数据处理与分析相对表达量用平均值±标准差(x±SD)表示。对1.2.4、1.2.5和1.2.6的数据利用SPSS 19.0进行单因素方差分析(One-way ANOVA), 采用LSD、SNK和Duncan法进行差异显著性分析, P < 0.05表示具有显著性差异。其中1.2.5是在同一频率下对不同时间点进行单因子内的多重比较分析。
2 结果与分析 2.1 CaM-like cDNA的全长序列通过RACE PCR克隆出瘤背石磺CaM-like基因的cDNA, 全长为2321 bp (GenBank登录号: MK986829), 包括5′非编码区(UTR)366 bp, 3′非编码区(UTR) 1337 bp, 618 bp的开放阅读框。3′UTR区含有多聚核苷酸的加尾信号序列AATAAA和Poly(A)尾巴(图 1)。经过各种软件预测CaM-like基因共编码206个氨基酸, 多肽链分子量23029.64 kD, 理论等电点4.64, 分子式是C1018H1544N274O320S9。N端信号肽由29个氨基酸组成, 表明该蛋白跨膜到细胞外发挥作用。氨基酸序列多重比对结果显示, 瘤背石磺CaM-like基因的氨基酸序列与其他无脊椎动物CaM-like基因的氨基酸序列具有较高的同源性(图 2)。构建系统进化树分析瘤背石磺CaM-like氨基酸序列的进化关系(图 3), 结果显示瘤背石磺与静水椎实螺(Lymnaea stagnalis)聚合为一支, 再与光滑双脐螺(Biomphalaria glabrata)聚合为一支, 这也与传统形态学的分类相吻合, 均为软体动物门腹足纲贝类, 体现了无脊椎动物在进化过程中CaM-like基因具有保守性。

|
图 1 瘤背石磺CaM-like基因的cDNA全长和预测的氨基酸序列 ORF框区域用双下划线显示; UTR区域用波浪线表示; 单下划线表示EFh家族保守序列; 字母加粗表示信号肽序列; 加尾信号序列AATAAA和Poly(A)尾巴用红色字体表示. Fig.1 The full-length cDNA sequence and predicted amino acid sequence of Ohchidium reevesii CaM-like gene The ORF region is indicated by double underline; the UTR region is represented by wavy line; the single underline indicates a conserved sequence of the EFh family; the bold portion indicates the signal peptide sequence; the tailing signal sequences AATAAA and Poly (A) tails are indicated in red font. |

|
图 2 瘤背石磺CaM-like氨基酸序列与其他无脊椎动物CaM-like氨基酸序列的多重比对 高度保守的氨基酸序列用黑色表示, 相似的氨基酸序列用粉红色表示. Fig.2 Multiple alignments of the amino acid sequences of Ohchidium reevesii CaM-like with other shellfish species The high conserved cysteine residues were indicated in black and similar residues were shown in pink. |

|
图 3 应用邻接法构建的基于瘤背石磺CaM-like与其他物种的CaM-like氨基酸序列的系统进化树 利用MEGA 6.0, 设定1000次自主重复, 其他参数使用默认值. Fig.3 The phylogenetic tree based on the amino acid sequence homology of CaM-like in Ohchidium reevesii and other species The value of bootstrap was set 1000 replications by MEGA 6.0, and the other parameters were set default values. |
以管家基因18S rRNA为内参[25], qRT-PCR检测CaM-like基因在不同组织中的分布(图 4)。结果显示CaM-like mRNA在不同组织都有分布, 但是在各组织中的表达量不同。CaM-like mRNA在瘤背石磺神经节部位的表达量最高, 显著高于其他组织(P < 0.05), 其次在背部皮肤、腹足和口器等组织中都有较高的表达量, 但背部皮肤、腹足和口器组织中的表达量差异不显著(P > 0.05)。
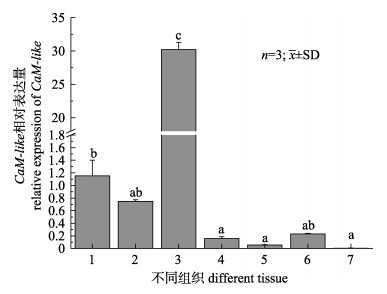
|
图 4 CaM-like mRNA在瘤背石磺不同组织中的表达情况 1:背部皮肤; 2:腹足; 3:神经节; 4:肠; 5:肝胰腺; 6:口器; 7:蛋白腺.不同字母表示差异显著(P < 0.05). Fig.4 Expression of CaM-like mRNA in different tissues of Ohchidium reevesii 1: dorsal skin; 2: pleopod; 3: ganglion; 4: intestine; 5: hepatopancreas; 6: mouthparts; 7: albumen gland. Different letters indicate significant difference (P < 0.05). |
实验中声音频率的设置模拟潮间带潮汐产生的低频声波(5~100 Hz)。以18S rRNA管家基因为内参基因, 荧光定量PCR检测神经节中的CaM-like基因在正弦波声波频率为0 Hz、5 Hz、25 Hz、50 Hz、75 Hz和100 Hz下分别连续刺激6.2 h、12.4 h和24 h时表达量, 在同一频率下对不同时间点的表达量进行差异显著性分析(图 5)。图 5显示声波频率25 Hz刺激下, 12.4 h时CaM-like基因在神经节中的表达量最高, 显著高于0 h、6.2 h和24 h时的表达量(P < 0.05);声波频率50 Hz刺激下, 12.4 h时CaM-like基因在神经节中的表达量最高, 显著高于0 h、6.2 h和24 h时的表达量(P < 0.05)。CaM-like基因在声波频率刺激下12.4 h的表达量随着声波频率的增大显现出先升高后下降的趋势, 且在25 Hz时表达量达到最高, 其次是在5 Hz和25 Hz时也有较高的表达量。
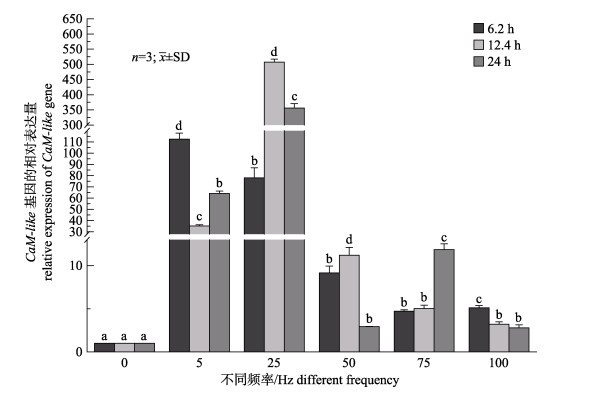
|
图 5 不同声波频率刺激下不同时间点CaM-like基因在瘤背石磺神经节中的表达量 不同字母表示在同一频率下不同时间点之间有显著差异(P < 0.05). Fig.5 CaM-like mRNA expression in ganglion of Ohchidium reevesii under different sound frequency stimulation Different letters indicate significant difference in CaM-like mRNA expression among different time under the same frequency (P < 0.05). |
在2.3数据分析结果的基础上选取刺激12.4 h这一时间点, 分析CaM-like基因的下游基因CaMKII基因的表达量。以18S rRNA管家基因为内参基因, 荧光定量PCR检测CaMKII基因在神经节中在正弦波声波频率为0 Hz、5 Hz、25 Hz、50 Hz、75 Hz和100 Hz刺激下在12.4 h的表达量(图 6)。CaMKII基因在神经节中的表达量随着声波频率的增大呈现出先升高后下降并趋于平稳。实验组中CaMKII基因表达量显著高于对照组(P < 0.05), 25 Hz时, CaMKII基因的表达量最高, 且显著高于其他实验组(P < 0.05), 其次在50 Hz时, CaMKII基因的表达量较高, 且显著高于声波频率为5 Hz、75 Hz和100 Hz的实验组(P < 0.05)。
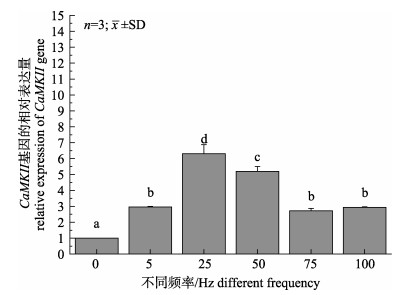
|
图 6 不同声波频率刺激下12.4 h CaMKII基因在瘤背石磺神经节中的表达量 不同字母表示有显著差异(P < 0.05). Fig.6 CaMKII mRNA expression in ganglion of Ohchidium reevesii at 12.4 h under different sound frequency stimulation Different letters indicate significant difference (P < 0.05). |
类钙调素因子(CaM-like)作为EF-hand超家族成员之一, 广泛分布真核生物中, 因具有多种生理功能而被广泛研究。本研究中由瘤背石磺神经节组织克隆出CaM-like基因的cDNA全长, 氨基酸序列比对构建系统进化树, 结果显示瘤背石磺与静水椎实螺(L. stagnalis)、滑双脐螺(B. glabrata)聚为一支, 说明瘤背石磺CaM-like基因与软体动物门腹足纲贝类的亲缘关系非常接近, 与林亚等[26]构建栉孔扇贝(Chlamys farreri)钙调素类似蛋白基因的进化关系相似, 体现出物种由低等到高等, 从海洋到陆地的进化过程。对CaM-like基因的进化关系的分析为研究物种进化奠定了基础。
瘤背石磺不同组织的qRT-PCR分析结果显示, CaM-like基因在神经节部位高表达, CaM-like基因可能参与神经系统信息传递, 具调节离子通道蛋白的构型。徐天乐等[27]在研究CaM在文昌鱼(Branchiostoma belcheri Gray)神经系统的分布情况时也发现, 神经中含有大量CaM-IR阳性物质, 可能是因为CaM参与神经系统中神经递质的合成和释放。王凤山等[28]研究铅对仔鼠CaM基因表达与学习记忆的关系时表明, 铅诱导下仔鼠悬崖回避实验完成率明显低于对照组, CaM基因表达量下降, 导致行动迟缓, 学习记忆能力下降。其次, CaM-like基因在瘤背石磺背部皮肤和腹足都有较高的表达, 这可能与瘤背石磺的肌肉收缩、快速运动逃避潮水淹没有关。Ryder等[29]用低频率的电刺激小鼠时发现CaM基因参与骨骼肌肌肉的发育, 对骨骼肌收缩具有明显的促进作用。qRT-PCR结果显示CaM-like基因在肠和肝胰腺都有一定的表达量, 这可能是与CaM-like蛋白参与瘤背石磺的先天免疫有关, 其长期栖息潮间带高潮区的环境中, 容易遭受病原微生物等的入侵和有害生物毒害。据有关报道[30], 利用LPS注射克氏原鳌虾(Procambarus clarkii)时, 肠、鳃和肝胰腺中的CaM-like基因在不同时间点的表达量有明显的变化, 这说明CaM-like基因在免疫反应中具有重要的作用。
栖息于潮间带的海洋动物, 其生理状态和行为很大程度受到潮汐周期的影响, 而一个半日潮的潮汐周期约为12.4 h[31]。本实验通过模拟潮汐低频声音频率范围, 实验室内刺激瘤背石磺, qRT-PCR结果显示25 Hz和50 Hz低频声音频率连续刺激12.4 h时, CaM-like基因和CaMKII基因的表达量较高, 均显著高于对照组(P < 0.05), 它们的表达量趋势几乎一致。这可能与瘤背石磺长期感知潮汐后, 形成12.4 h的潮汐节律有关。Sun等[32]在研究慢性多重应激对大鼠学习记忆的影响时, qRT-PCR结果显示应激组CaM和CaMKII mRNA的表达量均高于对照组, 表明CaM和CaMKII增强慢性多重应激对学习和记忆具有重要作用。在一些生物中, 从细菌到人类, 它们在进化过程中形成了24 h的节律[33]。然而在一些栖息于潮间带的海洋动物中主要表现为12.4 h的节律。如潮汐节律调节青蟹(Carcinus maenas)在夜间每隔12.4 h出现活动频繁的现象[34]。而栖息于潮间带的金刚蟹(Emerita talpoida), 从大眼幼体、仔虾、幼虾、性腺成熟的雄性和雌性等都具有12.4 h的潮汐节律, 在涨潮后1~2 h活动非常频繁[35]。齿轮帽贝(Cellana rota)同样也表现出12.4 h的潮汐节律, 在退潮后至涨潮期间活动频繁, 相关调控基因的表达量较高[36]。这与瘤背石磺在低频频率25 Hz和50 Hz连续刺激12.4 h后基因的表达量相对较高的结果一致。
4 结论本实验从瘤背石磺神经转录组数据库中获得CaM-like、CaMKII基因的部分片段, 利用RACE技术克隆出CaM-like基因的cDNA全长, qRT-PCR的结果表明CaM-like基因于所测的各组织中均有表达, 但主要于神经节部位高表达, 推测其可能参与神经系统的可塑性调节。潮汐低频声波25 Hz和50 Hz连续刺激12.4 h时, CaM-like、CaMKII基因在神经节的表达量较高。由此推测瘤背石磺能感知潮汐低频声波, 形成25~50 Hz的12.4 h半日潮潮汐周期节律, 产生潮汐记忆能力。这将为探索瘤背石磺感知潮汐节律的机制提供重要参考, 也为进一步理解海洋无脊椎动物对海洋环境适应的机理奠定良好基础。
| [1] |
Wang J Q, Cheng Y X, Wu X G, et al. Morphological characteristics, living habitus and reproductive behavior of Onchidium struma[J]. Chinese Journal of Zoology, 2005, 40(1): 32-40. [王金庆, 成永旭, 吴旭干, 等. 瘤背石磺的形态、习性和生殖行为[J]. 动物学杂志, 2005, 40(1): 32-40.] |
| [2] |
Wu X F, Shen H D, Wu W J, et al. Comparison on morphology of Onchidiidae in eastern coast of China[J]. Chinese Journal of Zoology, 2010, 45(6): 92-100. [吴旭峰, 沈和定, 吴文健, 等. 我国华东沿海4种石磺形态学比较[J]. 动物学杂志, 2010, 45(6): 92-100.] |
| [3] |
Arey L B, Crozier W J. The homing habits of the pulmonate Mollusk Onchidium[J]. Proceedings of the National Academy of Sciences of the United States of America, 1918, 4(11): 319-321. DOI:10.1073/pnas.4.11.319 |
| [4] |
Kang C K, Lee Y W, Choy E J, et al. Microphytobenthos seasonality determines growth and reproduction in intertidal bivalves[J]. Marine Ecology Progress Series, 2006, 315(8): 113-127. |
| [5] |
Zhang G S, Gu X X, Xing B B, et al. The classification and the impact of marine environment noise on marine animals[J]. Journal of Dalian Ocean University, 2012, 27(1): 89-94. [张国胜, 顾晓晓, 邢彬彬, 等. 海洋环境噪声的分类及其对海洋动物的影响[J]. 大连海洋大学学报, 2012, 27(1): 89-94. DOI:10.3969/j.issn.1000-9957.2012.01.018] |
| [6] |
Kaifu K, Segawa S, Tsuchiya K. Behavioral responses to underwater sound in the small benthic octopus Octopus ocellatus[J]. The Journal of the Marine Acoustics Society of Japan, 2007, 34(4): 266-273. DOI:10.3135/jmasj.34.266 |
| [7] |
Buscaino G, Filiciotto F, Gristina M, et al. Acoustic behavior of the European spiny lobster Alinurus elephas[J]. Marine Ecology Progress Series, 2011, 441: 177-184. |
| [8] |
Peng C, Zhao X G, Liu S X, et al. Effects of anthropogenic sound on digging behavior, metabolism, Ca2+/Mg2+ ATPase activity, and metabolism-related gene expression of the bivalve Sinonovacula constricta[J]. Scientific Reports, 2016, 6: 24266. DOI:10.1038/srep24266 |
| [9] |
Huang Z K, Chen Z Y. The response analyses of tides[J]. Journal of Shandong College of Oceanology, 1983, 13(2): 13-20. [黄祖珂, 陈宗镛. 潮汐响应分析[J]. 山东海洋学院学报, 1983, 13(2): 13-20.] |
| [10] |
Liu J, Huang J T. The relationship between Onchidium struma's spawning regularity and astronomical tides[J]. Journal of Shanghai Ocean University, 2018, 27(1): 73-78. [刘杰, 黄金田. 瘤背石磺产卵规律与天文潮汐关系[J]. 上海海洋大学学报, 2018, 27(1): 73-78.] |
| [11] |
Means A R, Chafouleas J G. Calmodulin in endocrine cells[J]. Annual Review of Physiology, 1982, 44(1): 667-682. |
| [12] |
Yabe D, Nakamura T, Kanazawa N, et al. Calumenin, a Ca2+-binding protein retained in the endoplasmic reticulum with a novel carboxyl-terminal sequence, HDEF[J]. Journal of Biological Chemistry, 1997, 272(29): 18232-18239. DOI:10.1074/jbc.272.29.18232 |
| [13] |
Cheung W. Calmodulin plays a pivotal role in cellular regulation[J]. Science, 1980, 207(4426): 19-27. DOI:10.1126/science.6243188 |
| [14] |
Means A R, Dedman J R. Calmodulin-an intracellular calcium receptor[J]. Nature, 1980, 285(5760): 73-77. DOI:10.1038/285073a0 |
| [15] |
Caceres A, Bender P, Snavely L, et al. Distribution and subcellular localization of calmodulin in adult and developing brain tissue[J]. Neuroscience, 1983, 10(2): 449-461. DOI:10.1016/0306-4522(83)90145-8 |
| [16] |
Ikura M, Osawa M, Ames J B. The role of calcium-binding proteins in the control of transcription:Structure to function[J]. BioEssays, 2002, 24(7): 625-636. DOI:10.1002/bies.10105 |
| [17] |
Kasri N N, Sienaert I, Parys J B, et al. A novel Ca2+-induced Ca2+ release mechanism in A7r5 cells regulated by calmodulin-like proteins[J]. Journal of Biological Chemistry, 2003, 278(30): 27548-27555. DOI:10.1074/jbc.M302026200 |
| [18] |
Li S, Xie L P, Ma Z J, et al. cDNA cloning and characterization of a novel calmodulin-like protein from pearl oyster Pinctada fucata[J]. FEBS Journal, 2005, 272(19): 4899-4910. DOI:10.1111/j.1742-4658.2005.04899.x |
| [19] |
Rudd J J, Franklin-Tong V E. Unravelling response-specificity in Ca2+signalling pathways in plant cells[J]. New Phytologist, 2001, 151(1): 7-33. DOI:10.1046/j.1469-8137.2001.00173.x |
| [20] |
Soderling T R. Structure and regulation of calcium/calmodulin-dependent protein kinases Ⅱ and Ⅳ[J]. Biochimica et Biophysica Acta (BBA)-Protein Structure and Molecular Enzymology, 1996, 1297(2): 131-138. DOI:10.1016/S0167-4838(96)00105-7 |
| [21] |
Rongo C. A fresh look at the role of CaMKII in hippocampal synaptic plasticity and memory[J]. BioEssays, 2002, 24(3): 223-233. DOI:10.1002/bies.10057 |
| [22] |
Li G, Laabich A, Liu L O, et al. Molecular cloning and sequence analyses of calcium/calmodulin-dependent protein kinase Ⅱ from fetal and adult human brain-Sequence analyses of human brain calcium/calmodulin-dependent protein kinase Ⅱ[J]. Molecular Biology Reports, 2001, 28(1): 35-41. |
| [23] |
Pastalkova E, Serrano P, Pinkhasova D, et al. Storage of spatial information by the maintenance mechanism of LTP[J]. Science, 2006, 313(5790): 1141-1144. DOI:10.1126/science.1128657 |
| [24] |
Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [25] |
Yang T Z, Shen H D, Shi Y M, et al. Molecular cloning and characterization of EGF-like gene in Onchidium struma[J]. Marine Sciences, 2017, 41(3): 8-16. [杨铁柱, 沈和定, 史艳梅, 等. 瘤背石磺表皮生长因子基因的克隆、结构及进化分析[J]. 海洋科学, 2017, 41(3): 8-16.] |
| [26] |
Lin Y, Li S G, Xie L P, et al. Molecular cloning of calmodulin-like protein gene CaLP and comparison with expression profiles of CaM gene in scallop Chlamys farreri[J]. Fisheries Science, 2014, 33(11): 692-701. [林亚, 李世国, 谢莉萍, 等. 栉孔扇贝钙调素类似蛋白基因的克隆及其与钙调素基因表达特征的比较分析[J]. 水产科学, 2014, 33(11): 692-701. DOI:10.3969/j.issn.1003-1111.2014.11.006] |
| [27] |
Xu T L, Huang W Q, Su H C, et al. Distribution of calmodulin in the nervous system of Branchiostoma belcheri Gray[J]. Chinese Science Bulletin, 1993, 38(7): 650-652. [徐天乐, 黄威权, 苏慧慈, 等. 钙调素在文昌鱼神经系统中的分布[J]. 科学通报, 1993, 38(7): 650-652.] |
| [28] |
Wang F S, Liu Z H, Zhang J S, et al. Effects of lead exposure on protein kinase C and calmodulin expression in hippocampus of baby-rats[J]. Chinese Journal of Industrial Hygiene and Occupational Diseases, 2008, 26(3): 465-467. [王凤山, 刘忠慧, 张静姝, 等. 铅对仔鼠海马蛋白激酶C和钙调蛋白基因表达与学习记忆的影响[J]. 中华劳动卫生职业病杂志, 2008, 26(3): 465-467.] |
| [29] |
Ryder J W, Lau K S, Kamm K E, et al. Enhanced skeletal muscle contraction with myosin light chain phosphorylation by a calmodulin-sensing kinase[J]. Journal of Biological Chemistry, 2007, 282(28): 20447-20454. DOI:10.1074/jbc.M702927200 |
| [30] |
Zhu B J, Yu Y Y, Gao J, et al. Characterization and function of a novel calmodulin-like protein from crayfish, Procambarus clarkii[J]. Fish & Shellfish Immunology, 2017, 67: 518-522. |
| [31] |
Han Z Q. Analysis of the causes and laws of tidal phenomenon[J]. Journal of Physics Teaching, 2010, 28(2): 27-28. [韩忠全. 潮汐现象的成因及规律分析[J]. 物理教学探讨, 2010, 28(2): 27-28.] |
| [32] |
Sun C Y, Qi S S, Lou X F, et al. Changes of learning, memory and levels of CaMKII, CaM mRNA, CREB mRNA in the hippocampus of chronic multiple-stressed rats[J]. Chinese Medical Journal, 2006, 119(2): 140-147. DOI:10.1097/00029330-200601020-00008 |
| [33] |
Dunlap J C. Molecular bases for circadian clocks[J]. Cell, 1999, 96(2): 271-290. |
| [34] |
Tessmar-Raible K, Raible F, Arboleda E. Another place, another timer:Marine species and the rhythms of life[J]. BioEssays, 2011, 33(3): 165-172. DOI:10.1002/bies.201000096 |
| [35] |
Forward R B, Diaz H, Cohen J H. The tidal rhythm in activity of the mole crab Emerita talpoida[J]. Journal of the Marine Biological Association of the United Kingdom, 2005, 85(4): 895-901. DOI:10.1017/S0025315405011860 |
| [36] |
Schnytzer Y, Simon-Blecher N, Li J, et al. Tidal and diel orchestration of behaviour and gene expression in an intertidal mollusc[J]. Scientific Reports, 2018, 8: 4917. DOI:10.1038/s41598-018-23167-y |
 2020, Vol. 27
2020, Vol. 27

