2. 中国环境监测总站, 北京 100012
2. China National Environmental Monitoring Centre, Beijing 100012, China
辽宁省濒临黄渤海, 大陆海岸线长2110 km, 近海水域面积6.8万km2, 近海生物资源丰富, 有浮游、底栖、游泳3大类生物500余种, 形成了辽东湾渔场、海洋岛渔场等诸多天然渔场。然而, 随着近些年人为活动的加剧, 如大规模捕捞、沿海基建、滩涂养殖、围圈养殖、工农业和生活排污入海等, 辽宁沿海的生态环境和生物种群多样性受到了一定影响。因此, 通过沿海生物野生种群的遗传特征研究, 有助于了解辽宁沿海生态环境的发展水平。目前, 学者已开展了一系列相关研究, 物种涉及鱼类[1]、贝类[2-4]、甲壳类[5]和其他种类[6]。在以上研究物种中, 由于贝类的移动性相对较差, 因此更适合于研究不同地理群体的遗传多样性和遗传分化, 目前已研究的贝类主要为毛蚶(Scapharca subcrenata)[4]、香螺(Neptunea cumingi)[3]、长牡蛎(Crassostrea gigas)[7]和蛤仔(Ruditapes philippinarum)[2]等具有较高经济价值的物种。为了减少研究物种受人为采捕和增养殖活动的影响, 体现自然野生物种的遗传特征, 本研究选取了在辽宁沿海广泛分布且经济价值较低的贝类——托氏琩螺(Umbonium thomasi)。托氏琩螺属软体动物门(Mollusca)、腹足纲(Gastropoda)、钟螺目(Trochida)、马蹄螺科(Trochidae)、琩螺属, 俗称玻璃牛或海钱, 个体较小, 贝壳表面有波纹状花纹。托氏琩螺栖息于河口区沙滩或泥沙滩, 密度较大, 因其体型较小、采捕费时, 因此较少利用, 经济价值较低。关于托氏琩螺的已有研究报道主要为生态资源调查, 集中于辽宁[8]、山东[9]和江苏[10]沿海, 未见遗传特征研究。
传统的生物群体遗传特征分析方法主要为分子标记法, 包括AFLP、SSR、RAPD、ISSR和SNP标记等。在本研究中, 采用了基于简化基因组测序的基因分型技术(genotyping-by-sequencing, GBS)[11], 在基因组水平开发SNP标记, 分析种群遗传特征, 具有更高的覆盖度和准确性。GBS是基于第二代高通量测序的简化基因组分析技术, 通过限制性内切酶对基因组进行酶切, 测序分析后获得基因组水平的高密度SNP标记, 通过几十万甚至上百万个SNP标记进行遗传特征分析[12]。目前, GBS技术已在遗传学研究、图谱构建和种质鉴定等领域广泛应用[13-14]。
本研究利用GBS技术对辽宁黄渤海沿岸的5个托氏琩螺自然群体进行遗传多样性和遗传分化研究, 以期揭示沿海非经济物种的遗传多样性水平和种群遗传分化特征, 为辽宁沿海的自然资源开发和保护利用提供依据。
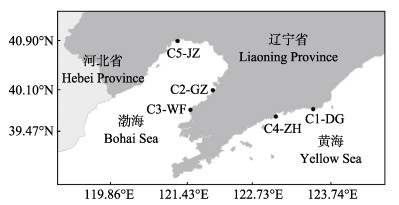
1 材料与方法 1.1 样品采集本研究于2016年9—10月于辽宁省黄渤海沿岸采集托氏琩螺群体样品(图 1), 包括5个采样群体: C1-DG群体(东港市沿岸: 39.78°N, 123.56°E)、C2-GZ群体(盖州市沿岸: 40.12°N, 121.99°E)、C3-WF群体(瓦房店市沿岸: 39.78°N, 121.49°E)、C4-ZH群体(庄河市沿岸: 39.71°N, 123.27°E)、C5-JZ群体(锦州市沿岸: 40.89°N, 121.20°E)。采集方法为人工潮间带捡拾。每个群体随机选取30个个体, 取足部肌肉组织于95%乙醇中, 带回辽宁省海洋水产科学研究院保存备用。
|
图 1 辽宁黄渤海沿岸5个托氏琩螺群体样品的采集地点 C1-DG为东港市沿岸采样群体, C2-GZ为盖州市沿岸采样群体, C3-WF为瓦房店市沿岸采样群体, C4-ZH为庄河市沿岸采样群体, C5-JZ为锦州市沿岸采样群体. Fig.1 Sampling locations of the five Umbonium thomasi populations along the Yellow Sea and Bohai Sea in Liaoning Province C1-DG indicates the population from Donggang coast; C2-GZ indicates the population from Gaizhou coast; C3-WF indicates the population from Wafangdian coast; C4-ZH indicates the population from Zhuanghe coast; C5-JZ indicates the population from Jinzhou coast. |
利用海洋动物组织基因组提取试剂盒(天根生化, 北京)提取足部肌肉组织的基因组DNA。纯化后的DNA样品经琼脂糖凝胶电泳检测和OD260/OD280检测合格。由于已有研究表明适量的个体混合不会对研究结果造成影响[15], 因此为了便于分析和降低检测成本, 在前期预实验发现不会明显降低SNP获取水平的前提下, 将个体DNA进行等量混样, 每个群体利用4~5个混合DNA样品进行后续分析。采用限制性内切酶Hind Ⅲ+Bfa I对全基因组DNA进行完全酶切, 回收插入片段长度220~450 bp。按照Double Digest Genotyping-by-Sequencing (dd-GBS)方法进行建库。
1.2.2 测序和数据质控采用Illumina Hiseq2000平台, 对dd-GBS文库进行测序, 测序方式为双末端测序(paired-end, 2×150 bp)。对原始下机数据(raw data)进行数据的质控过滤, 质控标准如下: (1)数据质量评估:利用FastQC对碱基质量分布、base content分布、GC content分布和序列平均质量分布进行检测, 确保数据质量满足分析要求; (2)酶切位点识别:若出现双末端reads中read-1的5′端前6 bp非酶切位点序列TGCAG或read-2的5′端前4 bp非酶切位点序列CGG, 则去除该双末端reads; (3)接头污染去除:采用AdapterRemoval软件(version 2)去除3′端的接头污染。
1.3 统计分析 1.3.1 标签聚类对于数据质控获得的高质量序列, 利用stacks软件中的cstacks程序对每个样品reads进行聚类(参数设置-m 4)。聚类后的同一个stack代表 1个酶切位点, 称为1个标签。对每一个样品的标签及其测序深度进行统计, 确保符合分析要求。
1.3.2 SNP检测利用cstacks程序将所有样品的标签合并, 获得每个标签的catalog consensus序列(参数设置-n 2)。利用sstacks程序将每个样品的标签序列与catalog consensus序列进行比对。最后利用populations程序过滤并输出所有样品的SNP位点(参数设置-p 1; -r 0.50)。
1.3.3 群体遗传学分析利用stacks软件中的populations程序进行群体遗传学分析。利用PHYLIP软件的NJ和UPGMA算法构建进化树(P-distance; bootstrap: 1000)。利用GCTA软件进行主成分分析(principal component analysis, PCA)(MAF≥0.05)。利用structure软件进行群体遗传结构分析。
2 结果与分析 2.1 基础数据GBS测序共得到测序数据39.30 Gb, 其中Q20 > 93%, Q30 > 83%。经数据质控过滤后, 得到高质量reads 271640516条, 高质量数据33.03 Gb, 平均每个样品数据量1.38 Gb, 质控后的数据质量满足数据分析需要(表 1)。Reads聚类后得到标签共9919784个, 平均测序深度16.7。标签序列经比对过滤后共得到SNP位点1315987个。
|
|
表 1 质控后数据统计和标签聚类情况 Tab.1 Data statistics after quality control and clustering tags |
根据得到的SNP位点, 对5个托氏琩螺群体的观测杂合度(Ho)、观测纯合度(OH)、期望杂合度(He)、期望纯合度(EH)、核苷酸多样性(Pi)和群体近交系数(Fis)进行分析, 从而评价不同群体的遗传多样性。结果表明, 5个托氏琩螺群体的观测杂合度为0.0851~0.1161, 期望杂合度为0.1424~0.1627, 观测纯合度为0.8839~0.9149, 期望纯合度为0.8373~0.8576(表 2)。5个托氏琩螺群体的观测杂合度均低于期望杂合度。在5个群体中, C4-ZH群体的杂合度处于较低水平, 纯合度处于较高水平, 同时核苷酸多样性在5个群体中较低, 群体近交系数在5个群体中最高。C2-GZ和C5-JZ群体的杂合度在5个群体中处于较高水平, 纯合度处于较低水平。
|
|
表 2 辽宁黄渤海沿岸5个托氏琩螺群体的遗传多样性 Tab.2 Genetic diversity of five Umbonium thomasi populations along the Yellow Sea and Bohai Sea in Liaoning Province |
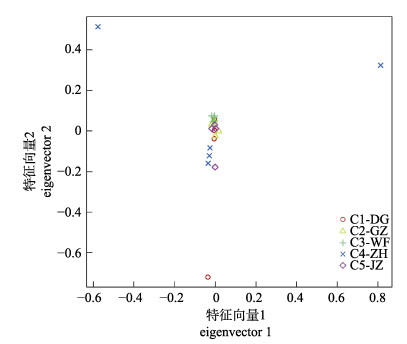
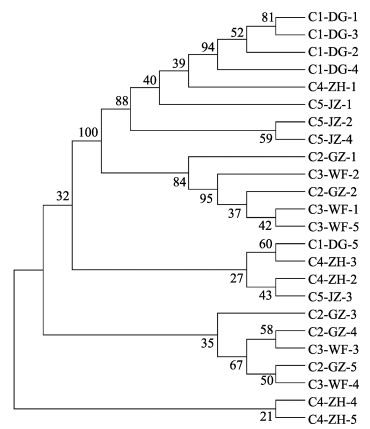
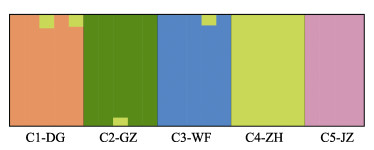
为了研究5个托氏琩螺群体的遗传分化程度, 我们首先进行了主成分分析(图 2), 结果发现除个别样品因统计误差偏离较大以外, 大部分样品都聚为一处, 未发现明显的遗传分化。基于不同群体间的遗传分化指数(Fst), 本研究发现5个托氏琩螺群体间的分化指数为0.0933~0.1183(表 3), 其中C5-JZ群体与其他4个群体的分化指数均处于相对较高水平, 为0.1129~0.1183, 基因流(Nm) < 2, C2-GZ和C3-WF群体的分化指数最低(0.0933~0.1183)。该结果与系统进化树的分析结果较为一致, 其中C5-JZ群体趋向于聚为一支, C2-GZ和C3-WF群体趋向于聚在一起(图 3)。为了验证以上结果, 又进行了群体遗传结构分析(图 4), 结果发现, 5个托氏琩螺群体可以较好地分为5个亚群, 其中C1-DG、C2-GZ、C3-WF和C4-ZH群体之间有少量基因交流, 而C5-JZ群体则与其他4个群体间隔开。
|
图 2 辽宁黄渤海沿岸5个托氏琩螺群体样品的主成分分析 C1-DG为东港市沿岸采样群体, C2-GZ为盖州市沿岸采样群体, C3-WF为瓦房店市沿岸采样群体, C4-ZH为庄河市沿岸采样群体, C5-JZ为锦州市沿岸采样群体. Fig.2 Principal component analysis of the five Umbonium thomasi populations along the Yellow Sea and Bohai Sea in Liaoning Province C1-DG indicates the population from Donggang coast; C2-GZ indicates the population from Gaizhou coast; C3-WF indicates the population from Wafangdian coast; C4-ZH indicates the population from Zhuanghe coast; C5-JZ indicates the population from Jinzhou coast. |
|
|
表 3 辽宁黄渤海沿岸5个托氏琩螺群体的遗传分化与基因流 Tab.3 Genetic differentiations and gene flow of the five Umbonium thomasi populations along the Yellow Sea and Bohai Sea in Liaoning Province |
|
图 3 辽宁黄渤海沿岸5个托氏琩螺群体样品的系统进化树 C1-DG为东港市沿岸采样群体, C2-GZ为盖州市沿岸采样群体, C3-WF为瓦房店市沿岸采样群体, C4-ZH为庄河市沿岸采样群体, C5-JZ为锦州市沿岸采样群体. Fig.3 Phylogenetic tree of the samples from the five Umbonium thomasi populations along the Yellow Sea and Bohai Sea in Liaoning Province C1-DG indicates the population from Donggang coast; C2-GZ indicates the population from Gaizhou coast; C3-WF indicates the population from Wafangdian coast; C4-ZH indicates the population from Zhuanghe coast; C5-JZ indicates the population from Jinzhou coast. |
|
图 4 辽宁黄渤海沿岸5个托氏琩螺群体的遗传结构分析 C1-DG为东港市沿岸采样群体, C2-GZ为盖州市沿岸采样群体, C3-WF为瓦房店市沿岸采样群体, C4-ZH为庄河市沿岸采样群体, C5-JZ为锦州市沿岸采样群体. Fig.4 Genetic structure analysis of the five Umbonium thomasi populations along the Yellow Sea and Bohai Sea in Liaoning Province C1-DG indicates the population from Donggang coast; C2-GZ indicates the population from Gaizhou coast; C3-WF indicates the population from Wafangdian coast; C4-ZH indicates the population from Zhuanghe coast; C5-JZ indicates the population from Jinzhou coast. |
基于以上遗传分化研究结果发现, 在本研究的5个托氏琩螺群体中, C5-JZ群体的遗传分化较大。因此, 我们利用所有SNP位点的野生型/突变型在不同群体中的特异性对SNP进行了筛选, 获得了3个C5-JZ群体的特异性SNP位点(表 4), 它们在C5-JZ群体中的突变基因型比例均≥75%, 而在其他群体中突变基因型比例均≤25%。这3个SNP位点可以作为C5-JZ群体的特征SNP, 用于今后托氏琩螺的群体遗传研究。
|
|
表 4 辽宁黄渤海沿岸托氏琩螺C5-JZ群体的特征性SNP标记 Tab.4 SNP markers for Umbonium thomasi C5-JZ populatios along the Yellow Sea and Bohai Sea in Liaoning Province |
遗传特征研究主要包括遗传多样性分析和遗传分化分析等, 研究方法主要有形态学标记、细胞学标记、生化标记和分子标记等, 其中分子标记最为常用, 因其具有检测方便、数量大和多态性高等优点。传统分子标记方法主要有AFLP、SSR、RAPD、ISSR标记, 以及进化保守单基因(如COI、16s rRNA)的SNP标记等。传统分子标记方法技术成熟, 支持文献丰富, 是生物遗传特征研究的主要手段。然而, 传统分子标记方法也存在不足, 如实验过程繁琐, 所选标记在基因组中的覆盖度低等, 不同分析方法的研究结果无法进行横向比较的情况时有发生。本研究所采用的GBS技术使得在全基因组水平上筛选高密度SNP成为可能。通过对全基因组进行酶切、测序和分析, 本研究共获得了1315987个SNP。由于简化基因组分析所获得的SNP在基因组上的分布具有高密度性、均匀性等特点[15], 因此本研究结果相比传统分析方法, 能够更加全面、准确地揭示研究物种的遗传特征, 并为今后相关研究的开展提供参考。
3.2 5个托氏琩螺群体的遗传多样性托氏琩螺经济价值较低, 国内尚未见专题科研报道, 已有报道主要出现于生物资源调查中。韩国学者对其进行了一定研究, 主要包括线粒体序列、性腺发育和繁殖特征等方面[16-17]。托氏琩螺是中国北方沿海潮间带的常见优势物种, 因受人为采捕和增养殖影响较小, 相对于其他经济物种, 其遗传多样性能够更好地反映沿海非经济物种的多样性水平。在遗传多样性评价指标中, 杂合度是生物群体在单个或多个基因位点上的遗传变异水平, 是评价群体遗传多样性的关键指标。学者已对辽宁沿海贝类的遗传多样性开展了研究, 主要集中于贝类经济物种。隋娜[3]利用SSR分子标记对黄渤海沿岸5个香螺(Neptunea cumingi Crosse)地理群体的遗传多样性进行了研究, 发现其观测杂合度为0.173~0.865, 期望杂合度为0.230~0.676;赵文等[4]利用ISSR分子标记对辽宁沿海5个毛蚶(Scapharca subcrenata)群体的遗传多样性进行了研究, 发现平均杂合度为0.3092~0.3301;牛泓博[2]对辽宁沿海8个蛤仔群体的遗传多样性进行了研究, 发现观测杂合度和期望杂合度分别为0.163~0.922和0.298~0.882。本研究中5个托氏琩螺群体的观测杂合度为0.0851~0.1161, 期望杂合度为0.1424~0.1627, 在已有研究物种中处于较低水平。张国范等[18]指出, 与其他海洋无脊椎动物比较, 贝类遗传多样性较高, 平均杂合度常在0.15以上, 即使按此标准, 本研究的杂合度水平依然较低。作者推测导致杂合度水平偏低的主要原因是本研究的分析方法与传统分子标记有较大不同, 由于传统分子标记方法只选择少数的标记位点作为全基因组多样性水平的代表, 因此与基于基因组水平的简化基因组分析结果无法直接比较。于思梦等[7]利用简化基因组技术研究了辽宁沿海2个长牡蛎群体的遗传多样性, 发现其多样性水平明显低于已有长牡蛎的其他研究; 翟正晓[15]利用简化基因组技术分析了16个鸡(Gallus gallus)品种的遗传多样性, 发现杂合度与之前报道的中国地方鸡品种相比明显偏低, 认为可能是与传统分子标记所选用的遗传标记数量太少有关。尽管利用GBS和传统分子标记方法得出的遗传多样性分析结果不具有很好的可比性, 但是由于GBS分析所选用的遗传标记数量多, 能够覆盖全基因组, 因此具有更高的准确性和稳定性, 可能是将来遗传多样性研究手段发展的方向, 同时本研究结果也将为今后遗传多样性研究的多元化提供参考。
本研究中5个托氏琩螺群体的观测杂合度均低于期望杂合度, 同时群体近交系数为正值, 说明本研究中辽宁沿海5个托氏琩螺群体均出现杂合子缺失现象。生物群体的杂合子缺失会导致生物多样性的降低, 从而减弱群体对环境的适应能力, 一些贝类研究中同样发现了杂合子缺失, 但这是否会导致贝类种质衰退, 还需要进行长期的监测和对比分析。常亚青等[19]和柳明等[20]分别对多个虾夷扇贝(Patinopecten yessoensis)和大珠母贝(Pinctada maxima)群体的群体多样性进行了分析, 发现均存在普遍杂合子缺失, 建议采取有效措施保护种质资源。在5个托氏琩螺群体中, C4-ZH群体的遗传多样性水平较低, 可能是由于种群规模过小导致的。笔者在采样时发现, C4-ZH群体在潮间带的分布数量和密度均较低, 同时发现了一定规模的泥螺(Bullacta exarata)群体。吴文广等[21]认为, 泥螺在一定区域内的大量增殖, 将通过争夺溶解氧和大量排氨来影响托氏琩螺的种群数量。黄渤海多处沿海地区已发现托氏琩螺等优势物种已逐渐被外来物种泥螺所取代[22-23]。因此作者推测, 可能是由于泥螺的过度繁殖导致托氏琩螺C4-ZH群体规模和多样性水平的降低。此外本研究还发现, 位于红沿河核电站附近的C3-WF群体的遗传多样性在5个群体中处于中间水平, 说明托氏琩螺C3-WF群体的遗传多样性未受到人为活动的明显影响。
3.3 黄渤海沿岸托氏琩螺群体的遗传分化由于贝类移动能力较弱, 相较于鱼类等其他大范围活动的物种, 贝类更易产生遗传分化。在本研究中, 基于PCA分析, 并未发现明显的遗传分化现象, 这与遗传分化指数分析结果一致, 5个托氏琩螺群体的分化指数为0.0933~0.1183, 处于中等程度的遗传分化水平(0.05≤Fst≤0.15)[24]。遗传分化可能由长期缺乏基因交流产生, 也可能与人为活动或遗传漂变有关[25-27]。已报道的辽宁沿海香螺、毛蚶和蛤仔的不同地理群体的分化指数分别为0.006~0.188[3]、0.0634~0.1866[4]和0.115~0.364[2]。本研究中5个托氏琩螺群体的遗传分化在已有研究中处于较低水平, 出现了一定程度的分化, 但是还未形成明显的种群地理隔离。
基于分化指数和系统进化分析, 本研究发现C5-JZ群体相对更加独立, C2-GZ和C3-WF群体遗传分化水平相对较低。C5-JZ群体的采样地点位于养殖滩涂上, 日常有专人看管, 形成了一个相对独立的区域, 而其他4个采样点均为开放式, 这可能是造成C5-JZ群体遗传分化更加明显的主要原因。C2-GZ和C3-WF群体的采样地点均位于渤海东岸, 且都有人类活动和渔船作业, 这可能是C2-GZ和C3-WF群体遗传分化水平相对较低的原因。在群体遗传结构分析中发现, C1-DG、C2-GZ、C3-WF和C4-ZH群体存在基因交流, 且都是由C4-ZH群体向C1-DG、C2-GZ、C3-WF群体中渗入, 其中的具体原因目前还不明确。
综上, 本研究利用GBS技术分析了辽宁黄渤海沿岸5个托氏琩螺自然群体的遗传多样性水平和遗传分化特征, 发现5个群体均存在杂合子缺失现象, 并形成中等程度的种群遗传分化。研究揭示了辽宁黄渤海沿岸托氏琩螺部分自然群体的遗传水平, 研究结果有助于了解辽宁沿海非经济物种的多样性水平, 为自然资源的保护和利用提供依据。
| [1] |
Peng S M, Shi Z H, Hou J L, et al. Genetic diversity of silver pomfret (Pampus argenteus) populations from the China Sea based on mitochondrial DNA control region sequences[J]. Biochemical Systematics and Ecology, 2009, 37(5): 626-632. DOI:10.1016/j.bse.2009.09.003 |
| [2] |
Niu H B. Investigation the germplasm resources on wild group of Ruditapes philippinarum in Liaoning coastal[D]. Dalian: Dalian Ocean University, 2014: 1-44. [牛泓博.辽宁沿海蛤仔群体种质资源调查研究[D].大连: 大连海洋大学, 2014: 1-44.]
|
| [3] |
Sui N. The analysis of the genetic diversity of different populations of Neptunea cumingi Crosse using SSR marker[D]. Dalian: Liaoning Normal University, 2008: 1-57. [隋娜.利用微卫星标记分析不同地理亚群香螺群体的遗传多样性[D].大连: 辽宁师范大学, 2008: 1-57.] http://cdmd.cnki.com.cn/Article/CDMD-10165-2009122025.htm
|
| [4] |
Zhao W, Zhang L, Bi J H. ISSR analyses of genetic diversity within and among five stocks of Scapharca subcrenata alone Liaoning coast[J]. Journal of Fisheries of China, 2011, 35(6): 854-862. [赵文, 张雷, 毕进红. 辽宁沿海5个毛蚶群体遗传多样性的ISSR分析[J]. 水产学报, 2011, 35(6): 854-862.] |
| [5] |
Li Y L, Kong X Y, Yu Z N, et al. Genetic diversity and historical demography of Chinese shrimp Feneropenaeus chinensis in Yellow Sea and Bohai Sea based on mitochondrial DNA analysis[J]. African Journal of Biotechnology, 2009, 8(7): 1193-1202. |
| [6] |
Dong Z, Liu Z, Liu D, et al. Low genetic diversity and lack of genetic structure in the giant jellyfish Nemopilema nomurai in Chinese coastal waters[J]. Marine Biology Research, 2016, 12(7): 769-775. DOI:10.1080/17451000.2016.1196818 |
| [7] |
Yu S M, Gao L, Wang W, et al. Genetic diversity of oyster in the Yellow Sea and Bohai Sea in Liaoning Province[J]. Journal of Economic Animal, 2017, 21(4): 215-220. [于思梦, 高磊, 王伟, 等. 辽宁黄渤海沿岸长牡蛎遗传多样性分析[J]. 经济动物学报, 2017, 21(4): 215-220.] |
| [8] |
Zhang A G, Yuan X T, Yang X L, et al. Temporal and spatial distributions of intertidal macrobenthos in the sand flats of the Shuangtaizi Estuary, Bohai Sea in China[J]. Acta Ecologica Sinica, 2016, 36(3): 172-179. DOI:10.1016/j.chnaes.2016.04.003 |
| [9] |
Wang X C, Li X Z, Wang H F, et al. Macrobenthic ecology of the intertidal zones of Chajiandao, Dakouhedao and Wangzidao of Yellow River Estuary in autumn[J]. Chinese Journal of Zoology, 2008, 43(6): 77-82. [王晓晨, 李新正, 王洪法, 等. 黄河口岔尖岛、大口河岛和望子岛潮间带秋季大型底栖动物生态学调查[J]. 动物学杂志, 2008, 43(6): 77-82. DOI:10.3969/j.issn.0250-3263.2008.06.011] |
| [10] |
Zhou H X, Liu J E, Qin P. Impacts of an alien species (Spartina alterniflora) on the macrobenthos community of Jiangsu coastal inter-tidal ecosystem[J]. Ecological Engineering, 2009, 35(4): 521-528. DOI:10.1016/j.ecoleng.2008.06.007 |
| [11] |
Elshire R J, Glaubitz J C, Sun Q, et al. A robust, simple genotyping-by-sequencing (GBS) approach for high diversity species[J]. PLoS ONE, 2011, 6(5): e19379. DOI:10.1371/journal.pone.0019379 |
| [12] |
Nimmakayala P, Levi A, Abburi L, et al. Single nucleotide polymorphisms generated by genotyping by sequencing to characterize genome-wide diversity, linkage disequilibrium, and selective sweeps in cultivated watermelon[J]. BMC Genomics, 2014, 15(1): 767. DOI:10.1186/1471-2164-15-767 |
| [13] |
Lin M, Cai S B, Wang S, et al. Genotyping-by-sequencing (GBS) identified SNP tightly linked to QTL for pre-harvest sprouting resistance[J]. Theoretical and Applied Genetics, 2015, 128(7): 1385-1395. DOI:10.1007/s00122-015-2513-1 |
| [14] |
Bajaj D, Das S, Badoni S, et al. Genome-wide high-throughput SNP discovery and genotyping for understanding natural (functional) allelic diversity and domestication patterns in wild chickpea[J]. Scientific Reports, 2015, 5: 12468. DOI:10.1038/srep12468 |
| [15] |
Zhai Z X. SNP mapping and population genetic analyses for 13 Chinese indigenous chicken breeds using rad sequencing[D]. Shanghai: Shanghai Jiao Tong University, 2014: 1-67. [翟正晓.基于RAD简化基因组测序技术的13种中国地方优良鸡品种SNPs多态性图谱构建及群体遗传学分析[D].上海: 上海交通大学, 2014: 1-67.] http://cdmd.cnki.com.cn/Article/CDMD-10248-1015029011.htm
|
| [16] |
Lee J H. Gonadal development and reproductive cycle of the sand snail, Umbonium thomasi[J]. Korean Journal of Fisheries and Aquatic Sciences, 2002, 35(6): 702-708. DOI:10.5657/kfas.2002.35.6.702 |
| [17] |
Kim H, Yu C, Kim H J, et al. The complete mitochondrial genome of a marine gastropod:Umbonium thomasi (Trochida, Trochidae)[J]. Mitochondrial DNA Part B, 2019, 4(1): 37-38. DOI:10.1080/23802359.2018.1535864 |
| [18] |
Zhang G F, Liu X, Que H Y, et al. The theory and application of hybridization and heterosis in marine mollusks[J]. Marine Sciences, 2004, 28(7): 54-60. [张国范, 刘晓, 阙华勇, 等. 贝类杂交及杂种优势理论和技术研究进展[J]. 海洋科学, 2004, 28(7): 54-60. DOI:10.3969/j.issn.1000-3096.2004.07.013] |
| [19] |
Chang Y Q, Chen X X, Ding J, et al. Genetic diversity in five scallop populations of the Japanese scallop (Patinopecten yessoensis)[J]. Acta Ecologica Sinica, 2007, 27(3): 1145-1152. [常亚青, 陈晓霞, 丁君, 等. 虾夷扇贝(Patinopecten yessoensis)5个群体的遗传多样性[J]. 生态学报, 2007, 27(3): 1145-1152. DOI:10.3321/j.issn:1000-0933.2007.03.040] |
| [20] |
Liu M, Yu D H, Huang G J, et al. Genetic variation in Pinctada maxima populations of different year-class from Sanya, Hainan, China[J]. South China Fisheries Science, 2011, 7(1): 26-31. [柳明, 喻达辉, 黄桂菊, 等. 中国海南三亚大珠母贝不同年代种群的遗传变异研究[J]. 南方水产科学, 2011, 7(1): 26-31. DOI:10.3969/j.issn.2095-0780.2011.01.004] |
| [21] |
Wu W G, Zhang J H, Fang J G, et al. Effects of salinity on oxygen consumption and ammonia excretion rate of different sizes of Bullacta exarata[J]. Chinese Journal of Ecology, 2013, 32(9): 2457-2461. [吴文广, 张继红, 方建光, 等. 盐度对不同规格泥螺耗氧率和排氨率的影响[J]. 生态学杂志, 2013, 32(9): 2457-2461.] |
| [22] |
Tang B P, Zhang D Z, Ge B M, et al. Sustainable utilization of biological resources from coastal wetlands in China[J]. Chinese Science Bulletin, 2013, 58(19): 2270-2275. DOI:10.1007/s11434-012-5354-1 |
| [23] |
Song L, Wang N B, Yang G J, et al. The stress response of biological communities in China's Yalu River Estuary and neighboring waters[J]. Acta Ecologica Sinica, 2013, 33(9): 2790-2802. [宋伦, 王年斌, 杨国军, 等. 鸭绿江口及邻近海域生物群落的胁迫响应[J]. 生态学报, 2013, 33(9): 2790-2802.] |
| [24] |
Tang X T, Ji Y, Chang Y W, et al. Population genetic structure and migration patterns of Liriomyza sativae in China:Moderate subdivision and no Bridgehead effect revealed by microsatellites[J]. Bulletin of Entomological Research, 2016, 106(1): 114-123. DOI:10.1017/S0007485315000905 |
| [25] |
Bilton D T, Paula J, Bishop J D D. Dispersal, genetic differentiation and speciation in estuarine organisms[J]. Estuarine, Coastal and Shelf Science, 2002, 55(6): 937-952. DOI:10.1006/ecss.2002.1037 |
| [26] |
Wilke T, Davis G M. Infraspecific mitochondrial sequence diversity in Hydrobia ulvae and Hydrobia ventrosa (Hydrobiidae:Rissooidea:Gastropoda):Do their different life histories affect biogeographic patterns and gene flow?[J]. Biological Journal of the Linnean Society, 2000, 70(1): 89-105. DOI:10.1111/j.1095-8312.2000.tb00202.x |
| [27] |
Pierce J C, Tan G N, Gaffney P M. Delaware Bay and Chesapeake Bay populations of the horseshoe crab Limulus polyphemus are genetically distinct[J]. Estuaries, 2000, 23(5): 690-698. DOI:10.2307/1352895 |
 2020, Vol. 27
2020, Vol. 27

