核酸适配体通常是指在体外利用指数富集配体的系统进化技术(Systematic Evolution of Ligands by Exponential Enrichment, SELEX), 即SELEX技术, 筛选得到的寡核苷酸分子包括ssDNA和RNA[1]。它具有亲和特异性高、靶标范围广、稳定性好等特点[2], 已在生命科学研究、药物筛选、靶点鉴定、环境监测等多个领域受到广泛关注[3]。亲和特异性是选择和判断核酸适配体的一个重要指标。一个好的核酸适配体必须对靶目标有较高的亲和特异性[4]。因此, 测定核酸适配体的亲和力, 研究其对靶目标的亲和特异性, 既是对靶目标识别鉴定的前提, 也是核酸适配体研究开发的基础。目前有关核酸适配体亲和力的测定方法众多, 比较常用的方法是对核酸适配体进行标记后, 再对相应标记进行检测[5-9], 如较为经典的流式细胞术, 一般都需要对核酸适配体进行荧光标记[5-7], 这种方法由于引入了标记基团, 有可能会导致核酸适配体空间结构的变化, 影响其亲和力, 而且标记的引入也大大增加了检测成本, 而如果采用流式细胞仪等大型仪器进行检测, 则检测成本还要大幅增加[10]; 此外, 一些学者还报道了一些较新颖的亲和力检测方法, 如表面等离子体共振法(SPR)[11]、高效液相色谱法(HPLC)[12]、荧光定量PCR法[13]、纳米金颗粒法[14-15], 但这些方法尚不普及, 其应用效果还有待进一步确认。
哈维氏弧菌(Vibrio harveyi)属于弧菌科, 弧菌属, 菌体呈短杆状, 长1~2 μm, 极生单鞭毛, 最适温度为20~35 ℃[16]。它可以感染鲈、金头鲷、石斑鱼、大黄鱼等多种海水养殖鱼类, 也是养殖对虾的主要病原体, 对海水养殖业有巨大危害[17-18]。因此, 对哈维氏弧菌进行准确有效的检测和鉴定是十分必要。而哈维氏弧菌的核酸适配体在哈维氏弧菌的识别鉴定中展现出较好的应用前景。
通过前期研究笔者实验室获得了哈维氏弧菌的一系列核酸适配体, 并采用地高辛−过氧化物酶方法对这些核酸适配体的亲和特异性进行了初步研究[19]。但后期的研究发现, 地高辛−过氧化物酶的检测方法过于繁琐。本研究利用核酸适配体的单链DNA特性, 将核酸适配体与靶细菌——哈维氏弧菌孵育结合, 通过测定哈维氏弧菌上结合的单链DNA浓度, 来表征该核酸适配体对哈维氏弧菌的亲和力, 进而研究其对哈维氏弧菌的亲和特异性, 最后通过荧光显微镜来验证其亲和特异性, 旨为水产病原微生物核酸适配体的开发应用提供参考。
1 材料与方法 1.1 材料 1.1.1 核酸适配体核酸适配体为笔者实验室前期筛选得到的哈维氏弧菌核酸适配体, 其序列为: 5ʹ−TCAGTCGCTTCGCCGTCTTCAGCCGGGGTG GTCAGTAGGAGCAGCACAAGAGGGAGACCCCAGAGGG−3ʹ。用于荧光显微镜鉴定直接观察法实验中的核酸适配体, 其5ʹ端标记有FAM荧光素。核酸适配体的合成和标记都由上海生工生物工程有限公司完成。
1.1.2 实验用菌哈维氏弧菌、溶藻弧菌(Vibrio alginolyticus)、嗜水气单胞菌(Aeromonas hydrophila)、迟钝爱德华氏菌(Edwardsiella tarda)和大肠杆菌(Escherichia coli)由集美大学水产病害实验室鉴定并提供。
1.1.3 培养基和缓冲液胰蛋白胨大豆肉汤培养基(TSB培养基), 用于哈维氏弧菌、溶藻弧菌、嗜水气单胞菌和迟钝爱德华氏菌的培养; LB培养基(Luria-Bertani培养基), 用于大肠杆菌的培养。
20×缓冲液:氯化钠(NaCl) 5.844 g, 氯化钾(KCl) 3.725 g, 三羟甲基氨基甲烷(Tris-HCl) 6.06 g, 六水合氯化镁(MgCl2·6H2O) 2.033 g。上述物质全部定容至100 mL超纯水中, 调pH至7.4。使用时用超纯水稀释为2×和1×缓冲液。
1.2 实验方法 1.2.1 核酸适配体的亲和力测定依次通过菌处理、核酸适配体处理、菌与核酸适配体结合、洗涤、加热分离和检测等步骤测定核酸适配体对相应细菌的亲和力, 具体如下。
菌处理:取一定量的菌液, 6000 r/min, 离心5 min后弃上清, 用1×缓冲溶液洗涤3次后再用2×缓冲溶液重悬。测其OD值, 根据其OD值和菌的标准曲线, 配制成浓度为108 cfu/mL的菌液。
核酸适配体处理:用2×缓冲溶液将10 μmol/L无荧光标记的核酸适配稀释至0.3 μmol/L后, 95 ℃变性5 min, 再4 ℃冰浴10 min。
菌与核酸适配体结合:取100 μL菌液(108 cfu/mL)和上述处理过的100 μL (0.3 μmol/L)核酸适配体混合作为实验组; 另取100 μL菌液(108 cfu/mL)和100 μL 2×缓冲溶液混合作为对照组; 每组3个平行。实验组和对照组都在30 ℃, 100 r/min摇床结合30 min。
洗涤:结合完毕后, 6000 r/min离心5 min, 去上清, 用200 μL 1×缓冲溶液洗涤3次并离心去上清, 以去除未结合菌的核酸适配体, 最后用100 μL 2×缓冲溶液重悬。
加热:菌液洗涤并重悬后, 在95 ℃加热5 min, 15000 r/min离心10 min, 取上清用于后续测定单链DNA浓度。
检测:用K5500超微量紫外可见分光光度计分别测定实验组和对照组的单链DNA浓度, 实验组的单链DNA浓度=菌的背景值+结合到菌上的核酸适配体的浓度, 对照组的单链DNA浓度则为菌的背景值; 用结合到菌上的核酸适配体的浓度表示的亲和力=实验组的单链DNA浓度−对照组的单链DNA浓度。
1.2.2 核酸适配体的亲和常数测定核酸适配体处理:用2×缓冲溶液将10 μmol/L无荧光标记的核酸适配依次稀释至10 nmol/L、20 nmol/L、40 nmol/L、60 nmol/L、80 nmol/L和100 nmol/L。
结合:取100 μL浓度为108 cfu/mL的菌液, 分别与100 μL浓度为10 nmol/L、20 nmol/L、40 nmol/L、60 nmol/L、80 nmol/L和100 nmol/L的上述核酸适配体结合, 作为实验组; 另取100 μL浓度为108 cfu/mL的菌液和100 μL 2×缓冲溶液结合作为对照组; 每组3个平行。实验组和对照组都在30 ℃, 100 r/min摇床结合30 min, 然后按照1.2.1的亲和力测定方法对实验组和对照组进行洗涤和亲和力的检测, 最后以核酸适配体浓度为横坐标, 以亲和力为纵坐标, 采用双曲线函数(hyperbola函数), 用Oringin9.0软件进行非线性拟合, 可得到相应核酸适配体的拟合曲线及其亲和常数和拟合常数。
1.2.3 利用荧光显微镜鉴定核酸适配体的亲和特异性直接观察法:用带有荧光标记的核酸适配体分别与各种菌孵育后, 在荧光显微镜下观察适配体和菌的结合情况。具体如下。
结合和洗涤:取100 μL浓度为108 cfu/mL的菌液与100 μL浓度为0.03 μmol/L的带有荧光标记的核酸适配体孵育, 作为实验组; 100 μL浓度为108 cfu/mL的菌液与100 μL 2×缓冲溶液孵育, 作为对照组; 实验组和对照组都在30 ℃、100 r/mim下摇床孵育30 min, 然后按1.2.1的方法进行洗涤、重悬。
涂片观察:取上面洗涤重悬后的菌悬液1 μL涂于玻片上, 酒精灯加热固定; 番红染色1 min后用适量超纯水洗净; 待其干后置于荧光显微镜下观察, 并在亮度为71%, 伽马值为0.65, 荧光强度2档的条件下分别拍摄明场(bright field)和荧光场(fluorescence field)下的图片。
荧光阻断法:先用没有荧光标记的核酸适配体分别与各种菌孵育, 再加入有荧光标记的核酸适配体孵育, 在显微镜下观察菌的荧光是否被阻断。具体如下:
结合和洗涤:取100 μL浓度108 cfu/mL的菌液先与100 μL浓度为0.03 μmol/L的无荧光标记的核酸适配体在30 ℃、100 r/mim下摇床孵育30 min, 然后按1.2.1的方法进行洗涤、重悬。重悬后再加入100 μL浓度为0.03 μmol/L的有荧光标记的核酸适配体, 在30 ℃、100 r/mim下孵育30 min, 然后用按1.2.1的方法进行洗涤、重悬。
涂片观察:同直接观察法。
1.3 统计分析所有实验数据都重复至少6次以上。利用EXCEL软件中的t检验对实验数据进行组间差异分析, P < 0.05为显著差异, P < 0.01为极显著差异。
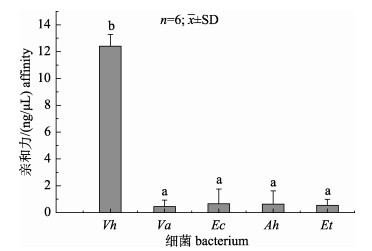
2 结果与分析 2.1 核酸适配体的亲和特异性研究如图 1所示, 核酸适配体对目标菌哈维氏弧菌的亲和力要显著高于溶藻弧菌、大肠杆菌、嗜水气单胞菌和迟钝爱德华氏菌等非目标菌(P < 0.01), 对哈维氏弧菌的亲和力约是非目标菌的15.2倍以上, 显示出该核酸适配体对哈维氏弧菌有较好的亲和特异性和识别鉴定效果。
|
图 1 核酸适配体对不同菌的亲和力比较 Ah:嗜水气单胞菌; Ec:大肠杆菌; Et:迟钝爱德华氏菌; Va:溶藻弧菌; Vh:哈维氏弧菌.不同字母表示差异显著(P < 0.05). Fig.1 Comparison of aptamer affinity with different bacteria Ah: Aeromonas hydrophila; Ec: Escherichia coli; Et: Edwardsiella tarda; Va: Vibrio alginolyticus; Vh: Vibrio harveyi. Different letters indicate significant difference (P < 0.05). |
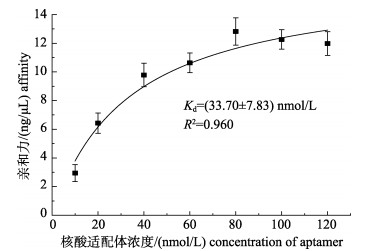
核酸适配体对目标菌哈维氏弧菌的亲和力随着核酸适配体浓度的增加而增加, 并在后期逐渐趋于稳定, 呈现出亲和常数常见的双曲线函数特征(图 2)。用双曲线函数对曲线进行拟合后可得其亲和常数Kd=(33.70±7.83) nmol/L, 相应的拟合系数R2=0.960, 呈现出较好的拟合效果。
|
图 2 核酸适配体的亲和常数饱和曲线 Fig.2 Saturation curve of affinity constants of the aptamer |
分别采用直接观察法和荧光阻断法鉴定核酸适配体的亲和特异性。直接观察法中(如图 3A), 在放大200倍的荧光显微镜下, 未结合过核酸适配体的5种菌(哈维氏弧菌、溶藻弧菌、大肠杆菌、嗜水气单胞菌、迟钝爱德华氏菌), 无论在明场还是在荧光场条件下都未呈现出明显的荧光(图 3A中的Vh、Va、Ec、Ah、Et), 说明这些菌在实验条件下自身是不带荧光的; 但与核酸适配体结合后, 靶细菌哈维氏弧菌在荧光场下显现出明亮的黄红色荧光, 而非目标菌溶藻弧菌、大肠杆菌、嗜水气单胞菌和迟钝爱德华氏菌在荧光场下依然没有明显荧光(图 3A), 说明带有荧光基团的核酸适配体能与靶细菌哈维氏弧菌较好地结合, 即有较好的亲和特异性, 但对其他4种非目标菌的亲和力较弱。荧光阻断法的结果则显示, 在放大200倍的荧光显微镜下, 包括靶细菌哈维氏弧菌在内的5种菌都未观察到明显的荧光(图 3B), 说明靶细菌哈维氏弧菌出现了荧光阻断现象, 这也进一步表明该核酸适配体对哈维氏弧菌有较好的亲和特异性。

|
图 3 荧光显微镜下核酸适配体对不同菌的识别效果
A.直接观察法; B.荧光阻断法. BF:明场; FLUO:荧光场; Ah:嗜水气单胞菌; Ah+Apt:与核酸适配体混合后的 嗜水气单胞菌; Ec:大肠杆菌; Ec+Apt:与核酸适配体 混合后的大肠杆菌; Et:迟钝爱德华氏菌; Et+Apt:与 核酸适配体混合后的迟钝爱德华氏菌; Va:溶藻弧菌; Va+Apt:与核酸适配体混合后的溶藻弧菌; Vh:哈维氏 弧菌; Vh+Apt:与核酸适配体混合后的哈维氏弧菌. Fig.3 Identification of the aptamer towards different bacteria under fluorescence microscope A. Experiment by direct observation; B. Experiment by fluorescence blocking method. BF: bright field; FLUO: fluorescence field; Ah: Aeromonas hydrophila; Ec: Escherichia coli; Et: Edwardsiella tarda; Va: Vibrio alginolyticus; Vh: V. haveyi; Ah+Apt: A. hydrophila mixed with aptamer; Ec+Apt: E. coli mixed with aptamer; Et+Apt: E.tarda mixed with aptamer; Va+Apt: V. alginolyticus mixed with aptamer; Vh+Apt: V. harveyi mixed with aptamer. |
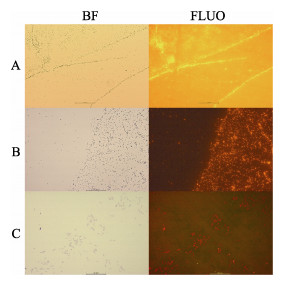
利用荧光显微镜在不同放大倍数下观察结合了核酸适配体的哈维氏弧菌(图 4), 从中可以看出, 在分别放大了200倍、400倍和1000倍的荧光显微镜下, 结合了核酸适配体后的哈维氏弧菌显现出明亮的黄红色荧光, 明显区别于背景, 说明该核酸适配体对哈维氏弧菌有较好的亲和力和识别效果。
|
图 4 不同放大倍数下核酸适配体对哈维氏弧菌的识别效果 BF:明场; FLUO:荧光场; A:放大200倍; B:放大400倍; C:放大1000倍. Fig.4 Recognition of Vibrio harveyi mixed with aptamer under fluorescence microscope of different magnifications BF: bright field; FLUO: fluorescence field; A: magnification of 200 times; B: magnification of 400 times; C: magnification of 1000 times. |
本研究利用核酸适配体对靶目标的高亲和特异性及其单链DNA特性, 通过测定靶目标上的单链DNA浓度来表征其亲和力, 进而研究该核酸适配体的亲和特异性和亲和常数, 结果显示该核酸适配体对哈维氏弧菌有较好的亲和特异性, 这与之前采用地高辛−过氧化物酶法所获得的结果是一致的[20]。而且, 之前的方法测得的该核酸适配体的亲和常数Kd=(34.75±5.61) nmol/L[20], 而此次采用单链DNA法测得该核酸适配体的亲和常数Kd=(33.70±7.83) nmol/L, 二者极为接近, 说明采用单链DNA浓度法来测定核酸适配体的亲和力和亲和常数是可行的。采用地高辛−过氧化物酶法测定核酸适配体亲和力, 需要使用地高辛标记核酸适配体与靶细菌孵育, 然后再加入辣根过氧化物酶标记的兔抗地高辛抗体, 形成菌−核酸适配体−地高辛−辣根过氧化物酶标记兔抗地高辛抗体复合物, 最后还要经过显色剂显色和酶标仪测定, 整个过程较为繁琐、复杂[19]。而本研究采用的单链DNA法, 不仅不需要标记核酸适配体, 而且核酸适配体与靶目标菌孵育洗涤后可以直接用超微量紫外分光光度计来测定其亲和力, 整个过程更为简便、快捷。
目前有关核酸适配体的亲和力测定方法并不统一, 不同学者常常根据自己研究的需要建立不同的亲和力测定方法[21]。其中, 相当多的方法都是要对核酸适配体进行标记, 然后再对相应标记进行检测的, 有的还需通过PCR进一步放大检测信号[5-9]。本研究通过测定靶目标上核酸适配体的单链DNA浓度来表征其亲和力, 无需对核酸适配体进行标记, 无需PCR扩增, 可直接用超微量紫外分光光度计进行测定, 不仅比之前的地高辛−过氧化物酶法的检测效率有了大幅提高, 比一些PCR法在检测效率上也有较好的提升[10]。该方法仪器设备要求简单、检测迅速、结果稳定、检测成本低, 在当前众多核酸适配体的亲和特异性检测方法之中, 不失为一种快捷、经济、可靠的检测手段。
有关水产病原弧菌的荧光显微镜鉴定已有学者进行了研究, 并取得了较好效果[22-23]。其主要原理就是通过一抗来特异性识别目标菌, 再通过带有荧光标记的二抗对目标菌进行荧光标记, 最后通过荧光显微镜实现对目标菌的鉴定。由于核酸适配体对靶细菌有较好的亲和特异性, 且容易进行荧光修饰, 因此, 本研究在直接观察法中采用带有荧光标记的核酸适配体来代替其中的一抗和二抗, 再通过荧光显微镜来进行鉴定, 结果显示, 用核酸适配体替代抗体进行荧光显微镜鉴定有较好的识别鉴定效果。本研究还采用荧光阻断法来鉴定该核酸适配体的亲和特异性, 即先用没有荧光标记的核酸适配体与菌孵育, 之后洗去未结合的核酸适配体, 再加入带有荧光标记的核酸适配体与菌孵育, 最后荧光显微镜观察, 结果发现目标菌哈维氏弧菌出现了荧光阻断现象, 这表明目标菌哈维氏弧菌在与荧光标记的核酸适配体结合之前, 菌上相应的结合位点已被无荧光标记的该核酸适配体所占据, 阻止了后续荧光适配体的结合, 从而出现荧光阻断现象。而对于其他4种非目标菌, 由于与该核酸适配体的亲和力较弱, 因此在这两种方法中, 它们的荧光都很弱。荧光显微镜的鉴定结果进一步证明了该核酸适配体对哈维氏弧菌有较好的亲和特异性, 也进一步表明用单链DNA浓度法测定核酸适配体的亲和力和亲和常数是准确可靠的。
| [1] |
Röthlisberger P, Hollenstein M. Aptamer chemistry[J]. Advanced Drug Delivery Reviews, 2018, 134: 3-21. DOI:10.1016/j.addr.2018.04.007 |
| [2] |
Kaur H. Recent developments in cell-SELEX technology for aptamer selection[J]. Biochimica et Biophysica Acta (BBA)-General Subjects, 2018, 1862(10): 2323-2329. DOI:10.1016/j.bbagen.2018.07.029 |
| [3] |
Acquah C, Danquah M K, Agyei D, et al. Deploying aptameric sensing technology for rapid pandemic monitoring[J]. Critical Reviews in Biotechnology, 2016, 36(6): 1010-1022. DOI:10.3109/07388551.2015.1083940 |
| [4] |
Yang Y, Yang D L, Schluesener H J, et al. Advances in SELEX and application of aptamers in the central nervous system[J]. Biomolecular Engineering, 2007, 24(6): 583-592. DOI:10.1016/j.bioeng.2007.06.003 |
| [5] |
Yu Q, Liu M Z, Su H F, et al. Selection and characterization of ssDNA aptamers specifically recognizing pathogenic Vibrio alginolyticus[J]. Journal of Fish Diseases, 2019, 42(6): 851-858. DOI:10.1111/jfd.12985 |
| [6] |
Suh S H, Dwivedi H P, Choi S J, et al. Selection and characterization of DNA aptamers specific for Listeria species[J]. Analytical Biochemistry, 2014, 459: 39-45. DOI:10.1016/j.ab.2014.05.006 |
| [7] |
Ababneh N, Alshaer W, Allozi O, et al. In vitro selection of modified RNA aptamers against CD44 cancer stem cell marker[J]. Nucleic Acid Therapeutics, 2013, 23(6): 401-407. DOI:10.1089/nat.2013.0423 |
| [8] |
Tang X M, Zheng J, Yan Q P, et al. Selection of aptamers against inactive Vibrio alginolyticus and application in a qualitative detection assay[J]. Biotechnology Letters, 2013, 35(6): 909-914. DOI:10.1007/s10529-013-1154-1 |
| [9] |
Bayrağ C, Eyidoğan F, Avni Öktem H. DNA aptamer-based colorimetric detection platform for Salmonella enteritidis[J]. Biosensors and Bioelectronics, 2017, 98: 22-28. DOI:10.1016/j.bios.2017.06.029 |
| [10] |
Tang X M, Lin W Y, Yan Q P, et al. Detection of Vibrio alginolyticus and its inactivated form based on aptamers and polymerase chain reaction[J]. Journal of Fishery Sciences of China, 2019, 26(2): 391-396. [汤学敏, 林文燕, 鄢庆枇, 等. 基于核酸适配体的PCR法检测溶藻弧菌及其灭活菌[J]. 中国水产科学, 2019, 26(2): 391-396.] |
| [11] |
Zhang C G, Chang S J, Settu K, et al. High-sensitivity glycated hemoglobin (HbA1c) aptasensor in rapid-prototyping surface plasmon resonance[J]. Sensors and Actuators B:Chemical, 2019, 279: 267-273. DOI:10.1016/j.snb.2018.09.077 |
| [12] |
Khodadadi M, Malekpour A, Mehrgardi M A. Aptamer functionalized magnetic nanoparticles for effective extraction of ultratrace amounts of aflatoxin M1 prior its determination by HPLC[J]. Journal of Chromatography A, 2018, 1564: 85-93. DOI:10.1016/j.chroma.2018.06.022 |
| [13] |
Guo X D, Wen F, Zheng N, et al. A qPCR aptasensor for sensitive detection of aflatoxin M1[J]. Analytical and Bioanalytical Chemistry, 2016, 408(20): 5577-5584. DOI:10.1007/s00216-016-9656-z |
| [14] |
Wang C S, Liu J, Han X Y, et al. UV-visible spectroscopic detection of kanamycin based on target-induced growth of gold nanoparticles[J]. Analytical Methods, 2017, 9(33): 4843-4850. DOI:10.1039/C7AY01685A |
| [15] |
Yuan H Y, Huang Y M, Yang J D, et al. An aptamer-based fluorescence bio-sensor for chiral recognition of arginine enantiomers[J]. Spectrochimica Acta Part A:Molecular and Biomolecular Spectroscopy, 2018, 200: 330-338. DOI:10.1016/j.saa.2018.04.038 |
| [16] |
Alsina M, Blanch A R. Improvement and update of a set of keys for biochemical identification of Vibrio species[J]. Journal of Applied Bacteriology, 1994, 77(6): 719-721. DOI:10.1111/j.1365-2672.1994.tb02824.x |
| [17] |
Vandenberghe J, Li Y, Verdonck L, et al. Vibrios associated with Penaeus chinensis (Crustacea:Decapoda) larvae in Chinese shrimp hatcheries[J]. Aquaculture, 1998, 169(1-2): 121-132. DOI:10.1016/S0044-8486(98)00319-6 |
| [18] |
Alvarez J D, Austin B, Alvarez A M, et al. Vihrio harveyi:A pathogen of penaeid shrimps and fish in Venezuela[J]. Journal of Fish Diseases, 1998, 21(4): 313-316. DOI:10.1046/j.1365-2761.1998.00101.x |
| [19] |
Hao J M, Zheng J, Tang X M, et al. A simplified system without purification for selection of aptamers against Vibrio alginolyticus[J]. African Journal of Microbiology Research, 2011, 5(21): 3564-3568. |
| [20] |
Zheng J, Hao J M, Song L S, et al. Selection and characterization of aptamers against Vibrio harveyi by SELEX[J]. Progress in Biochemistry and Biophysics, 2014, 41(7): 704-711. [郑江, 郝聚敏, 宋林生, 等. 哈维氏弧菌适配子的SELEX筛选及其亲和特异性研究[J]. 生物化学与生物物理进展, 2014, 41(7): 704-711.] |
| [21] |
Liu D L, Zhang Z L, Yin Y J, et al. Development and evaluation of a novel in situ target-capture approach for aptamer selection of human noroviruses[J]. Talanta, 2019, 193: 199-205. DOI:10.1016/j.talanta.2018.09.084 |
| [22] |
Yan Q P, Zou W Z, Ji R X, et al. Detection of Vibrio fluviails in Paralichthys olivaceus using indirect fluorescent antibody technique[J]. Marine Sciences, 2006, 30(4): 16-19. [鄢庆枇, 邹文政, 纪荣兴, 等. 应用荧光抗体技术检测牙鲆体内的河流弧菌[J]. 海洋科学, 2006, 30(4): 16-19. DOI:10.3969/j.issn.1000-3096.2006.04.004] |
| [23] |
Zhang X H, Zhong Y B, Chen J X. The biological characteristics, epidemiology and detection techniques of Vibrio harveyi[J]. Periodical of Ocean University of China, 2007, 37(5): 740-748. [张晓华, 钟英斌, 陈吉祥. 哈维氏弧菌的生物学特性、流行病学及检测技术[J]. 中国海洋大学学报(自然科学版), 2007, 37(5): 740-748.] |
 2020, Vol. 27
2020, Vol. 27

