2. 中国水产科学研究院淡水渔业研究中心, 江苏 无锡 214081
2. Freshwater Fisheries Research Center, Chinese Academy of Fishery Sciences, Wuxi 214081, China
传统池塘养殖强调“养鱼先养水, 养水先养泥”。池塘养殖过程中伴随着饵料连续投入, 产生大量的残饵和排泄代谢产物, 导致营养盐和有机质在养殖水体和底泥中不断富集, 形成富营养化的养殖环境[1]。在池塘养殖这种人为生态系统中, 底泥是最活跃、最具影响力的成分之一, 它与养殖对象及养殖水体间存在密切的关联[2]。养殖池塘底泥沉积有大量的水体营养物质, 能够缓冲养殖环境的剧烈变化, 并为养殖生态系统中的微生物、浮游动物和底栖动物等提供了生活栖息场所[3]。
微生物群落是养殖池塘生态系统的重要组成部分, 并在养殖池塘生态系统中发挥重要作用, 如, 营养盐循环、有机质降解和病害控制等[4]。微生物群落的丰度、多样性以及组成与养殖环境息息相关[4-5], 尤其是养殖池塘底泥富含营养盐和有机质, 细菌的丰度和多样性更高[6]。而水产养殖池塘微生物群落具有系统特异性, 近年来, 针对不同养殖种类池塘水体和底泥的微生物监测和调控相继开展[7-9]。但目前对不同养殖系统中微生物群落生态学知识依然缺乏, 水产养殖微生物群落调控与管理更是难以实现。因此, 对微生物群落与复杂的水产养殖生态系统之间的相互作用开展研究十分必要。本文对斑点叉尾鮰(Ictalurus punctatus)养殖池塘底泥细菌群落的周年变化开展了研究, 目的是了解斑点叉尾鮰养殖池塘底泥细菌群落组成与多样性的年度变化特征, 同时明确斑点叉尾鮰养殖池塘底泥细菌群落结构的主要影响因子, 这些研究结果将对斑点叉尾鮰养殖的精细化管理和健康养殖提供帮助。
1 材料与方法 1.1 试验系统及样品采集试验在江苏省淡水水产研究所国家斑点叉尾鮰遗传育种中心养殖基地开展。养殖池塘0.21 hm2 (50 m×42 m), 位于南京市江宁区禄口镇(31°77′03″N, 118°88′29″E)。养殖开始前1个月抽干池水, 漂白粉清塘杀除野杂鱼, 加水至1.5 m。池塘中央设置一台增氧机, 以保证池塘溶氧。池塘内放入2747尾斑点叉尾鮰鱼种(平均体重18.91 g), 养殖从11月底至次年12月初。每日早晚两次投喂漂浮性颗粒配合饲料(蛋白含量35%), 饲料投喂量为鱼体重3%左右。
养殖周期内, 每月的月末开展池塘底泥样品的采集。每次上午10: 00, 利用彼得森采泥器(5 L)采集池塘中央、池边及饲料投喂台3处的表层(15 cm)底泥样品, 放入灭菌塑料封袋。池塘中央和池边两处底泥呈黑色, 饲料投喂台处底泥表层含有一层土黄色有机质, 底部为黑色。水样4 ℃保存带回实验室, 分析时将3个点底泥样均匀混合后取样(D1~D12)。
1.2 环境因子测定利用多功能水质参数仪(YSI6920v2, USA)对底泥上覆水物理性质进行原位测定, 主要包括:温度(temperature, Temp)、pH、总溶解性悬浮物(total dissolved solids, TDS)以及溶解氧(dissolved oxygen, DO)。塞氏盘现场测定水体透明度(water transperancy, SD)。底泥先冷冻干燥研磨后, 采用重铬酸钾-硫酸(油浴)氧化-硫酸亚铁滴定法测定有机质(organic matter, OM)含量和总有机碳(total organic carbon, TOC)。底泥中总氮(total nitrogen, TN)先将风干研磨的泥样加入50 mL去离子水中混合, 加碱性过硫酸钾氧化消解后, 紫外分光光度法测定。总磷(total phosphorus, TP)也是将风干研磨的泥样加入50 mL去离子水中混合, 加碱性过硫酸钾氧化消解后, 采用钼锑抗分光光度法测定。
1.3 DNA提取和16S rRNA基因扩增利用QiagenDNeasy Blood & Tissue Kit试剂盒(Qiagen, CA, 美国)提取底泥微生物总DNA, 检测合格的DNA保存于‒20 ℃备用。使用引物为338F和806R[10], 扩增16S rRNA基因V3~V4高变区, 建库高通量测序在上海美吉生物医药科技有限公司完成。
1.4 数据优化与分析Trimmomatic软件[11]去除测序原始序列的引物和barcode标签后, 通过FLASH软件[12]进行序列拼接, 使用VSEARCH (1.9.6)将序列以97%相似性聚类成操作分类单元(operational taxonomic units, OTUs), 比对的参考数据库是Silva_132 16SrRNA database (http://www.arb-sliva.de)[13]。
所有统计分析及可视化分析均利用R语言中的vegan和ggplot2程序包(version 3.2.2, http://www.r-project.org)以及上海美吉生物医药科技有限公司Ⅰ-sanger云平台完成。进行多样性分析前, 根据每个样本中最少的序列数进行抽平, 以保证每个样本中的高通量数据在相同的水平上。利用mothur软件计算α多样性[14]。计算β多样性前, 对抽平后的物种数据进行Hellinger转化[15]。通过非度量多维尺度分析(nonmetric multi-dimensional scaling, NMDS), 反映不同样本间在多维空间上的差异程度[16]。利用冗余分析(redundancy analysis, RDA)对环境因子与微生物群落进行典范对应分析, 其中首先对环境因子进行999次Monte Carlo置换检验的前向选择[17]。利用相似性分析(analysis of similarities, ANOSIM)检验季节间是否存在显著性差异。最后, 通过多物种线性差异判别分析(linear discriminant analysis effect size, LEfSe), 根据分类学组成对样本按照不同分组条件进行线性判别分析(LDA), 找出对季节显著性差异影响的群落[18]。
2 结果与分析 2.1 底泥理化特征养殖试验过程中底泥与上覆水的年度理化特征见表 1。底泥上覆水温(Temp)呈现出明显的季节性变化, 从冬季1月末逐步升高, 夏季7月末是达到峰值33.18 ℃, 之后缓慢下降, 除春季和秋季外, 其他季节间上覆水温都存在显著性差异(P < 0.05)。总溶解性悬浮物(TDS)和水体透明度(SD)都呈现出持续下降的趋势, 冬春季节都显著高于夏秋季(P < 0.05)。上覆水pH介于7.49~9.50, 始终保持偏碱性, 冬季上覆水的碱性显著高于夏秋季(P < 0.05)。上覆水溶氧(DO)含量为3.32~ 16.30 mg/L, 冬季水体的溶氧含量显著高于其他3个季节(P < 0.05)。水体总氮(TN)和总磷(TP)浓度始终维持较高水平, 冬季的TN和TP浓度显著高于其他季节(P < 0.05), 其他3个季节间则变化不大。底泥的有机质(OM)含量和总有机碳(TOC)养殖周期内相对保持稳定, 季节间没有表现出显著差异。
|
|
表 1 斑点叉尾鮰养殖池塘底泥和上覆水的理化性质 Tab.1 Summary of physicochemical parameters for sediment and overlying water samples of Ictalurus punctatu pond |
经优化处理后, 共获得357602条序列, 平均每个样品拥有29800条高质量序列, 序列平均长度为441.28 bp, 这些序列被分配至3043个OTUs。稀释曲线中所有样本的都接近平缓, 表明试验抽样充分, 测序深度足以反映每个底泥样本细菌群落的生物信息。
为了了解养殖池塘底泥细菌群落的物种丰富度和多样性, 选取Ace指数、Chao1指数、Shannon指数和Simpson指数。养殖年度内, Ace指数和Chao1指数变化趋势较为一致, 呈W形变化, 最小值出现在秋季9月, 最大值为冬季的1月和春季的5月(图 1)。养殖池塘底泥细菌群落Shannon指数和Simpson指数变化趋势相反, 表明细菌群落多样性变化相对稳定, 预测结果较为一致。两个指数冬季和夏季相对稳定, 但是春秋季出现剧烈波动, 4月出现了年度极值, Shannon指数在秋季的3个月连续剧烈下降, 而对应的Simpson指数在秋季3个月中只是小幅波动。利用单因素方差分析(ANOVA)对4个季度的α多样性指数进行多重比较, 结果表明, 4个α多样性指数间均不存在显著性差异(P > 0.05)。
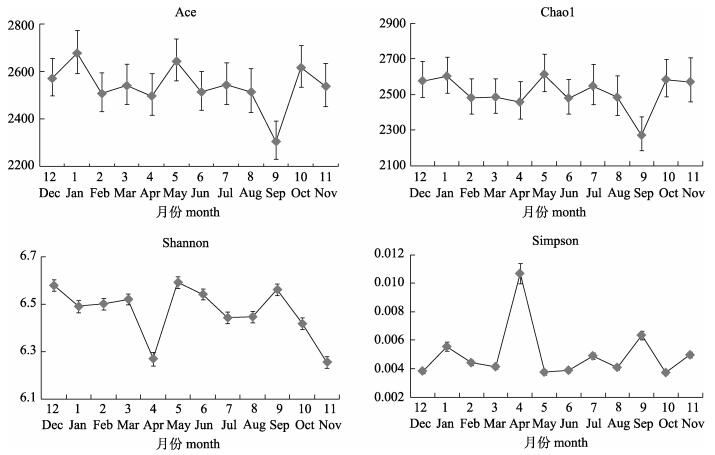
|
图 1 养殖池塘底泥微生物群落α多样性(n=3;x±SD) Fig.1 The alpha diversity of bacterial community in sediment samples (n=3;x±SD) |
利用非度量多维尺度分析(NMDS)反映养殖池塘底泥细菌群落的β多样性。从NMDS分布图(图 2A)可以看出, 夏季和秋季样本呈现出较好的季节性排列趋势, 季度内的样品间距离更近, 微生物群落也更加相似; 而冬季和春季的样本重叠在一起, 整体排在左上侧。NMDS分析的stress值为0.115, 表明排序结果相对可靠。细菌群落层次聚类分析结果与NMDS类似, 夏季(8月样品除外)和秋季样品聚为一簇, 而冬季和春季样品相互交叉总体聚为另一簇(图 2B)。
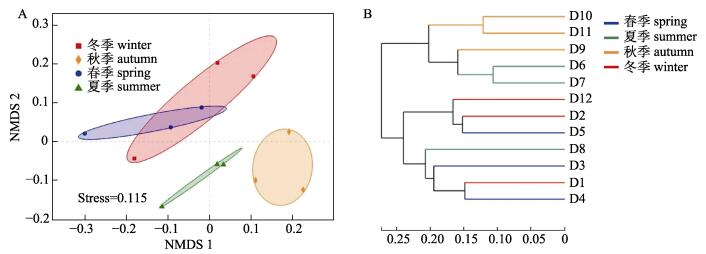
|
图 2 养殖池塘底泥微生物群落NMDS排序图(A)和层级聚类分析(B) Fig.2 NMDS plot (A) and hierarchical clustering analysis (B) of the compositional dissimilarities of the bacterial communities |
3043个OTU被注释为57个门(phyla), 128个纲(classes)和585个属(genera)。为了减少群落的复杂性和冗余性, 相对丰度低于1%的群落都归为稀有种, 并为“others”[19]。
韦恩图表明, 养殖池塘水体样本中OTU数量从高到低呈现为冬季(2751) > 春季(2665) > 秋季(2615) > 夏季(2599)。4个季节样本共享的OTU有2058个, 占全部OTU的67.63%, 3个季节和2个季节样品间共享OTU分别为548和317个, 占比18.01%和10.41%。而单季样品独有的OTU数量最少, 只有120个, 占比3.94%。其中, 冬季和秋季样品独有OTU最多, 分别为38和36个, 占比1.24%和1.18%, 夏季样品独有OTU最少, 仅16个, 占比0.52% (图 3)。
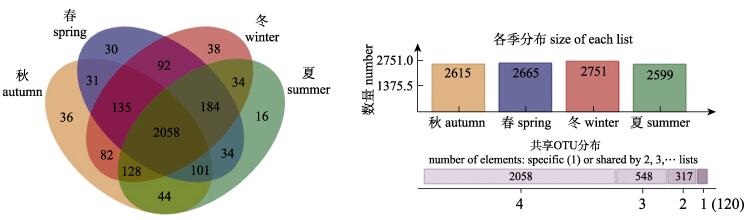
|
图 3 养殖池塘底泥4个季度间微生物群落组成韦恩图 Fig.3 Venn diagram representing shared OTUs among four seasons |
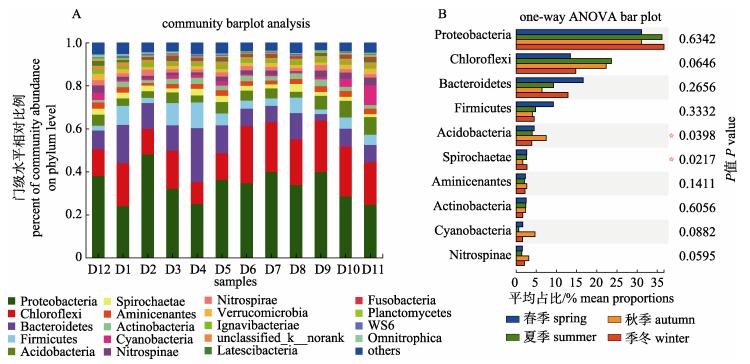
ANOSIM检验表明4个季度养殖池塘底泥样品间细菌群落组成存在显著性差异(P < 0.01)。4个季度养殖池塘水体样品细菌群落结构组成, 在门级水平上组成较为相似(图 4)。相对丰度从高到低依次为, 变形菌门Proteobacteria (34%), 绿弯菌门Chloroflexi (19%), 拟杆菌门Bacteroidetes (11%), 厚壁菌门Firmicutes (5.7%), 酸杆菌门Acidobacteria (4.9%), 这5个门占总群落的74.6% (图 4)。其后, 螺旋体门Spirochaetae (2.4%), Aminicenantes (2.3%), 放线菌门Actinobacteria (2.2%), 蓝藻门Cyanobacteria (2.2%), Nitrospinae (2.0%), 硝化螺旋菌门Nitrospirae (1.9%), 疣微菌门Verrucomicrobia (1.8%)和Ignavibacteriae (1.7%)相对丰度都低于3%。
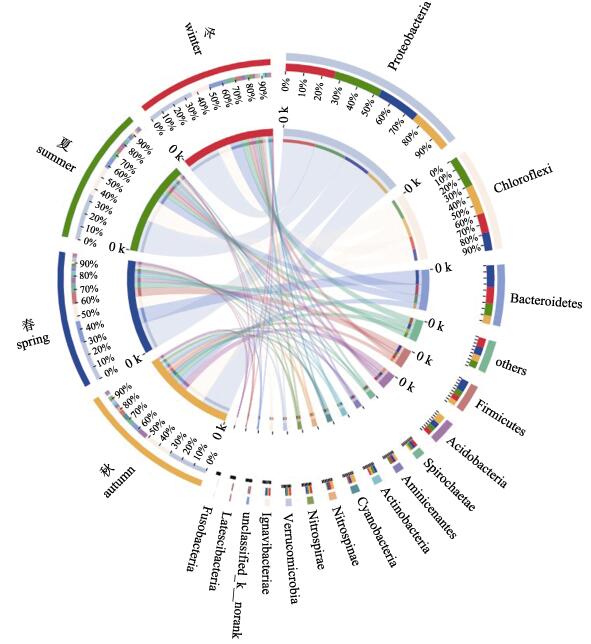
|
图 4 养殖池塘底泥4个季度间微生物群落组成 Fig.4 The most abundant phyla of bacterial community in sediment samples among the four seasons |
门级水平上, 冬夏两季样品主要菌门相对丰度排序为变形菌门 > 绿弯菌门 > 拟杆菌门 > 厚壁菌门 > 酸杆菌门, 春季样品中拟杆菌门取代绿弯菌门成为丰度第二的类群, 秋季样品中则是酸杆菌门 > 厚壁菌门。从时间角度上看, 养殖池塘底泥中5个主要菌门的相对丰度呈现出不同的变化趋势(图 5A)。变形菌门在养殖年度内始终保持较高数量, 绿弯菌门和酸杆菌门冬春季低于夏秋季, 而拟杆菌门和厚壁菌门刚好相反, 夏秋季的相对含量低于冬春季。蓝藻门、Nitrospinae菌门和硝化螺旋菌门都呈明显的单峰形变化。蓝藻门相对丰度一直较低(2%左右), 秋季的10月、11月以及冬季的12月含量明显上升, 最高峰出现在十一月(10.1%)。Nitrospinae菌门明显的单峰形变化主要出现在秋季(3.1%)和相邻的冬季12月份, 最高峰也出现在11月(3.6%)。硝化螺旋菌门相对丰度则是冬季较高, 最高峰出现在2月(2.9%)。
|
图 5 养殖池塘底泥微生物群落组成 (A)门级水平细菌群落结构的年度变动; (B)四季间10个主要菌门间相对丰度比较. Fig.5 Bacterial community composition (BCC) in sediment samples (A) Annual dynamic of BCC at the phylum level; (B) comparisons of 10 abundant phyla based on one-way ANOVA. |
对四个季度底泥样品间的细菌群落组成进行单因素方差分析(one-way ANOVA)检验, 结果表明, 酸杆菌门和螺旋体门在季度间存在显著性差异(P < 0.05)。其中, 秋季的酸杆菌门相对数量显著高于其他季节, 而螺旋体门刚好相反, 秋季的相对数量显著低于其他3个季节(图 5B)。
属级水平上, 养殖池塘底泥优势菌属主要为norank_f__Anaerolineaceae、norank_c__Acidobacteria、脱氯单胞菌属(Dechloromonas)和土杆菌属(Geobacter)。属级水平的聚类热图表明, 所有样品仍然聚为冬春和夏秋两簇(图 6)。其中, norank_f__ML635J-40_aquatic_grou、丹毒丝菌属(Erysipelothrix)和norank_f__SRB2的相对丰度在秋冬季明显低于春夏季。致病性的黄杆菌属(Flavobacterium)相对丰度在秋季明显低于其他3个季节。水产养殖中重要的益生菌芽孢杆菌属Bacillus相对丰度则是夏秋季明显低于冬春季。

|
图 6 养殖池塘底泥微生物属级水平的分层聚类热图 Fig.6 Bacterial community heatmap analysis in genus level |
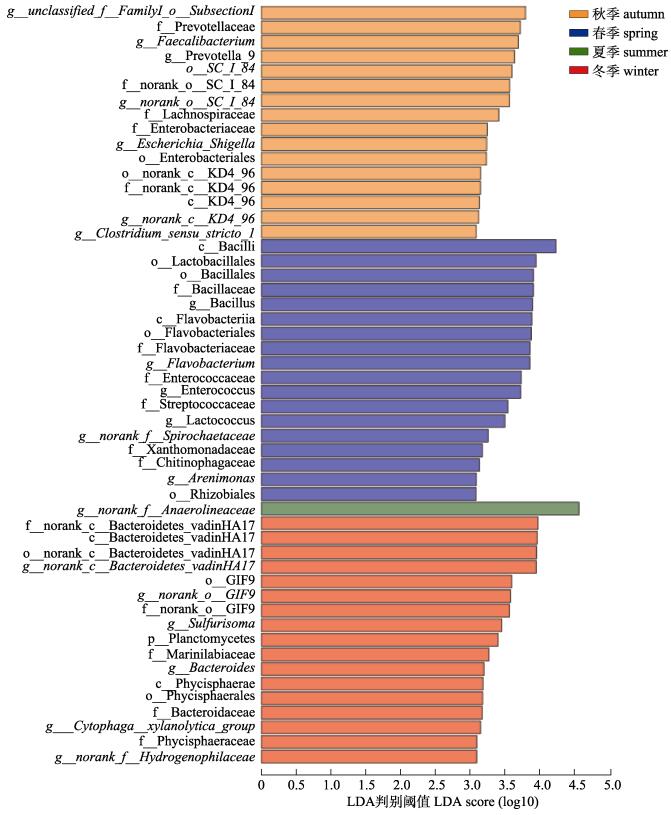
4个季度样品间的细菌群落单因素方差分析(one-way ANOVA)在2个菌门发现了显著性差异, 在时间维度上进一步采用LefSe软件养殖水体细菌群落样本进行线性判别分析(LDA), 以找到对各个季节显著性对应的群落或物种(P < 0.01)。LDA判别阈值大于3.0时, 共获得52个不同分类水平的细菌类群在4个季节间具有显著差异(图 7)。冬季底泥样品中共17个不同分类水平的细菌类群具有显著丰度, 主要在浮霉菌、拟杆菌门和绿弯菌门。优势性菌群在浮霉菌门中从门到Phycisphaeraceae科, 拟杆菌门中从VadinHA17纲到g__norank_c__Bacteroidetes_VadinHA17属以及绿弯菌门Chloroflexi从GIF9目到g__norank_ o__GIF9属。春季底泥样品的18个优势细菌类群, 集中于厚壁菌门和拟杆菌门。其中, 厚壁菌门中从杆菌纲(Bacilli)到芽孢杆菌属(Bacillus)和肠球菌属(Enterococcus)具有显著优势; 拟杆菌门中从黄杆菌纲Flavobacteriia到黄杆菌属(Flavobacterium)具有显著优势; 变形菌门中从黄单胞菌科(Xanthomonadaceae)到气单胞菌属(Aeromonas)具有显著优势。秋季底泥共有16个优势细菌类群, 但分布得极其分散。变形菌门中从肠杆菌目(Enterobacteriales)到肠杆菌属(Escherichia_Shigella)以及SC_Ⅰ_84目到norank_o__SC_Ⅰ_84属具有显著优势, 其他优势细菌类群点状散布于拟杆菌门、蓝藻门和厚壁菌门中。夏季优势细菌类群最少, 只有绿弯菌门中的厌氧绳菌属norank_f__ Anaerolineaceae这1个属具有显著优势。
|
图 7 斑点叉尾鮰养殖池塘底泥四季间差异性菌群多物种线性差异判别分析 Fig.7 LEfSe identified the most differentially abundant taxa in sediment samples among four season |
微生物群落结构与环境因子的影响紧密相关, 利用方差膨胀因子(variance inflation factor, VIF)分析对环境因子进行筛选, 过滤掉VIF大于10的存在共线性的环境因子pH、DO和OM。Monte Carlo排序检验表明剩下的因子中只有TDS和SD与细菌群落之间存在显著的相关性(P < 0.05)。6个因子对细菌群落格局的影响大小(r2)依次顺序是: SD > TDS > TN > Temp > TP > TOC (表 2)。
|
|
表 2 各环境因子对排序结果的相关性系数/及显著性检验值 Tab.2 Coefficient correlation and P values between physicochemical parameters and RDA axis |
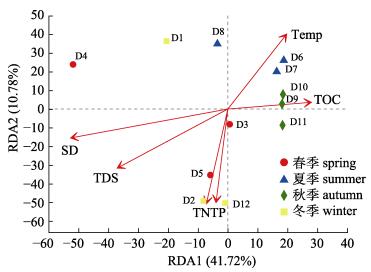
利用VIF分析挑选出来的6个与细菌群落显著相关的环境因子进行RDA排序分析, 结果表明, RDA1轴和RDA2轴分别解释了细菌群落变异的41.72%和10.78%, 主要菌群被聚为2簇(图 8)。第1簇由冬季和春季样品组成, 第2簇由夏季和秋季样品组成, 两簇被RDA2轴分隔。其中, 第1簇冬春季样本分布比较分散, 主要受SD、TDS、TN和TP的影响; 第2簇夏秋季样本分布较为紧密, 夏、秋季节的样品又独自聚类, 其中, 夏季样品主要受Temp的影响, 而秋季样品则更多受到TOC的影响。RDA结果显示, 斑点叉尾鮰养殖池塘底泥微生物具有比较明显的季节性。
|
图 8 细菌群落与环境因子之间的RDA分析 Fig.8 RDA bioplot of the distribution of bacterial community and environmental variables |
本文旨在通过分析斑点叉尾鮰养殖池塘底泥细菌群落的年度变化过程及其影响因素, 阐明斑点叉尾鮰养殖池塘底泥细菌群落的变化规律和影响机制。底泥在养殖系统中起着重要的作用, 养殖过程中投入的有机物和营养盐仅有很少的一部分被养殖对象利用, 而绝大部分(75%的N, 80%的P和75%的有机C)都以矿化、动物粪便、饵料残渣等形式富集于池塘的底泥中[20]。斑点叉尾鮰养殖池塘底泥在养殖年度内营养盐和有机质也持续保持较高浓度, 且季节间差异较小, 因此, 底泥细菌群落的多样性也基本保持稳定, 四季间没有显著差异, 仅在个别月份出现一定的波动。然而, 与养殖池塘水体相比, 斑点叉尾鮰养殖池塘底泥细菌群落丰富度是养殖池塘水体的4~5倍(Ace指数和Chao1指数), 底泥细菌群落的多样性也明显高于水体(Shannon指数和Simpson指数)。这主要是因为底泥中营养盐和有机碳的浓度比养殖水体浓度高数倍甚至几个数量级, 适合微生物大量繁殖栖息, 因此细菌的生物量能比上覆水高2~4个数量级[21]。
3.2 季节间菌群组成在内陆湖泊、水库以及海边等自然生态系统中, 底泥细菌群落结构都相对稳定, 只受到季节变化的微弱影响, 变形菌门、拟杆菌门和绿弯菌门往往是主要的优势菌群[22-24]。而对养殖池塘的研究表明, 只有变形菌门在不同养殖池塘是广泛分布的, 且通常为最优菌群, 其他优势菌群则随养殖品种不同存在较大差异[7-9]。斑点叉尾鮰养殖池塘底泥除变形菌门为最优菌群外, 其他优势菌群为绿弯菌门、拟杆菌门、厚壁菌门和酸杆菌门(图 4), 与底栖杂食性的罗非鱼(Oreochroms mossambcus)相近[9], 而与植食性草鱼(Ctenopharyngodon idellus)主养池塘底泥菌群差别较大[7]。养殖品种食性的不同会导致饲料投入的差别, 进而造成底泥有机物和营养盐浓度的差异。除食性和饲料的因素外, 鱼类生活习性也对养殖池塘底泥微生物群落起到重要影响。斑点叉尾鮰幼鱼喜爱集群, 成年雄鱼则有筑巢习性, 喜欢用尾巴在池塘底部扫出洞穴。生物扰动也增加底泥微生物的数量, 从而增加细菌群落多样性, 还能明显改变细菌群落结构[25-26]。因此, 斑点叉尾鮰贪食、底栖和筑巢的习性, 导致其养殖池塘底泥的细菌群落结构具有自己的特异性。
ANOSIM检验表明斑点叉尾鮰养殖池塘4个季度底泥样品间细菌群落组成存在显著性差异(P < 0.01), NMDS分析和聚类分析(图 2)也显示了所有样本呈冬春和夏秋两组的排列趋势。斑点叉尾鮰养殖池塘底泥优势菌中, 酸杆菌门秋季的相对数量显著高于其他季节, 而螺旋体门则是秋季相对数量显著低于其他3个季节(P < 0.05, 图 7)。LefSe分析进一步表明, 冬春季的差异菌群数量最多。冬季浮霉菌门和拟杆菌门的相对丰度显著高于其他及季节。浮霉菌门被发现广泛分布于各类水体和底泥中[27-28], 室内培养表明, 浮霉菌门更青睐良好的光照条件[29]。斑点叉尾鮰养殖池塘冬季水体透明度达到最大, 因此冬季池塘底部的光照较强, 为浮霉菌门的生长提供了最适当的条件。同时秋季开始, 底泥中蓝藻显著增加, 蓝藻的降解产生了大量的H2S并提高了锰、铁等离子的浓度, 也促进了冬季浮霉菌门的增殖[30]。
与养殖水体一样, 夏季斑点叉尾鮰养殖池塘底泥优势菌群种类也是年度最低, 只有绿弯菌门的厌氧蝇菌属1个菌属具有显著优势(图 7)。夏季养殖池塘水体蓝藻门相对含量大幅增加, 降低了水体的透明度, 并消耗了大量溶氧, 释因此, 养殖塘底泥上覆水的溶氧和pH均值达到年度最低, 为厌氧的绿弯菌门提供了最适的繁殖条件[31]。因此, 斑点叉尾鮰养殖过程中需要加强夏季换水, 保证水体和底泥溶氧, 减少蓝藻暴发, 从而提高池塘底泥微生物群落结构的多样性, 为底栖的斑点叉尾鮰提供舒适的夏季生活微生态环境。
秋季斑点叉尾鮰养殖池塘底泥优势菌群种类主要为蓝藻门、变形菌门和绿弯菌门(图 7)。其中, 蓝藻门优势菌群为g__unclassified_f__FamilyI_ o__SubsectionI属, 与养殖池塘水体秋季蓝藻的优势菌一致。该属归属于蓝藻纲Subsection Ⅰ, 皆为单细胞蓝藻, 有空泡, 并合成藻胆蛋白[32]。秋季开始, 底泥中蓝藻数量逐月升高, 并在11月末达到顶峰, 冬季12月含量也相对较高(图 5A)。这是因为蓝藻水华形成后, 尤其当生长环境条件不利时, 色球藻目的蓝藻(如微囊藻Microcystis)会聚集, 通过浮力调节沉降至相对黑暗、厌氧的表层沉积物中, 进入休眠状态, 帮助其越过环境条件恶劣的冬季[33]。
3.3 水产养殖益生菌和致病菌本研究发现, 斑点叉尾鮰养殖池塘底泥微生物中, 春季厚壁菌门和拟杆菌门的相对丰度显著高于其他及季节。其中, 厚壁菌门优势菌群集中于芽孢杆菌目和乳酸杆菌目, 这些菌群都是益生菌, 并被广泛应用于水产养殖中。芽孢杆菌和乳酸菌能够刺激机体的免疫系统或直接产生相关免疫物质进行免疫调节, 对多种病原菌都具有拮抗作用, 分泌或促进产生多种有益物质, 清除自由基, 抑制脂质过氧化, 从而提高养殖对象的免疫力, 促进其生长[34]。
拟杆菌门优势菌群集中于黄杆菌Flavobacteria类群, 该菌群能够利用和降解各类有机物[35], 但该菌还是条件致病菌, 当养殖环境较差(如高氨氮、低溶氧)是会造成养殖鱼类的鳍条、鱼鳃溃烂, 黄杆菌引起的柱状病(columnaris disease)已经成为美国斑点叉尾鮰产业面临的主要疾病之一[36], 也为我国斑点叉尾鮰养殖中的常见病害。柱状病主要爆发时间集中于春末。常规养殖管理认为, 鱼类经历了冬季低温停食后, 营养状况和免疫能力可能处于较差的状态, 容易在春季升温时被致病菌和病毒感染, 从而导致大面积的鱼类死亡, 因此伴随升温过程进行开春消毒工作。本文研究结果表明, 春季斑点叉尾鮰养殖池塘底泥优势菌群是益生菌和条件致病菌并存, 因此, 每年的开春消毒在杀灭致病菌的同时, 也会杀灭春季的优势益生菌群, 因此消毒工作完成后, 应该注意益生菌的补充, 恢复养殖环境益生菌的优势地位。同时, 黄杆菌与池塘有机质含量密切相关, 因此, 春节升温后的肥水工作要注意肥料使用种类和浓度, 过量有机肥会引起黄杆菌相对含量的激增, 还消耗水体溶氧, 增加了柱状病暴发概率。
3.4 主要影响因素Monte Carlo检验和RDA排序分析表明斑点叉尾鮰养殖池塘底泥细菌群落与环境因子间具有显著的相关性, 主要的环境因子包括SD和TDS (表 2)。2个环境因子都表现出明显的季节性差异, 冬春季的SD和TDS显著高于夏秋季(表 1)。SD对细菌群落的影响最大(r2 > 0.8), 水体透明度直接决定了光照射入池塘水体的深度, 控制了水体光合作用强度, 进而改变池塘底部的溶氧DO和pH, 并降低底泥细菌的活性, 最终影响有机物的降解。因此在进行方差膨胀因子(VIF)分析对环境因子进行筛选, pH、DO和OM与SD等环境因子多重共线性关系严重, 从而被剔除。在显著影响养殖池塘水体细菌群落结构的同时, TDS也显著影响这养殖池塘底泥的细菌群落结构, TDS能干扰水体的氧传递和降低细菌活性, 从而影响硝酸盐、有机物和COD的去除[37]。而营养盐和有机质全年都保持较高的浓度, 且季节间差异较小, 因此其对细菌群落结构的影响也最低。
本研究运用16S rDNA扩增子技术研究斑点叉尾鮰养殖池塘底泥微生物群落, 结果表明斑点叉尾鮰养殖池塘底泥细菌群落结构呈现出季节性变化, 冬春季样品相似性较高。优势菌群主要为变形菌门、绿弯菌门、拟杆菌门、厚壁菌门和酸杆菌门, 与水体细菌群落间有明显的差别。环境因子中, 透明度和总溶解性悬浮物对底泥细菌群落结构有显著的影响。
| [1] |
Boyd C E, Wood C W, Chaney P L, et al. Role of aquaculture pond sediments in sequestration of annual global carbon emissions[J]. Environmental Pollution, 2010, 158(8): 2537-2540. DOI:10.1016/j.envpol.2010.04.025 |
| [2] |
Avnimelech Y, Ritvo G. Shrimp and fish pond soils:Processes and management[J]. Aquaculture, 2003, 220(1-4): 549-567. DOI:10.1016/S0044-8486(02)00641-5 |
| [3] |
Yu T B, Shen L H. A preliminary study of the impact of humus on nitrogen nutritional salts of water quality environment[J]. Transactions of Oceanology and Limnology, 2006, 28(1): 82-85. [郁桐炳, 沈丽红. 池塘淤泥对水中氮营养盐影响的初步研究[J]. 海洋湖沼通报, 2006, 28(1): 82-85. DOI:10.3969/j.issn.1003-6482.2006.01.012] |
| [4] |
Bentzon-Tilia M, Sonnenschein E C, Gram L. Monitoring and managing microbes in aquaculture-towards a sustainable industry[J]. Microbial Biotechnology, 2016, 9(5): 576-584. |
| [5] |
Cardona E, Gueguen Y, Magré K, et al. Bacterial community characterization of water and intestine of the shrimp Litopenaeus stylirostris in a biofloc system[J]. BMC Microbiology, 2016, 16: 157. DOI:10.1186/s12866-016-0770-z |
| [6] |
Burford M A, Peterson E L, Baiano J C F, et al. Bacteria in shrimp pond sediments:Their role in mineralizing nutrients and some suggested sampling strategies[J]. Aquaculture Research, 1998, 29(11): 843-849. DOI:10.1111/j.1365-2109.1998.tb01110.x |
| [7] |
Cheng Y Y. Research on microbial community ecology in sediment of a pond cultured mainly with grass carp (Ctenopharyngodon idellus)[D]. Beijing: Graduate School of Chinese Academy of Sciences, 2011. [程莹寅.主养草鱼池塘底泥微生物群落生态学研究[D].北京: 中国科学院研究生院, 2011.] http://d.wanfangdata.com.cn/thesis/Y2056121
|
| [8] |
Zhao X G. Influence of the cultured Odontobutis obscurus to the microbial community, water quality and rice traits in rice-fish system[D]. Shanghai: Shanghai Ocean University, 2016. [赵翔刚.稻田养殖沙塘鳢对稻田微生物、水质及大米性状的影响[D].上海: 上海海洋大学, 2016.] http://cdmd.cnki.com.cn/Article/CDMD-10264-1016912264.htm
|
| [9] |
Fan L M, Barry K, Hu G D, et al. Characterizing bacterial communities in tilapia pond surface sediment and their responses to pond differences and temporal variations[J]. World Journal of Microbiology and Biotechnology, 2017, 33: 1. |
| [10] |
Masoud W, Takamiya M, Vogensen F K, et al. Characterization of bacterial populations in Danish raw milk cheeses made with different starter cultures by denaturating gradient gel electrophoresis and pyrosequencing[J]. International Dairy Journal, 2011, 21(3): 142-148. DOI:10.1016/j.idairyj.2010.10.007 |
| [11] |
Bolger A M, Lohse M, Usadel B. Trimmomatic:a flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [12] |
Reyon D, Tsai S Q, Khayter C, et al. FLASH assembly of TALENs for high-throughput genome editing[J]. Nature Biotechnology, 2012, 30(5): 460-465. DOI:10.1038/nbt.2170 |
| [13] |
Quast C, Pruesse E, Yilmaz P, et al. The SILVA ribosomal RNA gene database project:Improved data processing and web-based tools[J]. Nucleic Acids Research, 2012, 41(D1): D590-D596. DOI:10.1093/nar/gks1219 |
| [14] |
Schloss P D, Westcott S L, Ryabin T, et al. Introducing mothur:Open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied and Environmental Microbiology, 2009, 75(23): 7537-7541. DOI:10.1128/AEM.01541-09 |
| [15] |
Legendre P, Gallagher E D. Ecologically meaningful transformations for ordination of species data[J]. Oecologia, 2001, 129(2): 271-280. DOI:10.1007/s004420100716 |
| [16] |
Faith D P, Minchin P R, Belbin L. Compositional dissimilarity as a robust measure of ecological distance[J]. Vegetatio, 1987, 69(1-3): 57-68. DOI:10.1007/BF00038687 |
| [17] |
van den Wollenberg A L. Redundancy analysis an alternative for canonical correlation analysis[J]. Psychometrika, 1977, 42(2): 207-219. DOI:10.1007/BF02294050 |
| [18] |
Segata N, Izard J, Waldron L, et al. Metagenomic biomarker discovery and explanation[J]. Genome Biology, 2011, 12(6): R60. |
| [19] |
Galand P E, Casamayor E O, Kirchman D L, et al. Ecology of the rare microbial biosphere of the Arctic Ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(52): 22427-22432. |
| [20] |
Avnimelech Y, Lacher M. A tentative nutrient budget for intensive fish ponds[J]. Israeli Journal of Aquaculture-Bamidgeh, 1979, 31: 3-8. |
| [21] |
Ram N M, Zur O, Avnimelech Y. Microbial changes occurring at the sediment-water interface in an intensively stocked and fed fish pond[J]. Aquaculture, 1982, 27(1): 63-72. |
| [22] |
Bai Y, Shi Q, Wen D, et al. Bacterial communities in the sediments of Dianchi Lake, a partitioned eutrophic waterbody in China[J]. PLoS ONE, 2012, 7(5): e37796. DOI:10.1371/journal.pone.0037796 |
| [23] |
Mohit V, Archambault P, Lovejoy C. Resilience and adjustments of surface sediment bacterial communities in an enclosed shallow coastal lagoon, Magdalen Islands, Gulf of St. Lawrence, Canada[J]. FEMS Microbiology Ecology, 2015, 91(5): fiv038. |
| [24] |
Zhang J X, Yang Y Y, Zhao L, et al. Distribution of sediment bacterial and archaeal communities in plateau freshwater lakes[J]. Applied Microbiology and Biotechnology, 2015, 99(7): 3291-3302. |
| [25] |
Papaspyrou S, Gregersen T, Cox R P, et al. Sediment properties and bacterial community in burrows of the ghost shrimp Pestarella tyrrhena (Decapoda:Thalassinidea)[J]. Aquatic Microbial Ecology, 2005, 38: 181-190. |
| [26] |
Laverock B, Smith C J, Tait K, et al. Bioturbating shrimp alter the structure and diversity of bacterial communities in coastal marine sediments[J]. The ISME Journal, 2010, 4(12): 1531-1544. DOI:10.1038/ismej.2010.86 |
| [27] |
Musat N, Werner U, Knittel K, et al. Microbial community structure of sandy intertidal sediments in the North Sea, Sylt-Rømø Basin, Wadden Sea[J]. Systematic and Applied Microbiology, 2006, 29(4): 333-348. DOI:10.1016/j.syapm.2005.12.006 |
| [28] |
Miskin I P, Farrimond P, Head I M. Identification of novel bacterial lineages as active members of microbial populations in a freshwater sediment using a rapid RNA extraction procedure and RT-PCR[J]. Microbiology, 1999, 145(8): 1977-1987. DOI:10.1099/13500872-145-8-1977 |
| [29] |
Starr M P, Schmidt M J. Genus Planctomyces gimesi 1924[M]//Staley J T, Bryant M P, Pfennig N. Bergy's Manual of Systematic Bacteriology. Baltimore: Williams and Wilkins, 1989: 1946-1958.
|
| [30] |
Kristiansen J. On Planctomyces bekefii and its occurrence in Danish lakes and ponds[J]. Botansk Tidsskrift, 1971, 66: 293-302. |
| [31] |
Garrity G M, Holt J G. Chloroflexi phy. nov.[M]//Whitman W B, Rainey F, Kämpfer P, et al. Bergey's Manual of Systematics of Archaea and Bacteria. Chichester: John Wiley & Sons, 2015.
|
| [32] |
Herdman M, Iteman I, Rippka R, et al. Cyanobacteria/Subsection Ⅰ/Incertae Sedis[M]//Whitman W B, Rainey F, Kämpfer P, et al. Bergey's Manual of Systematics of Archaea and Bacteria. Chichester: John Wiley & Sons, 2015.
|
| [33] |
Brunberg A K, Blomqvist P. Benthic overwintering of Microcystis colonies under different environmental conditions[J]. Journal of Plankton Research, 2002, 24(11): 1247-1252. DOI:10.1093/plankt/24.11.1247 |
| [34] |
Irianto A, Austin B. Probiotics in aquaculture[J]. Journal of Fish Diseases, 2002, 25(11): 633-642. DOI:10.1046/j.1365-2761.2002.00422.x |
| [35] |
Kirchman D L. The ecology of Cytophaga-Flavobacteria in aquatic environments[J]. FEMS Microbiology Ecology, 2002, 39(2): 91-100. |
| [36] |
Arias C R, Cai W, Peatman E, et al. Catfish hybrid Ictalurus punctatus ×I. furcatus exhibits higher resistance to columnaris disease than the parental species[J]. Diseases of Aquatic Organisms, 2012, 100(1): 77-81. DOI:10.3354/dao02492 |
| [37] |
Yogalakshmi K N, Joseph K. Effect of transient sodium chloride shock loads on the performance of submerged membrane bioreactor[J]. Bioresource Technology, 2010, 101(18): 7054-7061. DOI:10.1016/j.biortech.2010.03.135 |
 2020, Vol. 27
2020, Vol. 27

