目前, 世界渔业发展迅速, 据世界粮农组织统计报告, 2016年全球鱼类的产量已高达约1.71亿t, 其中, 水产养殖占总产量的47%[1]。2018年, 中国水产养殖产量在世界产量中的比值高达60%[2]。与此同时, 高密度集约化养殖的弊端也逐渐显露出来。据报道, 在集约化养殖中, 莫桑比克罗非鱼(Oreochromis mossambicus)、虹鳟(Oncor- hynchus mykiss)、鲫(Carassius auratus)和鲤(Cyp- rinus carpio)等淡水养殖鱼类, 以及大黄鱼(Lari- michthys crocea)、黑鲷(Acanthopagrus schlegelii)、牙鲆(Paralichthys olivaceus)、鲈(Lateolabrax japonicus)、真鲷(Pagrus major)和大菱鲆(Scoph- thalmus maximus)等海水养殖鱼类的脂肪肝等代谢性疾病是困扰水产养殖者的重大难题之一[3]。因此, 营养代谢性疾病已经严重制约了水产养殖业的可持续健康发展, 预防和控制鱼类营养代谢性疾病迫在眉睫。
鱼体脂肪过度蓄积和脂肪肝等营养代谢性疾病的病因和发病机制极其复杂, 是内部遗传和外部环境共同作用所导致的能量代谢障碍性疾病[4-6]。现有研究多集中在环境、食物、药物和肠道微生物等外界环境因素上, 如高密度精养和集约化养殖模式下普遍存在养殖密度大、生产周期缩短、饲料营养失衡、强化投饲、饲料中含有有毒物质、乱用药和滥用药等问题[7]。这些问题直接导致养殖鱼类营养代谢紊乱, 免疫系统功能下降, 进而引发脂肪过度蓄积、瘦肌病、脂肪肝、肝硬化和高脂血症等营养性疾病的发生[8]。而且, 目前营养代谢性疾病的防治方法主要是通过适度降低鱼苗放养密度和尽量投喂营养均衡的饲料, 而在遗传基因层面的研究仍有一定的局限。但越来越多的研究表明, 内部遗传因素—基因在代谢性疾病中扮演着重要的角色[9-10]。寻找代谢性疾病相关基因, 特别是参与主要的能量物质(糖类、脂类和蛋白质)代谢通路以及调控能量物质代谢通路的基因, 研究其生物学功能和作用机理等逐步引起水产养殖学界和业界的关注和重视。
研究发现, 转录因子(PPARs、SREBP-1和LXRs等)[11-13]、脂肪细胞分泌因子(TNF-α、瘦素和胰岛素)[14-16]和主要能量物质代谢关键基因[葡萄糖激酶(GK)、己糖激酶(HK)、磷酸烯醇式丙酮酸激酶(PEPCK)、脂肪酸合成酶(FAS)、乙酰辅酶A羧化酶(ACC)和甘油三酯酶(ATGL)等][17-18]在鱼类营养代谢性疾病中发挥着关键作用。然而, 现有的研究结果多是关注某几个代谢基因或通路, 无法从基因组水平确定多基因如何调控鱼类营养性疾病发生发展的过程, 更无法确定这些基因如何单独或相互作用进而调控具体的代谢和疾病过程。随着测序技术的高速发展和广泛应用, 鱼类基因组的大量数据已开始涌现。组学数据为水产业的可持续发展提供了新的契机。但庞大的数据并不意味着有效的开发利用, 如何合理利用生物信息学方法在海量的数据中挖掘、处理、分析、阐释和存储有效的基因信息显得尤为重要。对渔业生产而言, 当务之急是将基因序列信息转变为有用的生物学知识, 进而使这些知识转化成技术并被产业化应用。即如何运用生物信息学这门技术挖掘鱼类基因库中的重要信息, 并筛选出关键的候选基因, 仍需加强[19]。为此, 本文主要综述了现阶段鱼类营养代谢性疾病及其相关基因数据库的研究进展, 并对数据库进行初步的挖掘和分析, 以期为代谢性疾病的预防与治疗提供新的研究视角。
1 鱼类营养代谢病概述 1.1 鱼类营养代谢病定义早在公元前2世纪, 《黄帝内经·素问》中就有饥荒时期关于营养不良出现浮肿等症状的记载, 并将此定义为人类的营养失调性疾病[20]。借鉴人类中营养代谢性疾病的概念, 大多数研究者将营养性疾病和新陈代谢障碍或紊乱性疾病统称为鱼类营养代谢性疾病。确切的说, 鱼类营养代谢性疾病是指由于某些营养元素过多、过少或配比不平衡引发鱼体代谢紊乱, 进而引发生理功能异常, 导致代谢性疾病的发生, 严重时导致鱼类死亡, 它是由环境和遗传共同作用的多基因遗传病[21]。
1.2 鱼类营养代谢性疾病的主要病因水体环境和饲料配比是目前导致代谢性疾病的主要因素。高密度养殖或者养殖水体受到污染时, 水体中氨氮、重金属或环境内分泌毒素污染含量可能会过高, 导致大量有毒有害物质蓄积在鱼体血液中, 从而引起鱼类代谢失调[22]。如果投喂的饲料中的某一种或者多种营养素不足时, 容易导致鱼体出现抗病力降低、生长缓慢和机体代谢发生紊乱等营养缺乏症。虽然营养物质是维持鱼类正常生理活动机能的基础, 但是鱼体一旦对其摄入过多, 反而会出现诸如厌食、营养性脂肪肝和抗病力下降等不利于鱼体健康生长的症状。饲料中能源物质过多对鱼体本身就是一种胁迫。比如蛋白质的积累会生产大量的氨氮等代谢废物, 并且将硬骨鱼类鱼体的血氨水平提高, 使鱼体出现毒性作用[23]; 同时脂类大量累积导致其氧化会产生酸类、醛类和酮类等代谢中间物[24]; 而糖类过多也会诱发脂肪肝等代谢性紊乱疾病及其炎性反应, 鱼体肝脏的解毒功能会大大降低, 最终导致鱼类死亡[25]。此外, 遗传因素对鱼类营养代谢病也起着关键性作用。例如, 脂肪酸合成酶(fatty acid synthase, FAS)基因和乙酰辅酶A羧化酶(acetyl-CoA carboxylase, ACC)基因是调控肉质性状、体脂沉积和脂肪合成速度的重要候选基因; 转录因子SREBP-1和PPARα在鱼类能量平衡中发挥着重要作用[26]。综上所述, 鱼类营养代谢性疾病受到多种因素的调控, 仅仅通过改善外界环境和优化饲料配比已经远远不能达到良好的疾病防控效果。因此, 从基因层面考虑并解决问题, 已经成为水产养殖业发展的趋势。
2 鱼类基因数据库研究现状组学技术的发展为系统的研究鱼类代谢性疾病提供了新的契机。利用比较基因组学构建鱼类代谢网络并筛选新的疾病候选基因已成为揭示代谢性疾病内在遗传机制的必由之路[27]。现存的鱼类基因组数据库是构建鱼类营养代谢数据库平台的主要数据来源。
2.1 NCBI数据库中的鱼类基因组数据库调研NCBI数据库(https://www.ncbi.nlm.nih.gov/)发现该数据库包含斑马鱼(Danio rerio)、虹鳟和鲫等共265种鱼类的基因组数据(表 1, 附表 1)。这些基因组数据可为后期鱼类代谢相关基因的发掘提供数据支撑。
|
|
表 1 NCBI中的主要鱼类数据库 Tab.1 Major fish database in NCBI |
|
|
附表 1 NCBI中的鱼类数据库 Attached table 1 Fish database in NCBI |
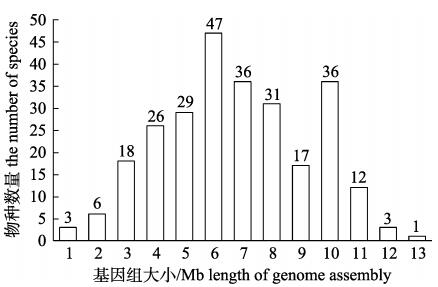
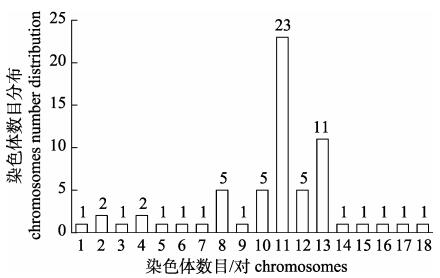
分析发现, 265种鱼类基因组为从0.001005~ 4470.98 Mb不等(图 1)。多数鱼类(47种)位于609.39~699.33 Mb的范围, 其次是分布在701.7~ 799.42 Mb的鱼类(36种)。同时, 我们列举并分析了14种世界水产养殖业生产的主要鱼类(表 1), 发现这14种鱼类基因组大小分布在544.29~ 2966.89 Mb之间, 其中在1001.52~1927.14 Mb中分布的最多为6种。另外, 该数据库还展示了64种鱼类的染色体数目(图 2), 不同鱼类染色体数目各异。其中, 鲫的染色体数目最多为59对, 遮目鱼(Chanos chanos)的染色体数目最少为16对; 另外, 64种鱼类中近40% (23种)的鱼类具有24对染色体。这些数据可为其他鱼类基因组物理图谱的构建提供参考。
|
图 1 NCBI中的鱼类基因组大小分布图 柱上数字为物种个数。横坐标表示基因组大小, 1~13分别表示基因组大小分布的不同区间, 1表示该物种的基因组大小尚未被测定出来; 2表示基因组大小分布在0.001005~73.43 Mb区间内; 3表示基因组大小分布在306.5~399.88 Mb区间内; 4表示基因组大小分布在401.04~498.28 Mb区间内; 5表示基因组大小分布在519.24~599.81 Mb区间内; 6表示基因组大小分布在609.39~699.33 Mb区间内; 7表示基因组大小分布在701.7~799.42 Mb区间内; 8表示基因组大小分布在800.49~899.65 Mb区间内; 9表示基因组大小分布在900.48~991.89 Mb区间内; 10表示基因组大小分布在1001.52~1927.14 Mb区间内; 11表示基因组大小分布在2068.07~2966.89 Mb区间内; 12表示基因组大小分布在3375.7~3811.04 Mb区间内; 13表示基因组大小分布在4470.98 Mb区间内。纵坐标表示分布各个区间的物种个数. Fig.1 Distribution of fish genome size in NCBI Species number is marked on the column.The abscissa represents the distribution interval of the genome size, 1 indicates that the genome size of this species has not been determined; 2 indicates that the genome size is distributed in the range of 0.001005-73.43 Mb; 3 indicates that the genome size is distributed in the 306.5-399.88 Mb interval; 4 indicates that the genome size is distributed in the 401.04-498.28 Mb interval; 5 indicates that the genome size is distributed in the 519.24-599.81 Mb interval; 6 indicates that the genome size is distributed in the 609.39-699.33 Mb interval; 7 indicates that the genome size is distributed in the 701.7-799.42 Mb interval; 8 indicates that the genome size is distributed in the 800.49-899.65 Mb interval; 9 indicates that the genome size is distributed in the range of 900.48-991.89 Mb; 10 indicates that the genome size is distributed in the range of 1001.52-1927.14 Mb; 11 indicates that the genome size is distributed in the range of 2068.07-2966.89 Mb; 12 indicates that the genome size is distributed in the range of 3375.7-3811.04 Mb; 13 indicates that the genome size is distributed in the range of 4470.98 Mb.And the ordinate represents the number of species. |
|
图 2 NCBI中的64种鱼类的染色体数目分布图 柱上数字为物种个数, 横坐标表示染色体数目, 纵坐标表示染色体数目分布。柱上数字1表示染色体数目为59对, 这样的物种有1种:鲫(Carassius auratus); 2表示染色体数目为51对, 这样的物种有2种:茴鱼(Thymallus thymallus)和条纹斑竹鲨(Chiloscyllium plagiosum); 3表示染色体数目为50对, 这样的物种有1种:鲤(Cyprinus carpio); 4表示染色体数目为40对, 这样的物种有2种:鳟(Salmo trutta)和白鲑(Coregonus); 5表示染色体数目为39对, 这样的物种有1种:北极红点鲑(Salvelinus alpinus); 6表示染色体数目为34对, 这样的物种有1种:帝王鲑(Oncorhynchus tshawytscha)。7表示染色体数目为30对, 这样的物种有1种:银鲑(Oncorhynchus kisutch); 8表示染色体数目为29对, 这样的物种包括斑点叉尾鮰(Ictalurus punctatus), 斑点雀鳝(Lepisosteus oculatus)等共5种鱼类; 9表示染色体数目为26对, 这样的物种有1种:大西洋鲱(Clupea harengus); 10表示染色体数目为25对, 这样的物种包括亚洲龙鱼(Scleropages formosus)、白斑狗鱼(Esox lucius)等共5种鱼类; 11表示染色体数目为24对, 这样的物种包括鮣鱼(Echeneis naucrates)、大黄鱼(Larimichthys crocea)等共23种鱼类; 12表示染色体数目为23对, 这样的物种包括大西洋鳕(Gadus morhua)、孔雀花鳉(Poecilia reticulata)等共5种鱼类; 13表示染色体数目为22对, 这样的物种包括红鳍东方鲀(Takifugu rubripes)、半滑舌鳎(Cynoglossus semilaevis)等共11种鱼类。14表示染色体数目为21对, 这样的物种有1种:泰国斗鱼(Betta splendens); 15表示染色体数目为20对, 这样的物种有1种:齿头鲱(Denticeps clupeoides)。16表示染色体数目为19对, 这样的物种有1种:弗氏假鳃鱂(Nothobranchius furzeri); 17表示染色体数目为18对, 这样的物种有1种是:芦鳗(Erpetoichthys calabaricus); 18表示染色体数目为16对, 这样的物种有1种:遮目鱼(Chanos chanos). Fig.2 Chromosome number distribution of 64 species of fish in NCBI The species numbers are marked on the columns.The abscissa represents the number of chromosomes, and the ordinate represents the distribution of the number of chromosomes.1 indicates that the number of chromosomes is 59 pairs.One such species is Carassius auratus.2 indicates that the number of chromosomes is 51 pairs.There are two species:Thymallus thymallus and Chiloscyllium plagiosum.3 indicates that the number of chromosomes is 50 pairs.One such species is Cyprinus carpio.4 indicates that the number of chromosomes is 40 pairs.There are two species:Salmo trutta and Coregonus.5 indicates that the number of chromosomes is 39 pairs.One such species is Salvelinus alpinus.6 indicates that the number of chromosomes is 34 pairs.One such species is Oncorhynchus tshawytscha.7 indicates that the number of chromosomes is 30 pairs.One such species is Oncorhynchus kisutch.8 indicates that the number of chromosomes is 29 pairs.Such species include five species of fish, such as Ictalurus punctatus and Lepisosteus oculatus, etc.9 indicates that the number of chromosomes is 26 pairs.One such species is Clupea harengus.10 indicates that the number of chromosomes is 25 pairs.Such species include 5 species of fish, such as Scleropages formosusand Esox lucius.11 indicates that the number of chromosomes is 24 pairs.Such species include a total of 23 species of fish, such as Echeneis naucrates and Larimichthys crocea, etc.12 indicates that the number of chromosomes is 23 pairs.Such species include a total of 5 species of fish, such as Gadus morhua and Poecilia reticulata.13 indicates that the number of chromosomes is 22 pairs.Such species include 11 species of fish such as the Takifugu rubripes and Cynoglossus semilaevis, etc.14 indicates that the number of chromosomes is 21 pairs.One such species is Betta splendens.15 indicates that the number of chromosomes is 20 pairs.One such species is Denticeps clupeoides.16 indicates that the number of chromosomes is 19 pairs.One such species is Nothobranchius furzeri.17 indicates that the number of chromosomes is 18 pairs.One such species is Erpetoichthys calabaricus.18 indicates that the number of chromosomes is 16 pairs.One such species is Chanos chanos. |
调研Ensembl数据库发现, 该数据库包含脂鲤目(Characiformes)、鲱形目(Clupeiformes)、鲤形目(Cypriniformes)、鳉形目(Cyprinodontifor- mes)、鳕形目(Gadiformes)、刺鱼目(Gasterosteifor- mes)、电鳗目(Gymnotiformes)、雀鳝目(Lepidostei- formes)、盲鳗目(Myxiniformes)、骨舌鱼目(Osteog- lossiformes)、鲈形目(Perciformes)、鲽形目(Pleu- ronectiformes)、多鳍鱼目(Polypteriformes)、鲑形目(Salmoniformes)、鲇形目(Siluriformes)、合鳃鱼目(Synbgranchiformes)和鲀形目(Tetraodontiformes)共17个目58种鱼类的基因组数据(表 2)。其中, 鱼类种类最多的是鲈形目有23种, 其次是鳉形目, 有12种。脂鲤目、鳕形目、电鳗目、盲鳗目、多鳍鱼目、合鳃鱼目、鲇形目及雀鳝目都只有1种鱼类。研究表明, 从无颌脊椎动物向四足动物演化的进程中, 出现过4次全基因组倍增事件, 斑点雀鳝(Lepisosteus oculatus)是基因组倍增事件的分界点[28], 作为相对原始的1种鱼, 其基因组没有出现鱼类特有的基因组倍增事件。对斑点雀鳝基因组信息的挖掘与分析, 可为鱼类关键代谢疾病相关基因的结构和功能演化提供数据支持。
|
|
表 2 Ensembl中的鱼类数据库 Tab.2 Fish database in Ensembl |
一些重要的模式生物或经济物种的基因组数据库也可为鱼类疾病数据库的构建提供参考。比如, 鱼类模式生物斑马鱼数据库(Zebrafish Model Organism Database, ZFIN, https://zfin.org/)包含斑马鱼的基因信息以及斑马鱼基因组图谱信息、突变品系、野生型品系和转基因品系等资源[29]。美国国家水产养殖基因组计划数据库(National Aqua- culture Genome Project, https://www.animalgenome.org/aquaculture/database/.)包括了鲇、虹鳟和三文鱼等重要经济鱼类的EST数据库以及其基因组物理图谱等[30]。中国科研工作者开发的主要经济鱼类草鱼基因组资源数据库(Grass Carp Genome Database, GCGD, http://bioinfo.ihb.ac.cn/gcgd/php/index.php)揭示了其草食性特征的遗传基础[31-32]。这些数据库可为经济鱼类的繁育、健康养殖及关键基因的分子机制研究提供参考。
3 鱼类营养代谢性疾病相关基因数据库目前, 鱼类疾病基因数据库的研究仍处于起步阶段。专门的鱼类代谢性疾病的数据库仍比较少。前期研究在人类2型糖尿病数据库[33-34]的基础上构建了斑马鱼中脂代谢疾病相关基因的数据库[27]。但仍需进一步发掘其他重要经济鱼类的代谢性疾病的数据库。为了更好地研发鱼类代谢性疾病相关基因数据库, 我们调研了以下三个重要的代谢和疾病数据库。
3.1 KEGG数据库中的鱼类代谢相关基因数据库KEGG数据库(http://www.genome.jp/kegg/)[35-36]中的代谢通路网站(KEGG PATHWAY)提供了详尽的代谢和调控通路图及基因, 可以从中下载到鱼类基因组中调控和参与主要能量物质(糖类、脂类和蛋白质)代谢的基因。氨基酸是蛋白质的基本组成单位, 本文的蛋白质代谢通路包括KEGG数据库中的氨基酸代谢通路(amino acid metabolism)和其他氨基酸代谢通路(metabolism of other amino acids)两类。调研KEGG数据库, 发现该数据库包含了包括斑马鱼、鲤和斑点叉尾鮰(Ictalurus punctatus)等41种硬骨鱼的数据库, 还包括米氏叶吻银鲛(Callorhinchus milii)和鲸鲨(Rhincodon typus)两种软骨鱼的代谢信息(表 3)。其中, 滇池金线鲃(Sinocyclocheilus grahami)糖类、脂类和蛋白质代谢相关基因数目最多, 为3817个; 米氏叶吻银鲛的数目最少, 为1589个。
|
|
表 3 KEGG中的鱼类数据库 Tab.3 Fish database in KEGG |
作为鱼类、人类乃至所有动物界疾病研究的重要的模式生物, 斑马鱼具有较为完善的基因组数据。根据现有的KEGG数据库中获得的斑马鱼代谢通路相关基因列表(表 4), 发现参与斑马鱼糖代谢通路的基因有591个, 其中调节磷酸肌醇代谢通路的基因最多有90个, 其次是糖酵解和糖异生通路的基因有78个。脂代谢通路的基因有573个, 其中调控甘油磷脂代谢通路的基因最多为101个, 其次是甘油酯代谢通路的基因有66个。蛋白质代谢通路的基因有636个, 其中调控赖氨酸降解通路的基因最多为77个, 其次是精氨酸和脯氨酸代谢的基因有61个。另外, 调控氰氨基酸代谢和D型丙氨酸代谢通路的基因数为0个。通过对斑马鱼主要代谢通路基因的研究, 可以为深入挖掘其他鱼类的代谢相关基因提供方法支撑。
|
|
表 4 以KEGG为准, 斑马鱼代谢通路相关基因列表 Tab.4 Zebrafish metabolism related gene pathway list in KEGG |
Reactome数据库(http://www.reactome.org)包含了人类中间代谢通路、调节通路和信号转导, 以及细胞周期等生物学过程的通路[37]。此外, 还覆盖了UniProt和OMIM数据库中的关键功能和疾病相关的蛋白质[38]。和KEGG数据库相比, 该数据库代谢通路图片分辨率高, 使用便捷, 是一个改良的基因搜索和挖掘工具。因此, 它也是研究鱼类疾病的一个重要参考数据库。
3.3 OMIM数据库中疾病相关基因OMIM (Online Mendelian Inheritance in Man)数据库(https://www.omim.org/), 是人类基因和遗传表型的知识库[39]。该数据库包含所有已知的孟德尔疾病的基因信息超过15000个。每个OMIM条目都有遗传表型和/或基因的全文摘要, 并且与其他遗传资源(如DNA和蛋白质序列, PubMed参考, 突变数据库和基因命名法等)有着丰富的联系[40]。可以根据人类的疾病数据, 寻找鱼类中相关基因的直系同源物, 构建鱼类代谢性疾病数据库[27]。
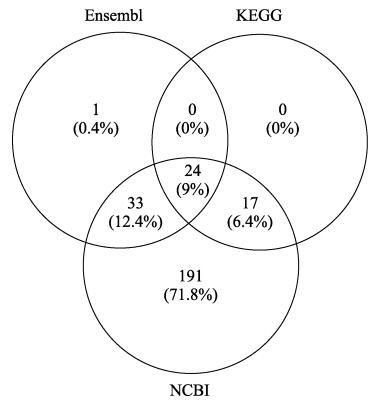
4 不同数据库鱼类基因组数据库比较分析为了整合不同数据库中现有的鱼类基因组数据, 本研究比较分析了NCBI、Ensembl和KEGG数据库中所包含的鱼类数据库信息(图 3)。结果发现NCBI中包含的鱼类数据库数量最多, 有191种是NCBI所特有的, 有1种是Ensembl特有的。另外, 3个数据库中共有的鱼类数据库有24种, Ensembl和NCBI中都包括的鱼类数据库有33种, KEGG和NCBI中共有的鱼类数据库有17种。综上所述, 目前三大数据库中共有266种鱼类基因组数据库, 这些数据是鱼类脂代谢研究的出发点, 可为寻找代谢性疾病相关基因以及其网络平台的建立提供数据支撑[41], 但如何有效深入挖掘这些基因信息仍需进一步研究。
|
图 3 NCBI、Ensembl、KEGG中的鱼类数据库Venn图 Fig.3 Venn diagram of fish database in NCBI, Ensembl and KEGG |
海量的组学数据为鱼类营养代谢病疾病的防治及水产业的可持续发展提供了新的契机。但庞大的数据并不意味着完全有效的开发利用, 现有测序数据大多处于“沉睡”的状态。因此, 本文首先对鱼类营养代谢病进行简单介绍, 然后对NCBI、Ensembl及KEGG三大数据库中所包含的鱼类基因组数据和代谢相关基因等基本信息进行分析, 发现每个数据库所包含的鱼类基因组信息都有各自的侧重点, 如何有效地整合和合理地利用这些基因组信息亟待加强。针对目前专门的鱼类代谢性疾病基因数据不足的现状, 在未来的研究中应该综合已有的数据信息, 利用比较基因组学等生物信息学和网络开发的方法, 深入研发完善的鱼类营养代谢性疾病相关基因数据库网络平台, 从而促进基因信息的高效使用, 并为鱼类营养代谢性疾病的分子机制研究奠定基础。
| [1] |
FAO. The state of world fisheries and aquaculture 2018-meeting the sustainable development goals[M]. Rome: Food Agricultural Organization of the United Nations, 2018: 2-3.
|
| [2] |
Bureau of Fisheries, Ministry of Agriculture and Rural Affairs, National Fisheries Technology Extension Center, China Society of Fisheries. China Fishery Statistical Yearbook, 2019[M]. Beijing: China Agriculture Press, 2019: 127-128. [农业农村部渔业渔政管理局, 全国水产技术推广总站, 中国水产学会. 2019中国渔业统计年鉴[M]. 北京: 中国农业出版社, 2019: 127-128.]
|
| [3] |
Vatsos I N, Angelidis P. Water quality and fish diseases[J]. Journal of the Hellenic Veterinary Medical Society, 2017, 61(1): 40-48. DOI:10.12681/jhvms.14875 |
| [4] |
Bouchard C. Gene-environment interactions in the etiology of obesity:Defining the fundamentals[J]. Obesity, 2008, 16(S3): S5-S10. |
| [5] |
Beckers J, Wurst W, de Angelis M H. Towards better mouse models:Enhanced genotypes, systemic phenotyping and envirotype modelling[J]. Nature Reviews Genetics, 2009, 10: 371-380. |
| [6] |
KilpelĠ inen T O, Zillikens M C, Stančákova A, et al. Genetic variation near IRS1 associates with reduced adiposity and an impaired metabolic profile[J]. Nature Genetics, 2011, 43(8): 753-760. DOI:10.1038/ng.866 |
| [7] |
Zhang S B. Causes of fatty liver in fish and its prevention and treatment[J]. China Fisheries, 2011(2): 55. [张水波. 鱼类脂肪肝的成因及防治对策[J]. 中国水产, 2011(2): 55. DOI:10.3969/j.issn.1002-6681.2011.02.027] |
| [8] |
Du Z Y. Causes of fatty liver in farmed fish:A review and new perspectives[J]. Journal of Fisheries of China, 2014, 38(9): 1628-1638. [杜震宇. 养殖鱼类脂肪肝成因及相关思考[J]. 水产学报, 2014, 38(9): 1628-1638.] |
| [9] |
Lutfi E, Gong N P, Johansson M, et al. Breeding selection of rainbow trout for high or low muscle adiposity differentially affects lipogenic capacity and lipid mobilization strategies to cope with food deprivation[J]. Aquaculture, 2018, 495: 161-171. DOI:10.1016/j.aquaculture.2018.05.039 |
| [10] |
Tahergorabi Z, Moodi M, Zardast M, et al. Metabolic syndrome and the risk of gastrointestinal cancer:A case-control study[J]. Asian Pacific Journal of Cancer Prevention, 2018, 19(8): 2205-2210. |
| [11] |
Caminada D, Fent K. Detection of three peroxisome proliferators-activated receptors (PPARs) in the fish cell line PLHC-1 and effects of fibrates[J]. Marine Environmental Research, 2008, 66(1): 71-71. DOI:10.1016/j.marenvres.2008.02.026 |
| [12] |
Ning L J, He A Y, Li J M, et al. Mechanisms and metabolic regulation of PPARα activation in Nile tilapia (Oreochromis niloticus)[J]. Biochimica et Biophysica Acta (BBA)-Molecular and Cell Biology of Lipids, 2016, 1861(9): 1036-1048. |
| [13] |
Dong X J, Tan P, Cai Z N, et al. Regulation of FADS2 transcription by SREBP-1 and PPAR-α influences LC-PUFA biosynthesis in fish[J]. Scientific Reports, 2017, 7: 40024. DOI:10.1038/srep40024 |
| [14] |
Raddatz D, Ramadori G. Carbohydrate metabolism and the liver:Actual aspects from physiology and disease[J]. Zeitschrift für Gastroenterologie, 2007, 45(1): 51-62. |
| [15] |
Plagnes-Juan E, Lansard M, Seiliez I, et al. Insulin regulates the expression of several metabolism-related genes in the liver and primary hepatocytes of rainbow trout (Oncorhynchus mykiss)[J]. Journal of Experimental Biology, 2008, 211(15): 2510-2518. DOI:10.1242/jeb.018374 |
| [16] |
Soengas J L. Contribution of glucose- and fatty acid sensing systems to the regulation of food intake in fish.A review[J]. General and Comparative Endocrinology, 2014, 205: 36-48. DOI:10.1016/j.ygcen.2014.01.015 |
| [17] |
Basaranoglu M, Basaranoglu G, Bugianesi E. Carbohydrate intake and nonalcoholic fatty liver disease:fructose as a weapon of mass destruction[J]. Zeitschrift für Gastroenterologie, 2015, 4(2): 109-116. |
| [18] |
Softic S, Cohen D E, Kahn C R. Role of dietary fructose and hepatic de novo lipogenesis in fatty liver disease[J]. Digestive Diseases and Sciences, 2016, 61(5): 1282-1293. DOI:10.1007/s10620-016-4054-0 |
| [19] |
Bei J X, Zhang Y, Li W S, et al. Application of genome databases and bioinformatics in exploration for fish genes[J]. Acta Hydrobiologica Sinica, 2008, 32(3): 387-392. [贝锦新, 张勇, 李文笙, 等. 鱼类基因数据库与生物信息学在鱼类基因开发上的应用[J]. 水生生物学报, 2008, 32(3): 387-392. DOI:10.3321/j.issn:1000-3207.2008.03.015] |
| [20] |
Zhu X Y. Internal Medicine:Endocrine Gland Disease, Kidney Disease, Nutritional Disorders Metabolic Disease[M]. Tianjin: Tianjin Scientific & Technical Publishers, 1981: 409. [朱宪彝. 内科学:内分泌腺疾病、肾脏疾病、营养障碍性疾病、新陈代谢疾病分册[M]. 天津: 天津科学技术出版社, 1981: 409.]
|
| [21] |
Leatherland J F, Woo P T F. Fish Diseases and Disorders[M]. Vol. 2. Wallingford: CABI, 2010.
|
| [22] |
Wang T, Wang W, Chen T Q, et al. Effects of acute copper stress from copper accumulation, oxidative stress, digestive enzymes, tissue lesions, and gene expression related to lipid metabolism in Takifugu fasciatus[J]. Journal of Fishery Sciences of China, 2019, 26(6): 1144-1152. [王涛, 王玮, 陈同庆, 等. 急性铜胁迫对暗纹东方鲀组织铜积累、氧化应激、消化酶、组织病变及脂代谢相关基因表达的影响[J]. 中国水产科学, 2019, 26(6): 1144-1152.] |
| [23] |
Zeng B H, Liu H P, Wang J, et al. Effects of dietary protein levels on muscular amino acids and protein metabolism of Schizopygopsis younghusbandi younghusbandi Regan[J]. Journal of Fishery Sciences of China, 2019, 26(6): 1153-1163. [曾本和, 刘海平, 王建, 等. 饲料蛋白质水平对拉萨裸裂尻鱼幼鱼肌肉氨基酸及蛋白质代谢的影响[J]. 中国水产科学, 2019, 26(6): 1153-1163.] |
| [24] |
Ai Q H, Yan J, Mai K S. Research progresses of lipids and fatty acids transport in fish[J]. Acta Hydrobiologica Sinica, 2016, 40(4): 859-868. [艾庆辉, 严晶, 麦康森. 鱼类脂肪与脂肪酸的转运及调控研究进展[J]. 水生生物学报, 2016, 40(4): 859-868.] |
| [25] |
Niu S J, Guo J L, Yang Z C, et al. A review:Research advances in glucose tolerance in fish[J]. Fisheries Science, 2015, 34(7): 459-463. [牛思佳, 郭金龙, 杨振才, 等. 鱼类葡萄糖耐量研究进展[J]. 水产科学, 2015, 34(7): 459-463.] |
| [26] |
Ning L J, He A Y, Li J M, et al. Mechanisms and metabolic regulation of PPARα activation in Nile tilapia (Oreochromis niloticus)[J]. Biochimica et Biophysica Acta (BBA)-Molecular and Cell Biology of Lipids, 2016, 1861(9): 1036-1048. |
| [27] |
Zhang Y R, Qin C B, Yang L P, et al. A comparative genomics study of carbohydrate/glucose metabolic genes:From fish to mammals[J]. BMC Genomics, 2018, 19: 246. DOI:10.1186/s12864-018-4647-4 |
| [28] |
Venkatachalam A B, Fontenot Q, Farrara A, et al. Fatty acid-binding protein genes of the ancient, air-breathing, ray-finned fish, spotted gar (Lepisosteus oculatus)[J]. Comparative Biochemistry and Physiology Part D:Genomics and Proteomics, 2018, 25: 19-25. |
| [29] |
Howe D G, Bradford Y M, Eagle A, et al. The zebrafish model organism database:New support for human disease models, mutation details, gene expression phenotypes and searching[J]. Nucleic Acids Research, 2017, 45(D1): D758-D768. DOI:10.1093/nar/gkw1116 |
| [30] |
Laghari M Y, Lashari P, Zhang Y, et al. Identification of quantitative trait loci (QTLs) in aquaculture species[J]. Reviews in Fisheries Science & Aquaculture, 2014, 22(3): 221-238. |
| [31] |
Wang Y P, Lu Y, Zhang Y, et al. The draft genome of the grass carp (Ctenopharyngodon idellus) provides insights into its evolution and vegetarian adaptation[J]. Nature Genetics, 2015, 47(6): 625-631. DOI:10.1038/ng.3280 |
| [32] |
Xu P, Wang J, Wang J T, et al. Generation of the first BAC-based physical map of the common carp genome[J]. BMC Genomics, 2011, 12: 537. DOI:10.1186/1471-2164-12-537 |
| [33] |
Agrawal S, Dimitrova N, Nathan P, et al. T2D-Db:An integrated platform to study the molecular basis of type 2 diabetes[J]. BMC Genomics, 2008, 9: 320. DOI:10.1186/1471-2164-9-320 |
| [34] |
Yang Z Z, Yang J H, Liu W, et al. T2D@ZJU:A knowledgebase integrating heterogeneous connections associated with type 2 diabetes mellitus[J]. Database, 2013, 2013: bat052. |
| [35] |
Kanehisa M, Furumichi M, Tanabe M, et al. KEGG:new perspectives on genomes, pathways, diseases and drugs[J]. Nucleic Acids Research, 2017, 45(D1): D353-D361. DOI:10.1093/nar/gkw1092 |
| [36] |
Kanehisa M, Sato Y, Furumichi M, et al. New approach for understanding genome variations in KEGG[J]. Nucleic Acids Research, 2019, 47(D1): D590-D595. |
| [37] |
Joshi-Tope G, Gillespie M, Vastrik I, et al. Reactome:A knowledgebase of biological pathways[J]. Nucleic Acids Research, 2005, 33(S1): D428-D432. |
| [38] |
Croft D, Mundo A F, Haw R, et al. The Reactome pathway knowledgebase[J]. Nucleic Acids Research, 2014, 42(D1): D472-D477. DOI:10.1093/nar/gkt1102 |
| [39] |
Amberger J S, Hamosh A. Searching Online Mendelian Inheritance in Man (OMIM):A knowledgebase of human genes and genetic phenotypes[J]. Current Protocols in Bioinformatics, 2017, 58(1): 1.2.1-1.2.12. |
| [40] |
Hamosh A, Scott A F, Amberger J S, et al. Online Mendelian Inheritance in Man (OMIM), a knowledgebase of human genes and genetic disorders[J]. Nucleic Acids Research, 2005, 33(S1): D514-D517. |
| [41] |
Zhang Y R, Zou X J, Ding Y H, et al. Comparative genomics and functional study of lipid metabolic genes in Caenorhabditis elegans[J]. BMC Genomics, 2013, 14: 164. DOI:10.1186/1471-2164-14-164 |
 2020, Vol. 27
2020, Vol. 27

